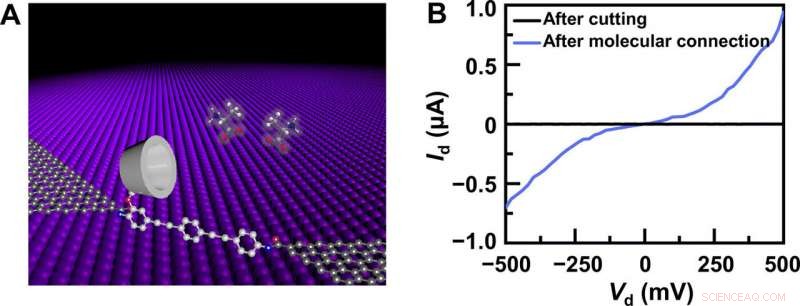
Enhetsstruktur och elektrisk karakterisering. (A) Schematisk representation av en PM-β-CD-baserad GMG-SMJ. Den molekylära maskinen med en PM-β-CD var kovalent ansluten till grafenpunktskontakter genom amidbindningar. (B) I-V-kurvor för GMG-SMJs efter syreplasmaskärning och efter ytterligare molekylär anslutning. Kreditera: Vetenskapens framsteg , doi:10.1126/sciadv.abe4365
Analytisk kemi syftar till att effektivt skilja mellan två aminosyror. I en ny rapport i Vetenskapens framsteg , Zihao Liu och ett forskarteam inom kemi, Fysik, och materialvetenskap i Kina och Japan, använde en elektrisk metod med en molekyl baserad på molekylära nanokretsar för att bilda stabila grafen-molekyl-grafen-enkelmolekylövergångar. Teamet utvecklade dessa molekylära korsningar genom att kovalent binda en molekylär maskin. Sedan använde de pH för att variera typen och laddningen av aminosyrorna för att hitta distinkta multimodala strömfluktuationer som härrörde från olika värdgästinteraktioner i förhållande till teoretiska beräkningar. Konduktansdata producerade karakteristiska utjämningstider och överföringshastigheter för varje aminosyra för att möjliggöra exakta och realtidsmätningar in situ. Forskarna testade fyra aminosyror och deras enantiomerer (kirala molekyler som är spegelbilder av varandra) för att visa förmågan att skilja mellan dem på några mikrosekunder för att tillhandahålla en enkel och exakt metod för aminosyraidentifiering såväl som enmolekylär protein sekvensering.
Byggstenar av proteiner
Aminosyror bildar byggstenar av proteiner, molekyler med speciella fysiologiska funktioner och viktiga strukturella element i läkemedel med tillämpningar inom biovetenskap. Forskare kan känna igen enantiomerer av aminosyror för att ge viktig information om kiral igenkänning och fysiologiska funktioner i biologiska system. Upptäcka strukturen, enantiomerisk renhet och aminosyrors dynamiska beteende kan främja produktionen av nya tekniker för proteinsekvensering och farmaceutiska undersökningar. Detektering och identifiering av aminosyror med olika strukturer och kiralitet är avgörande för proteomik, läkemedel och nanobioteknik. Dock, existerande molekylära maskiner fungerar bara genom att jämföra ensemblens termodynamiska parametrar, vilket är otillräckligt. Det är därför viktigt att utveckla en allmän molekylär teknik för att revolutionera de befintliga metoderna och känna igen målmolekyler med hög noggrannhet. I det här arbetet, Liu et al. demonstrerade en enkelmolekylteknik för att direkt identifiera olika proteinogena aminosyror och deras enantiomerer via exakta dynamiska mätningar av värd-gäst-interaktioner baserad på en enkelmolekyl-teknik känd som grafen-molekyl-grafen enkelmolekylövergångar förkortade som GMG-SMJs för att kovalent införliva individuella molekylära system som beter sig som den ledande kanalen i en elektrisk nanokrets.
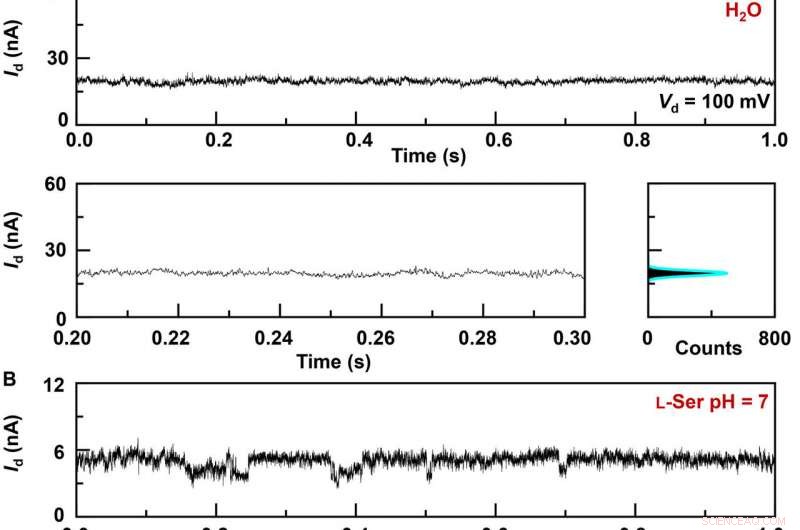
Aktuella inspelningar i realtid av värd-gästinteraktionsdynamik i GMG-SMJs. I-t-kurvor för olika GMG-SMJ (överst), de förstorade delarna (nedre till vänster), och deras motsvarande histogram (nedre till höger):(A) En fungerande enhet i ett rent vatten, (B) samma anordning i en 100 nM L-Ser vattenlösning, och (C) en annan anordning i en 100 nM L-Ala vattenlösning. Infällningar i histogrammen är de förstorade delarna av de små topparna. pH =7, T =298 K, och förspänningen =0,1 V. Kredit:Science Advances, doi:10.1126/sciadv.abe4365
Det nya tillvägagångssättet gav en robust plattform för att bilda enmolekylär elektronik för att skapa molekylära optoelektroniska enheter. För att utveckla konstruktionerna, Liu et al. kovalent inklämd en molekylär maskin mellan ett par nano-glappade grafenpunktkontakter för aminosyradetektering och kiralitetsigenkänning. Resultaten kommer att öppna nya vägar för att utveckla nanoteknologin för exakt enmolekylär proteinsekvensering för praktiska tillämpningar.
Enhetsutveckling, elektrisk karakterisering och realtidsmätningar
Forskargruppen syntetiserade enskiktsgrafen med kemisk ångavsättning på kopparfolier och överförde den till kiseldioxid/kiselwafers och mönstrade metallelektroderna med fotolitografi. För att utveckla grafenpunktskontaktelektroderna med nano-gap, de använde en streck-line litografisk metod. De konstruerade sedan GMG-SMJs (grafen-molekyl-grafen-enkelmolekylövergångar) genom att kovalent koppla en enda molekylär maskin med grafenelektroder via amidkopplingar. Teamet mätte ström-spänningskurvorna för enheterna i en mängd olika stadier för att identifiera bildandet av GMG-SMJs. Laddningstransporten genom korsningen resulterade från enkelmolekylära anslutningar. De utförde sedan tidsberoende elektriska karakteriseringar för att övervaka konduktansen av permetylerad-β-cyklodextrin (PM-β-CD) enkla molekylövergångar (SMJs) i realtid. De mätte först konstruktionerna i vatten och sedan i olika lösningar av olika aminosyror. De representativa ström-tidsbanorna avslöjade multipla toppfördelningar för olika aminosyror i lösning såsom L-serin och L-alanin. De multipla distinkta tillstånden för varje aminosyra härrörde endast från association/dissociationsprocessen mellan det funktionella PM-β-CD-centret och de omgivande aminosyrorna.
Beräkningsanalyser av L-Ala@PM-β-CD SMJs. (A) Schematiskt diagram över de beräknade gränsmolekylära orbitalerna för molekylmaskinen. (B till D) Typiska molekylära konfigurationer under associationen med olika laddade aminosyror:(B) katjon, (C) zwitterion, och (D) anjon. (E) Motsvarande transmissionsspektra vid en nollförspänning. Det röda, gul, och gröna kurvor visar transmissionen när anjonen, zwitterion, och katjonformer fanns i håligheten, respektive. Den blå kurvan visar tomrummet. Hela spektrat ges i fig. S4. (F till H) Schematisk representation av aminosyror i katjon, zwitterion, och anjonformer. Kredit:Science Advances, doi:10.1126/sciadv.abe4365 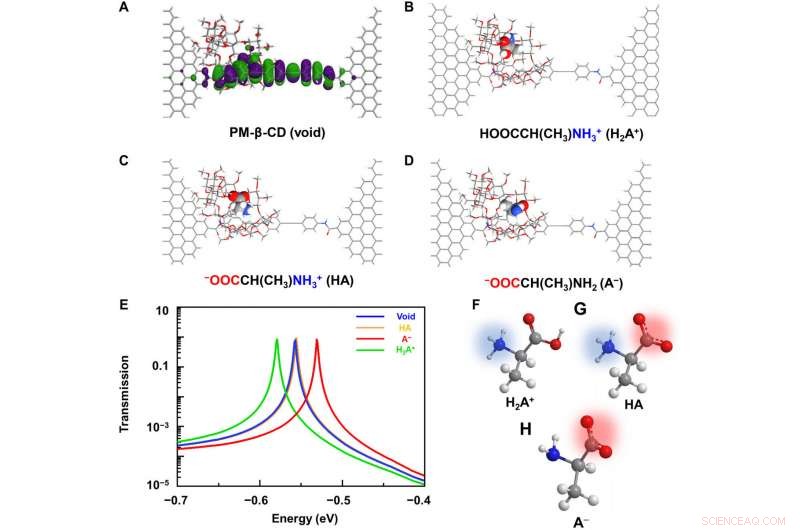
To better understand the correlation between host-guest interactions in the L-alanine based PM-β-CD single molecule junctions, Liu et al. calculated the transmission spectra of the PM-β-CD host with differently charged L-Alanine guests. För att åstadkomma detta, they used a nonequilibrium Green's function technique based on density functional theory as implied in the Atomistix Toolkit package. They noted the conductance contribution from the perturbed highest occupied molecular orbital (p-HOMO) to be dominant at low bias voltages as reflected with transmission spectra. The transmission spectra of the configurations were significantly different near the Fermi level of electrodes to afford different conductance stages. The researchers then analyzed the transitions between each level during amino acid recognition. They used L-alanine as an example to observe reversible transitions from the dissociation to form cations, zwitterions, and anion forms. The dissociation level dominated the device conductance, and the team collected the information using a four-state model, which described the transitions to understand the capacity to detect different structures of amino acids when interacting with PM-β-CD. The amino acids had at least two association processes for the carboxylic group and the other amino group. The greater the number of states recorded, the more accurate the recognition of results were.
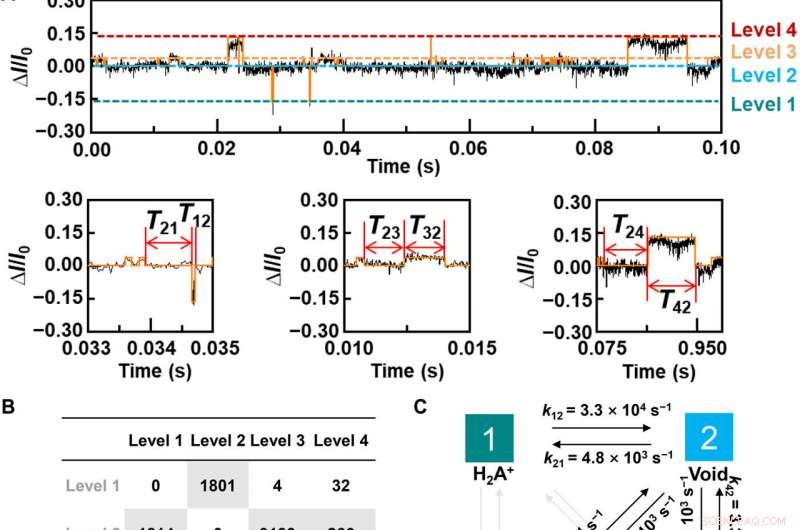
Dynamic analysis for L-Alanine detection. (A) Plot of ΔI/I0 as a function of time during the host-guest interaction. The red curve is the idealized fit by using a QuB software. pH =7, T =298 K, and the bias voltage =0.1 V. (B) Transition statistics between each state. The horizontal columns mean the initial state. (C) Kinetic model for the L-Ala recognition process. (D to I), Plots of time intervals of (D) level 1 to level 2, (E) level 2 to level 1, (F) level 2 to level 3, (G) level 3 to level 2, (H) level 2 to level 4, and (I) level 4 to level 2 at 298 K. Credit:Science Advances, doi:10.1126/sciadv.abe4365
Enantiomer recognition and outlook
Due to the similarity between enantiomers, it was more challenging to differentiate the structures compared to species identification. The associated energy differences were small and close to computational errors. To overcome these complexities, the team established a distinct "fingerprint database" for each amino acid to compare the conductance and kinetic data of different enantiomers. The team then achieved the current change and relaxation time data and compared them with the fingerprint database. Due to this universality, the method can also recognize chiral functional drug molecules to establish a broad approach for biomolecular detection at the single molecule-level.
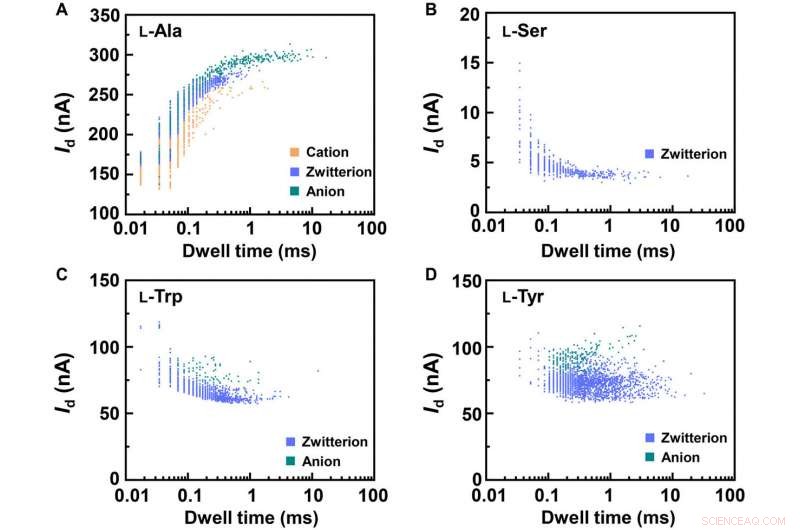
Statistical analyses of the translocation events for different amino acids. Translocation events (current as a function of the dwell time) were analyzed for (A) L-Ala, (B) L-Ser, (C) L-Trp, and (D) L-Tyr, respektive. The two-dimensional (2D) contour plots are composed of all the events in 10-s recording at pH =7, showing the capability of detecting the different structures of amino acids when interacting with PM-β-CD. Kredit:Science Advances, doi:10.1126/sciadv.abe4365
På det här sättet, Zihao Liu and colleagues presented a practical single-molecule approach for real-time electrical recognition of amino acids with different structures and chirality within microseconds. The host-guest dynamic processes could be observed for diverse amino acids at the level of the anion, zwitterion and cation shuttling. Using the permethylated-β-cyclodextrin (PM-β-CD) complex, the team recognized differently charged states of amino acids based on current fluctuation range and thermodynamic/kinetic parameters. The technique can revolutionize existing methods to accurately sequence single-molecule gene/protein sequencing toward universal applications. The method can also offer a universal tool to recognize many important molecules in environmental or biological systems to understand the basis of life at the molecular level.
© 2021 Science X Network