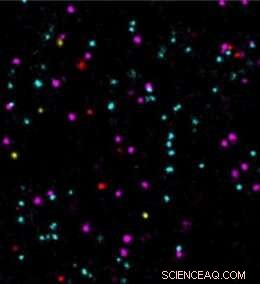
Kredit:S. Strauss/MPI of Biochemistry
Superupplöst fluorescensmikroskopi kan användas för att visualisera strukturer mindre än 200 nanometer, d.v.s. under ljusets diffraktionsgräns. En av mikroskopiteknikerna, kallad DNA-PAINT, utvecklades av Ralf Jungmann, forskargruppsledare vid MPI i biokemi och professor för experimentell fysik vid LMU, tillsammans med kollegor. Tekniken använder korta ”bildare”, färgmärkta DNA-strängar som tillfälligt binder till sina målmolekyler på ett komplementärt sätt för att producera nödvändigt "blinkande" för superupplösning av rekonstruktion av bilderna.
"Vi har nyligen förbättrat DNA-PAINT:s traditionellt ganska långsamma förvärvshastighet med en storleksordning genom att optimera DNA-sekvensdesign, "säger Jungmann." Men detta kom på bekostnad av att förlora multiplexing, vilket innebär att flera strukturer i cellen inte kan observeras samtidigt, "tillade Jungmann. Samtidig observation av flera proteiner, dock, är viktigt för bättre förståelse av komplexa signalkaskader mellan tumörer och normala celler.
Denna multiplexeringsförmåga var inte uppnåbar i hastighetsoptimerad DNA-PAINT, eftersom endast en optimerad sekvens med förbättrade hybridiseringsegenskaper var tillgänglig. "Vi frågade oss själva hur man tillåter multiplexerad bildbehandling och på samma gång, öka bildhastigheten ännu mer, "säger Sebastian Strauss, första författare till tidningen och medarbetare i Jungmanns grupp.
I den aktuella studien, forskarna presenterar ett nytt koncept som framgångsrikt har förbättrat bildhastigheten. De utnyttjade det faktum att bildfrekvensen för bilderna till deras målsträngar skalas linjärt med antalet tillgängliga bindningsställen. "Ju mer bindande platser det finns, ju snabbare bildförvärvet fortskrider. Dock, helt enkelt sammankoppling av bindningsställen skulle leda till oönskat långa dockningssekvenser, potentiellt reducerbar bildupplösning och ökande icke-specifik bindning, "säger Strauss. För att kringgå dessa frågor, forskarna utformade repetitiva sekvensmotiv, t.ex. (TCC) n, som skulle kunna sammanfogas för att tillhandahålla överlappande bindningsställen men endast något ökande stränglängd. "Vi designade sex individuella, periodiska sekvensmotiv, vilket gör att vi kan introducera multiplexering till hastighetsoptimerad DNA-FÄRG, "sa Strauss." I kombination med tidigare förbättringar, vi kan nu påskynda DNA-PAINT med en faktor 100, "tillägger Jungmann.
För att optimera de nya sekvensmotiven och jämföra deras förbättringar, gruppen använde DNA origami -strukturer, som är självmonterade, DNA-objekt i nanometerstorlek som autonomt viks till fördefinierade former. Dessa strukturer kan användas för att arrangera DNA-PAINT-bindningsställen med avstånd från varandra exakt på t.ex. 5 nm avstånd. Detta gjorde det möjligt för forskarna att utvärdera förbättringarna av DNA-PAINT under definierade förhållanden. "De nya optimerade DNA -sekvenserna gjorde att vi kunde lösa sex olika DNA -origamistrukturer istället för bara en, på bara några minuter, "förklarar Strauss.
"Vi är glada över att tillämpa den nu ytterligare förbättrade bildhastigheten i DNA-PAINT för att ta itu med biologiska frågor. Till exempel, tumörmarkörer kunde tidigare endast undersökas långsamt och inte tydligt på en enda molekylnivå. I vår studie, mätningen av fyra olika tumörmarkörer bekräftar en snabb och noggrann analys av deras molekylära positioner och interaktioner. Detta kan ge viktiga insikter för läkemedelsutveckling och deras verkningsmekanismer, "avslutar Jungmann.