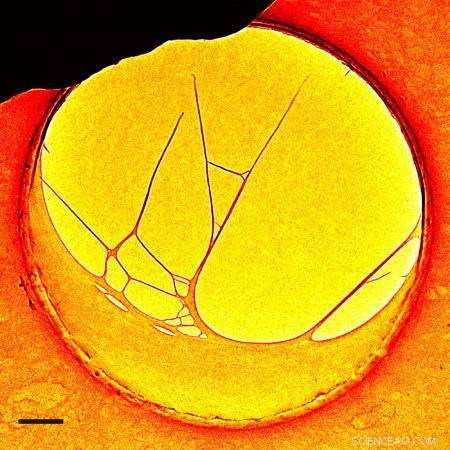
En DNA-struktur sett genom 4D-elektronmikroskopet som uppfanns på Caltech. Kredit:Zewail &Lorenz/Caltech
(Phys.org)—Varje bra struktur, från Empire State Building till Golden Gate Bridge, beror på specifika mekaniska egenskaper för att förbli stark och pålitlig. Styvhet - ett material styvhet - är av särskild betydelse för att bibehålla den robusta funktionaliteten hos allt från kolossala byggnader till de minsta nanoskala strukturer. I biologiska nanostrukturer, som DNA-nätverk, det har varit svårt att mäta denna styvhet, som är väsentligt för deras egenskaper och funktioner. Men forskare vid California Institute of Technology (Caltech) har nyligen utvecklat tekniker för att visualisera beteendet hos biologiska nanostrukturer i både rum och tid, så att de direkt kan mäta styvhet och kartlägga dess variation i hela nätverket.
Den nya metoden beskrivs i den tidiga upplagan av den 4 februari Proceedings of the National Academy of Sciences ( PNAS ).
"Den här typen av visualisering tar oss in i domäner inom de biologiska vetenskaperna som vi inte utforskat tidigare, säger Nobelpristagaren Ahmed Zewail, Linus Pauling professor i kemi och professor i fysik vid Caltech, som skrev tidningen tillsammans med Ulrich Lorenz, en postdoktor i Zewails laboratorium. "Vi tillhandahåller metoden för att ta reda på - direkt - styvheten hos ett biologiskt nätverk som har egenskaper i nanoskala."
Att känna till de mekaniska egenskaperna hos DNA -strukturer är avgörande för att bygga robusta biologiska nätverk, bland andra applikationer. Enligt Zewail, denna typ av visualisering av biomekanik i rum och tid bör vara tillämplig på studier av andra biologiska nanomaterial, inklusive de onormala proteinsammansättningarna som ligger bakom sjukdomar som Alzheimers och Parkinsons.
Zewail och Lorenz kunde se, för första gången, rörelsen av DNA-nanostrukturer i både rum och tid med hjälp av det fyrdimensionella (4D) elektronmikroskopet som utvecklats vid Caltechs Physical Biology Center for Ultrafast Science and Technology. Centret regisseras av Zewail, som skapade den 2005 för att främja förståelsen av den grundläggande fysiken för kemiskt och biologiskt beteende.
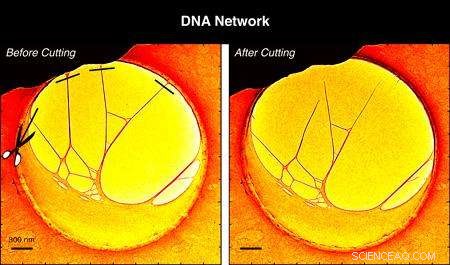
Med hjälp av elektronerna i mikroskopet, flera DNA-filament skars bort från kolfilmen (till vänster) så att en tredimensionell, fristående struktur uppnåddes under 4D-mikroskopet (höger).
"I naturen, materiens beteende bestäms av dess struktur – arrangemangen av dess atomer i rymdens tre dimensioner – och av hur strukturen förändras med tiden, den fjärde dimensionen, " förklarar Zewail. "Om du ser en häst galoppera i slow motion, du kan följa galoppens tid, och du kan se i detalj vad, till exempel, varje ben klarar sig över tiden. När vi kommer till nanometerskalan, det är en annan historia - vi måste förbättra den rumsliga upplösningen till en miljard gånger hästens för att visualisera vad som händer. "
Zewail tilldelades 1999 Nobelpriset i kemi för sin utveckling av femtokemi, som använder ultrakorta laserblixtar för att observera fundamentala kemiska reaktioner som inträffar på femtosekundens tidsskala (en miljondels miljarddels sekund). Även om femtokemi kan fånga atomer och molekyler i rörelse, ger tidsdimensionen, den kan inte samtidigt visa utrymmets dimensioner, and thus the structure of the material. This is because it utilizes laser light with wavelengths that far exceed the dimension of a nanostructure, making it impossible to resolve and image nanoscale details in tiny physical structures such as DNA .
To overcome this major hurdle, the 4D electron microscope employs a stream of individual electrons that scatter off objects to produce an image. The electrons are accelerated to wavelengths of picometers, or trillionths of a meter, providing the capability for visualizing the structure in space with a resolution a thousand times higher than that of a nanostructure, and with a time resolution of femtoseconds or longer.
The experiments reported in PNAS began with a structure created by stretching DNA over a hole embedded in a thin carbon film. Using the electrons in the microscope, several DNA filaments were cut away from the carbon film so that a three-dimensional, free-standing structure was achieved under the 4D microscope.
Nästa, the scientists employed laser heat to excite oscillations in the DNA structure, which were imaged using the electron pulses as a function of time—the fourth dimension. By observing the frequency and amplitude of these oscillations, a direct measure of stiffness was made.
"It was surprising that we could do this with a complex network, " says Zewail. "And yet by cutting and probing, we could go into a selective area of the network and find out about its behavior and properties."
Using 4D electron microscopy, Zewail's group has begun to visualize protein assemblies called amyloids, which are believed to play a role in many neurodegenerative diseases, and they are continuing their investigation of the biomechanical properties of these networks. He says that this technique has the potential for broad applications not only to biological assemblies, but also in the materials science of nanostructures.