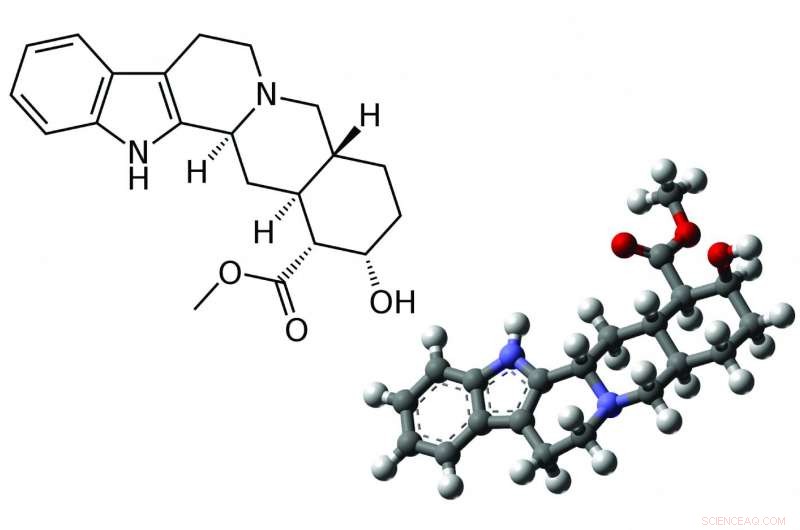
Ett datorprogram kan ta reda på 3D-strukturen (höger) av molekyler som Yohimbine med en knapptryckning. Molekyler med samma 2D-struktur (vänster) kan ha olika 3D-strukturer. Kredit:2D-struktur:acdx (Wikipedia)3D-struktur:MindZiper (Wikipedia)
Ett internationellt team av forskare under ledning av Carnegie Mellon University-kemisten Roberto R. Gil och Universidade Federal de Pernambuco-kemisten Armando Navarro-Vázquez har utvecklat ett program som automatiserar processen att ta reda på en molekyls tredimensionella struktur. Tekniken, beskrivs i en tidning i Angewandte Chemie , komprimerar en process som vanligtvis tar dagar in i minuter och som kan förkorta utvecklingen av läkemedel genom att minska mänskliga fel.
Att ta reda på den kemiska strukturen hos en molekyl från grunden är en viktig del av forskningen om kemikalier som kommer från naturen, eller "naturprodukter". För substanser med möjlig farmaceutisk användning, den strukturen kan avslöja hur ämnet kan interagera med människokroppen.
"Om molekylen ska vara en drog, du behöver veta formen på molekylen för att veta hur den kommer att interagera med en receptor, säger Gil, en professor vid institutionen för kemi vid Carnegie Mellon's Mellon College of Science.
Det första steget för att bestämma en molekyls struktur är att bestämma dess atomära byggstenar, följt av att ta reda på dess tvådimensionella struktur, som visar hur varje atom är kopplad till varandra. Medan vissa atombindningar är styva, andra kan rotera runt en led, gör det möjligt för molekyler med samma komponenter och tvådimensionella (2D) strukturer att ha olika tredimensionella (3D) former.
Små skillnader i form kan översättas till stora förändringar i hur droger verkar i kroppen. Till exempel, Att rotera en bindning i det populära smärtstillande medlet ibuprofen gör den helt inaktiv. Liknande, stärkelse och cellulosa delar samma 2D-struktur men har olika 3D-former. Den skillnaden är anledningen till att människor kan smälta spannmål och inte ved.
Gil och Navarro-Vázquez har arbetat i åtta år för att förenkla processen att hitta och sortera genom möjliga 3D-former för en given 2D-struktur. Och med laboratorieverktygen för att samla in data om en molekyls 3D-struktur blir mer utbredda och tillgängliga, det var dags att utveckla en metod för att automatisera och effektivisera processen.
Forskarna skapade ett program, skrivet i programmeringsspråket Python, som använder information om Residual Dipolar Coupling (RDC), ett mått på avståndet mellan atomer som sträcker sig från roterande bindningar. Matas med data om en given molekyl från RDC-experiment, programmet genererar möjliga sätt att molekylen kan existera i tre dimensioner, och väljer det mest troliga alternativet.
Tekniken är mest effektiv för att hantera 3D-strukturen hos organiska molekyler som är små till medelstora, och relativt stel, med kolatomer packade i ringar snarare än länkade till långa, böjda kedjor. Teamet testade sitt program på sex sådana molekyler, inklusive naltrexon, ett läkemedel som används för att blockera effekterna av opioider, och stryknin, ett bekämpningsmedel.
Först, de bestämde 2D-strukturen för varje molekyl med hjälp av ett program för datorstödd strukturuppklaring (CASE), med hjälp av ett samarbete med Clemens Anklin, vice ordförande för NMR-applikationer och utbildning på Bruker Corporation. De matade den informationen, tillsammans med RDC-data om molekylen, in i deras nya program. I varje fall, deras program kunde välja ut den korrekta 3D-strukturen.
"Du trycker på en knapp, och med lite mänsklig eller ingen mänsklig inblandning, du går från 2D- till 3D-struktur i ett skott, säger Gil.
Men lika viktigt som programmets hastighet är dess grundlighet.
"Mängden naturliga produkter som rapporteras felaktigt, där den rapporterade strukturen inte överensstämmer med den verkliga strukturen, är riktigt stor, säger Navarro-Vázquez, en kemiprofessor vid Universidade Federal de Pernambuco i Brasilien. Detta program kan vara ett värdefullt sätt att leta efter möjliga strukturer som forskare annars skulle kunna missa, hjälpa dem att undvika sina egna inneboende fördomar.
Gil spårar sin vision av denna process tillbaka till sin tid som doktorand och ett samtal med en mentor för 30 år sedan om idén att kemister en dag skulle kunna stoppa in ett ämne i en maskin och se dess struktur med en knapptryckning. Snart, han hoppas, program som hans och Navarro-Vázquez skapade kommer att integrera de tidigare stegen i processen, föra den visionen ännu närmare verkligheten.
"Vi är väldigt nära, " säger Gil. "Detta är drömmen för vilken kemist som helst."