Med hjälp av strukturstyrd Cas13 modifierade forskare vid Rice University och University of Connecticut ett genredigeringsverktyg för att fungera som ett mycket känsligt diagnostiskt test för närvaron av SARS-CoV-2-viruset. De använde en elektrokemisk sensor från hyllan för att leverera resultat. Kredit:Jie Yang/Rice University
En konstruerad CRISPR-baserad metod som hittar RNA från SARS-CoV-2, viruset som orsakar COVID-19, lovar att göra testning för den och andra sjukdomar snabb och enkel.
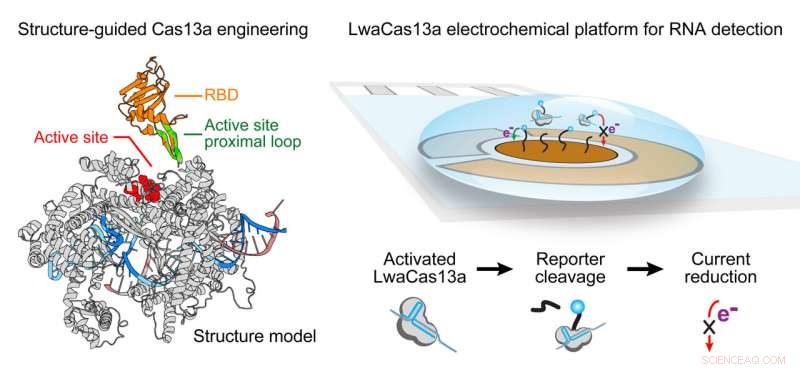
Samarbetspartners vid Rice University och University of Connecticut konstruerade vidare det RNA-redigerande CRISPR-Cas13-systemet för att öka deras kraft för att upptäcka små mängder SARS-CoV-2-virus i biologiska prover utan det tidskrävande RNA-extraktions- och amplifieringssteget som krävs i guldstandard PCR-testning.
Den nya plattformen var mycket framgångsrik jämfört med PCR, och hittade 10 av 11 positiva och inga falskt positiva för viruset i tester på kliniska prover direkt från näsprover. Forskarna visade att deras teknik hittar tecken på SARS-CoV-2 i attomolär (10 -18 ) koncentrationer.
Studien ledd av kemi- och biomolekylär ingenjör Xue Sherry Gao vid Rices George R. Brown School of Engineering och postdoktorala forskarna Jie Yang från Rice och Yang Song från Connecticut visas i Nature Chemical Biology .
Cas13, liksom sin mer kända kusin Cas9, är en del av det system genom vilket bakterier naturligt försvarar sig mot invaderande fager. Sedan upptäckten har CRISPR-Cas9 anpassats av forskare för att redigera levande DNA-genom och visar ett stort löfte om att behandla och till och med bota sjukdomar.

Rice Universitys postdoktorala forskare Jie Yang ledde ett försök att anpassa Cas13-genomredigeringsverktyg för att fungera som en mycket känslig detektor för närvaron av SARS-CoV-2-viruset, som orsakar COVID-19. Kredit:Jeff Fitlow/Rice University
Och den kan användas på andra sätt. Cas13 på egen hand kan förbättras med guide-RNA för att hitta och klippa mål-RNA-sekvenser, men också för att hitta "collateral", i detta fall närvaron av virus som SARS-CoV-2.
"Det konstruerade Cas13-proteinet i detta arbete kan lätt anpassas till andra tidigare etablerade plattformar," sa Gao. "Stabiliteten och robustheten hos konstruerade Cas13-varianter gör dem mer lämpade för vårdcentralsdiagnostik i områden med låga resurser när dyra PCR-maskiner inte är tillgängliga."
Yang sa att vildtyp Cas13, hämtad från en bakterie, Leptotrichia wadei, inte kan upptäcka attomolär nivå av viralt RNA inom en tidsram på 30 till 60 minuter, men den förbättrade versionen som skapats på Rice gör jobbet på ungefär en halvtimme och upptäcker SARS -CoV-2 i mycket lägre koncentrationer än de tidigare testerna.
Hon sa att nyckeln är en väl dold, flexibel hårnålsögla nära Cas13s aktiva sida. "Det är i mitten av proteinet nära den katalytiska platsen som bestämmer Cas13s aktivitet," sa Yang. "Eftersom Cas13 är stort och dynamiskt var det utmanande att hitta en sida för att infoga en annan funktionell domän."
Forskarna fusionerade sju olika RNA-bindande domäner till slingan, och två av komplexen var klart överlägsna. När de hittade sina mål fluorescerade proteinerna och avslöjade närvaron av viruset.
"Vi kunde se att den ökade aktiviteten var fem eller sex gånger mer än vildtyp Cas13," sa Yang. "Denna siffra verkar liten, men det är ganska häpnadsväckande med ett enda steg av proteinteknik.
"But that was still not enough for detection, so we moved the whole assay from a fluorescence plate reader, which is quite large and not available in low-resource settings, to an electrochemical sensor, which has higher sensitivity and can be used for point-of-care diagnostics," she said.
With the off-the-shelf sensor, Yang said the engineered protein was five orders of magnitude more sensitive in detecting the virus compared to the wild-type protein.
The lab wants to adapt its technology to paper strips like those in home COVID-19 antibody tests, but with much higher sensitivity and accuracy. "We hope that will make testing more convenient and with lower cost for many targets," Gao said.
The researchers are also investigating improved detection of the Zika, dengue and Ebola viruses and predictive biomarkers for cardiovascular disease. Their work could lead to rapid diagnosis of the severity of COVID-19.
"Different viruses have different sequences," Yang said. "We can design guide RNA to target a specific sequence that we can then detect, which is the power of the CRISPR-Cas13 system."
But because the project began just as the pandemic took hold, SARS-CoV-2 was a natural focus. "The technology is quite amenable to all the targets," she said. "This makes it a very good option to detect all kinds of mutations or different coronaviruses."
"We are very excited about this work as a combinational effort of structure biology, protein engineering and biomedical device development," Gao added. "I greatly appreciate all the efforts from my lab members and collaborators."
Co-authors of the paper are Rice postdoctoral researcher Xiangyu Deng, undergraduate Jeffrey Vanegas and graduate student Zheng You; graduate students Yuxuan Zhang and Zhengyan Weng of the University of Connecticut; microbiology supervisor Lori Avery and Kevin Dieckhaus, a professor of medicine, of UConn Health; Yi Zhang, an assistant professor of biomedical engineering at the University of Connecticut; and Yang Gao, an assistant professor of biosciences at Rice. + Utforska vidare