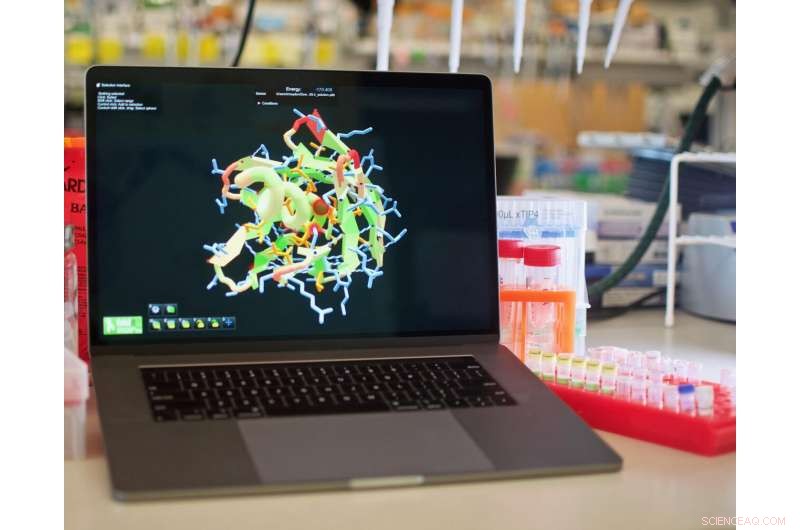
Genom att spela videospelet Foldit, pusselentusiaster kan nu designa proteiner som aldrig funnits tidigare, och hjälp med att skapa nya vacciner och behandlingar för cancer och andra allvarliga sjukdomar. Kredit:Institute for Protein Design/UW Medicine
Ett team av forskare kodade in sina specialkunskaper i datorspelet Foldit för att göra det möjligt för medborgarforskare att framgångsrikt designa syntetiska proteiner för första gången.
De första resultaten av detta samarbete visas i 5 juni -numret av Natur . Institute for Protein Design vid University of Washington School of Medicine ledde den multiinstitutionella insatsen.
"Det finns fler möjliga proteiner än det finns atomer i universum. Det är spännande att tänka att nu kan vem som helst hjälpa till att utforska detta stora utrymme av möjligheter, "sa senior medförfattare David Baker, professor i biokemi vid UW School of Medicine och chef för Institute for Protein Design.
"Mångfalden av molekyler som dessa spelare kom på är häpnadsväckande, "sa huvudförfattaren Brian Koepnick, postdoktor vid Institute for Protein Design. "Dessa nya proteiner är ingalunda sämre än de saker som en forskare på doktorandnivå kan göra."
Foldit skapades 2008 som ett sätt att 'gamifiera' proteinforskning. Proteiner är viktiga biomolekyler som finns i varje cell i varje organism. Deras invecklade tredimensionella strukturer ger upphov till deras olika funktioner, som inkluderar matsmältning, sårläkning, autoimmunitet och mycket mer.
Genom gameplay, Foldit-spelare har hjälpt till att bestämma strukturen för ett HIV-relaterat protein och förbättrat aktiviteten hos användbara enzymer. Tills nu, dock, Foldit -spelare kunde bara interagera med proteiner som redan fanns. Det fanns inget sätt att designa nya.
"Att designa helt nya proteiner som inte fanns i naturen har varit vårt mål med Foldit länge, "sa senior medförfattare Seth Cooper, biträdande professor vid Khoury College of Computer Sciences vid Northeastern University. "Denna nya uppsättning resultat visar att det är möjligt."
För att göra Foldit till en plattform för proteindesign, forskarna kodade in biokemisk kunskap i spelet. På det sättet, designermolekyler som fick bra poäng i Foldit skulle vara mer benägna att fälla upp som avsett i den verkliga världen.
"Vi höll inte [Foldit -spelare] några föreläsningar eller sa åt dem att läsa någonting. I stället vi justerade koden som har kört spelet under många år, "sa senior medförfattare Firas Khatib, biträdande professor i datavetenskap vid University of Massachusetts Dartmouth.
Forskarna testade 146 proteiner som designats av Foldit -spelare i laboratoriet. 56 befanns vara stabila. Denna upptäckt föreslog att spelarna hade producerat några realistiska proteiner. Forskarna samlade in tillräckligt med data om fyra av dessa nya molekyler för att visa att konstruktionerna antog sina avsedda strukturer.
"Jag hade aldrig trott att de skulle få det så bra, men Foldits spelare slutar aldrig att förvåna oss. "sa Khatib.
Att tillverka nya proteiner är lite som att försöka knyta aldrig tidigare skådade knutar med ett rep som är en miljon gånger tunnare än ett människohår. Hittills, bara en liten grupp experter med intim kunskap om hur biomolekyler vrider och vänder stör sig på denna oerhört komplexa uppgift. De flesta använder automatiserade algoritmer för molekylär design, och de flesta designalgoritmer misslyckas mycket oftare än de lyckas.
"Vi försöker alltid göra algoritmerna bättre, men det mänskliga elementet är nyckeln, "sa Khatib." Faktum är att genom Foldit -design, spelare har till och med upptäckt brister i Rosetta energifunktion-vår toppmoderna metod för proteindesign. "
Proteindesign är en framväxande vetenskaplig disciplin. Under de senaste fem åren har experter vid Institute for Protein Design och deras kollegor har skapat proteiner som stimulerar immunsystemet att bekämpa cancer och andra som fungerar som potenta vaccinkandidater. I april, Institute for Protein Design fick ett åtagande på 45 miljoner dollar i finansiering genom The Audacious Project, ett filantropiskt samarbete som organiseras av TED, att designa proteinbaserade vacciner, läkemedel och material.
Kan spelare skapa nästa blockbuster -läkemedel?
"Foldit -spelare är ett nytt tillskott till forskningsarsenalen, "sa Khatib." De är inte en silverkula, men de är en fantastisk resurs. "
Alla som är intresserade av att hjälpa till att lösa pussel för vetenskap kan lära sig mer på fold.it/portal/