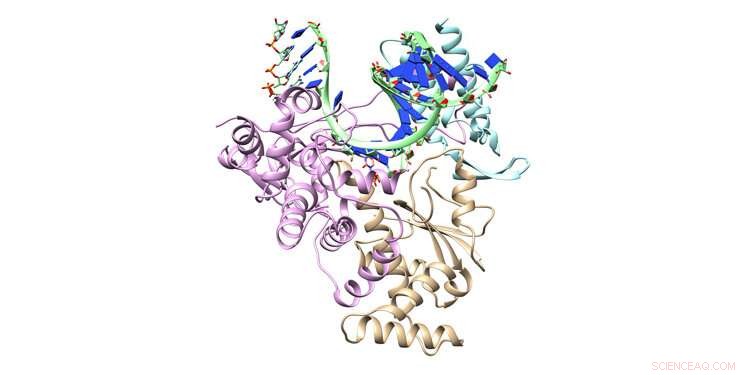
Antiviralt läkemedel remdesivir utgör huvudlinjen i FDA-godkänt terapeutiskt försvar mot COVID-19-viruset. Forskare vid University of North Texas använder Frontera superdator för att modellera hur remdesivir blockerar reproduktion av coronaviruset, i hopp om att utveckla förbättringar av läkemedlet. Här visas kristallstrukturerna för den RNA-beroende RNA-polymeras ternära komplexmodellen med dubbelsträngat RNA och inkommande remdesivirtrifosfat. Kredit:Cisneros Research Group, UNT
Nyckelproteiner som används av coronaviruset för dess reproduktion modelleras på NSF-finansierad Frontera superdator av Andres Cisneros forskargrupp vid University of North Texas. Forskningsmål inkluderar att hitta sätt att förbättra det terapeutiska remdesivir för covid-19. NSF-finansierad Frontera-tilldelning tilldelas Cisneros genom COVID-19 High Performance Computing Consortium.
I maj 2020, US Food and Drug Administration godkände det antivirala läkemedlet remdesivir för akut behandling av covid-19, en av endast fyra läkemedel som för närvarande har denna status. Remdesivir stoppar det kemiska maskineriet som coronaviruset använder för att kopiera sig själv, binder till ett enzym som gör sammansättningen. Medan remdesivir har visat lovande när det gäller att hjälpa patienter att återhämta sig från covid-19, forskare undersöker sätt att förbättra dess effektivitet.
Ett team av forskare ledda av G. Andres Cisneros från University of North Texas modellerar nyckeldelarna av coronaviruset som det använder för att kopiera sig själv. Simuleringarna görs på superdatorer Stampede2 och Frontera vid Texas Advanced Computing Center (TACC).
"Vi hade mycket tur att få en tilldelning på Frontera för att kunna arbeta med att undersöka mekanismen för läkemedel som riktar sig mot två specifika proteiner i COVID-19, " sa Cisneros. Hans arbete undersöker hur remdesivir och andra tillgängliga läkemedel hämmar proteinerna NSP-12 och huvudproteaset, båda enzymerna som coronaviruset behöver för replikering. "Genom att titta på hur dessa droger fungerar, kanske kan denna information användas för att förbättra dem."
NSP-12-proteinet sätter ihop nukleotiderna som utgör viralt RNA, förkortas som A, U, G, och C, bygga kompletta uppsättningar genetiskt material för nya coronavirus -kopior. NSP-12 är faktiskt en del av en större struktur som kallas det RNA-beroende RNA-polymeraset (RDRP) som kopierar hela RNA:t. Remdesivir binder till RDRP, koppla ihop maskineriet.
"Vi undersöker hur den här processen går till, " sade Cisneros. "Genom att göra detta, kanske det kan finnas ett sätt för oss och andra forskare att komma med idéer om huruvida och hur remdesivir kan förbättras."
Det andra proteinet Cisneros studerar kallas huvudproteaset. Det separerar ett polyprotein som produceras av SARS-CoV-2 översatt från viralt RNA till funktionella proteiner som sätter "kött" på dess virala ben. Stoppa proteasen, och du stoppar viruset från att bildas. Detta gör det till ett utmärkt läkemedelsmål.
Cisneros förklarade att han använder den grundläggande matematiken och fysiken i Newtons ekvationer och kvantmekanik för att beräkna egenskaperna hos proteinerna, inklusive allt som är relevant för dess funktion, såsom RNA och vatten. Ett tillvägagångssätt som kallas klassisk molekylär dynamik använder Newtons ekvationer för att simulera hur proteinerna rör sig och interagerar dynamiskt i tiden. "Vi pratar om system som vi simulerar som finns i hundratusentals atomer, " sa Cisneros.
Han simulerar också de kemiska reaktionerna inuti proteinerna för att undersöka hur läkemedlen stoppar RDRP eller proteaset. En hybridmetod som kallas QM/MM (kvantmekanik/molekylär mekanik) sparar beräkningstid och pengar genom att fokusera mer intensivt på interaktioner på den aktiva platsen, använder den mer ungefärliga raka molekylära dynamiken för allt annat.
Cisneros-gruppen utvecklade och underhåller ett program som heter LICHEM som låter dem använda QM/MM-metoden. "En av egenskaperna hos LICHEM är att den tillåter oss att använda tillvägagångssätt för den klassiska mekanikdelen som inkluderar en bättre beskrivning av fysiken som händer mellan molekylerna i den klassiska miljön, specifikt, AMOEBA-potentialen "sa Cisneros. AMOEBA är utvecklat av Pengyu Ren från UT Austin, Jay Ponder från University of Washington; och Jean-Philip Piquemal vid Sorbonne University i Paris med bidrag från Cisneros-gruppen för joniska vätskor.
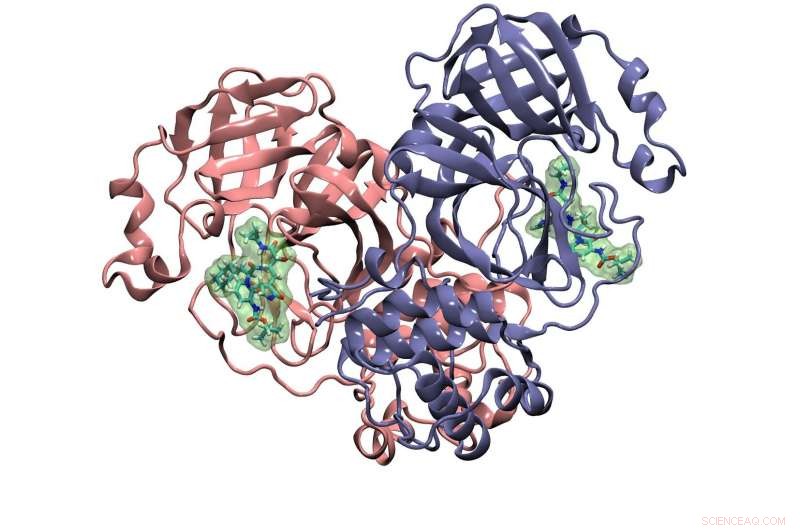
Ett annat forskningsmål för Cisneros-gruppen som modelleras på Frontera är ett protein som kallas huvudproteaset. Det klyver ett polyprotein som produceras av viruset som bygger upp de funktionella proteinerna i kopiorna av sig själva som det genererar. Kristallstruktur av coronavirusets huvudproteas med bunden inhibitor som visas här. Kredit:Cisneros Research Group, UNT
"Frontera, med inte bara beräkningskraft utan kommunikationen mellan noderna, tillåter oss att köra dessa QM/MM-beräkningar med mycket högre, inte bara hastighet, men också genomströmning, "Sa Cisneros. Frontera befriade dem att köra flera system åt gången." I min grupp, Jag har fem olika forskare, doktorander och post docs, som arbetar på båda dessa system, men i olika pusselbitar. Alla har tillgång till dessa resurser. Det är definitivt mycket användbart, och vi uppskattar mycket tilldelningen."
Det som fick Cisneros att gå var nyheter i april 2020 om kristallstrukturen för SARS-CoV-2 RDRP som rapporteras. "Jag kontaktade min grupp och berättade att med denna information, det finns något vi kan göra för att hjälpa till med pandemin, " han sa.
Inom två dagar efter nyheten, Cisneros föreslog framgångsrikt sin forskning om läkemedelsmål för coronavirus till COVID-19 High Performance Computing Consortium. Dussintals nationella och internationella superdatoranläggningar, industri, och organisationer inklusive TACC har frivilligt lämnat sina resurser till konsortiet för att stödja forskarnas ansträngningar att bekämpa coronaviruset.
Tilldelningen tilldelades ursprungligen bara på TACC:s Stampede2, Supercomputing flaggskeppet för National Science Foundation (NSF) som är rankad 21:a snabbast i världen och #2 för akademiska system enligt Top500. "Sedan blev vi kontaktade av TACC och tacksamma för att vi fick tillgång till Frontera. Nu har vi tillgång till båda systemen, vilket är riktigt bra, " sa Cisneros.
Fronteras superdator är den #1 snabbaste akademiska superdatorn och #8 snabbaste i världen. Både Frontera och Stampede2 finansieras av NSF.
"Vi är mycket nöjda med det här systemet. Vi kunde överföra en del av den kunskap vi hade från Stampede2 till Frontera, " sa Cisneros. En av hans nyligen utexaminerade studenter, Erik Vazquez Montelongo, ställa in alla beräkningar för LICHEM på Frontera baserat på vad han lärde sig på Stampede2. "Det har verkligen varit en välsignelse. Frontera för våra beräkningar har fungerat riktigt bra. Vi är verkligen nöjda med det."
En av postdoktorerna i The Cisneros Group, Sehr Nazeem-Kahn, skapade modellen för RDRP, remdesivir och andra läkemedelskandidater, allt på den aktiva sidan. Med det i handen, de började köra simuleringar.
"Vi var mycket glada över att se att hennes modell faktiskt var väldigt nära den experimentella strukturen. Det är verkligen användbart för oss, eftersom det validerar modellen som har byggts av gruppen och visar att vi är på rätt väg, " han lade till.
För närvarande, Dr. Naseem-Khan kör molekylära dynamiksimuleringar av denna modell med remdesivir på Frontera. "Vi börjar också med våra QM/MM -beräkningar för RDRP. I fallet med huvudproteaset, det fanns strukturer som också behövde modelleras och som därefter bekräftades. Det var också mycket tillfredsställande, " sa Cisneros.
Med den strukturdata, de tittar på sex olika inhibitormolekyler. "En sån, vi börjar redan QM/MM-beräkningar på Frontera, och ytterligare en på Stampede2, " sa Cisneros. Om allt går bra, han hoppas få resultat inom de kommande fem till sex månaderna. "Det här är mycket dyra beräkningar, " lade han till. "Också, att köra analysen tar tid. Om vi bara skulle använda resurserna hemma, det skulle ta flera år."