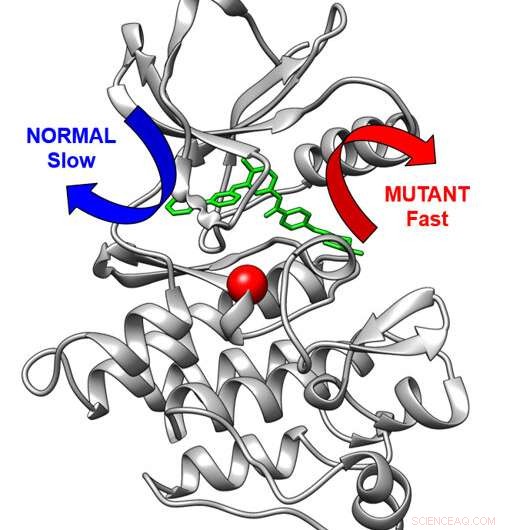
Tredimensionell struktur av cancerläkemedlet Abl-kinas (grå) bundet till anti-cancerläkemedlet imatinib. Normalt går läkemedlet ut långsamt via den blå pilen. En modifiering av kinaset (röd sfär) gör att läkemedlet går ut via en snabb väg (röd pil). Kredit:Aziz M. Rangwala
Att förstå varför och hur resistens mot kemoterapi uppstår är ett stort steg mot att optimera behandlingar för cancer. Ett team av forskare inklusive Markus Seeliger, Ph.D., från Stony Brook Cancer Center och Renaissance School of Medicine vid Stony Brook University, tror att de har hittat en ny process genom vilken läkemedelsresistens uppstår. De använder en datorsimuleringsmodell som hjälper dem att förstå exakt hur molekyler interagerar med cancerläkemedlet Imatinib (känd som Gleevec) i den kemoterapiresistenta processen. Imatinib behandlar kronisk myeloid leukemi (KML) mycket effektivt, men många patienter i sent stadium upplever läkemedelsresistens, vilket gör läkemedlet minimalt effektivt i det skedet.
Forskningen belyses i en artikel publicerad i Angewandte Chemie och bygger på tidigare forskning detaljerad 2021 i PNAS .
Imatinib hämmar BCR-Abl-proteinkinaset, ett alltför aktivt cellulärt signaleringsmaskineri vid KML. I PNAS studie visade forskare att variationer i byggplanen för kinaset kan göra det svårare för Imatinib att binda till kinaset och även påskynda läkemedelsfrisättningen från kinaset. I Angewandte Chemie uppsatsen tog forskargruppen den beräkningsmetod – utvecklad av medförfattaren Pratyush Tiwary från University of Maryland – som gjorde det möjligt för dem att studera den mycket långsamma frisättningen av imatinib från kinaset.
"Denna metod i sig är en stor teknisk prestation som utökar beräkningsförmågan för forskning om läkemedelsresistens, och viktigast av allt ledde till att vi kunde förutsäga hur snabbt friska och mutanta proteiner skulle frigöra detta läkemedel", säger Seeliger, docent vid institutionen för farmakologisk Vetenskaper. "För första gången kunde vi se frisättningen av ett läkemedel från ett protein så detaljerat och exakt. Dessutom kunde vi visa att mutationen förändras fundamentalt inom läkemedlets utträdesväg från proteinet.
"Detta är viktigt eftersom hastigheten på läkemedelsfrisättningen kan vara lika viktig för den terapeutiska effekten av ett läkemedel som hur hårt ett läkemedel binder till proteinet."
Seeliger förklarar vidare att metoden kan ge en grund för att förstå de molekylära mekanismerna bakom kemoterapiresistens.
Mer allmänt är implikationerna av vad de upptäckte att om forskare kan förstå hur läkemedel frigörs från deras proteiner, kan de kanske utforma läkemedel med en långsammare frisättning och högre terapeutisk effekt. Dessutom, om snabb läkemedelsfrisättning kan orsaka läkemedelsresistens, och läkare kan visa att detta händer, kan de kanske återaktivera läkemedlets effektivitet genom att be patienten att ta läkemedlet oftare.
Grunden för mutationstestningen via beräkningsmetoden beskrevs i PNAS papper. Seeliger och kollegor testade hur imatinib binder till mutationer hos patienter med imatinib-resistent KML. De fann att majoriteten av mutationerna lätt binder till imatinib, så det ställde frågan hur dessa mutationer orsakar resistens hos patienter? Forskarna identifierade sedan flera mutanter som binder imatinib lätt men de släpper läkemedlet mycket snabbare.
Efter att ha identifierat dessa mutanter med en snabbare frisättning av läkemedel, använde teamet kärnmagnetisk resonans (NMR) och molekylär dynamik för att koppla proteinet till läkemedelsdisassociation - vilket ligger bakom vikten av läkemedelsdisassociationskinetik för läkemedelseffektivitet. Detta gjorde det möjligt för dem att identifiera en ny mekanism för imatinibresistens.
Arbetet som resulterade i uppsatsen publicerad i PNAS involverade samarbetet mellan Seeliger och hans kollegor vid Stony Brook, och forskare vid Memorial Sloan Kettering Cancer Center och vid Goethe-universitetet i Frankfurt, Tyskland.
Forskning som resulterade i den nyare artikeln leddes av Tiwary och kollegor vid University of Maryland, i samarbete med Seeliger och forskare vid Broad Institute vid MIT och Harvard University. + Utforska vidare