NIST-forskare siktar på att förvandla cellen till en biologisk datorfabrik genom att designa och infoga DNA i en cells genom. Cellproteiner skulle producera RNA baserat på DNA:t genom transkription. RNA-strängen skulle sedan vikas, binda till sig själv och delas i två, tack vare en speciell självklyvande sekvens av RNA som kallas ett ribozym. Den resulterande strukturen, en RNA-kretsgrind, skulle bara lossna och utlösa ytterligare kemiska reaktioner under vissa förhållanden. Kredit:N. Hanacek/NIST
Små biologiska datorer gjorda av DNA kan revolutionera sättet vi diagnostiserar och behandlar en mängd sjukdomar när tekniken väl är färdig. En stor stötesten för dessa DNA-baserade enheter, som kan fungera i både celler och flytande lösningar, har dock varit hur kortlivade de är. Bara en användning och datorerna är slut.
Nu kan forskare vid National Institute of Standards and Technology (NIST) ha utvecklat långlivade biologiska datorer som potentiellt kan finnas kvar inuti celler. I en artikel publicerad i tidskriften Science Advances , författarna avstår från det traditionella DNA-baserade tillvägagångssättet och väljer istället att använda nukleinsyra-RNA för att bygga datorer. Resultaten visar att RNA-kretsarna är lika pålitliga och mångsidiga som deras DNA-baserade motsvarigheter. Dessutom kan levande celler skapa dessa RNA-kretsar kontinuerligt, något som inte är lätt möjligt med DNA-kretsar, vilket ytterligare positionerar RNA som en lovande kandidat för kraftfulla, långvariga biologiska datorer.
Ungefär som den dator eller smarta enhet du förmodligen läser detta på, kan biologiska datorer programmeras för att utföra olika typer av uppgifter.
"Skillnaden är att istället för att koda med ettor och nollor skriver du strängar av A, T, C och G, som är de fyra kemiska baserna som utgör DNA", säger Samuel Schaffter, NIST postdoktor och huvudförfattare till studien .
Genom att sätta samman en specifik sekvens av baser till en sträng av nukleinsyra kan forskare diktera vad den binder till. En sträng kan konstrueras för att fästa till specifika bitar av DNA, RNA eller vissa proteiner som är associerade med en sjukdom, och sedan utlösa kemiska reaktioner med andra strängar i samma krets för att bearbeta kemisk information och så småningom producera någon form av användbar utdata.
Den utsignalen kan vara en detekterbar signal som kan hjälpa medicinsk diagnostik, eller det kan vara ett terapeutiskt läkemedel för att behandla en sjukdom.
DNA är dock inte det robustaste materialet och kan snabbt lossna under vissa förhållanden. Celler kan vara fientliga miljöer, eftersom de ofta innehåller proteiner som hackar upp nukleinsyror. Och även om DNA-sekvenser håller kvar tillräckligt länge för att upptäcka deras mål, gör de kemiska bindningarna de bildar dem värdelösa efteråt.
"De kan inte göra saker som att kontinuerligt övervaka mönster i genuttryck. De är en användning, vilket betyder att de bara ger dig en ögonblicksbild," sa Schaffter.
Eftersom det också är en nukleinsyra, delar RNA många av DNA:s problem när det kommer till att vara en biologisk datorbyggsten. Den är mottaglig för snabb nedbrytning, och efter att en sträng kemiskt binder till en målmolekyl är den strängen färdig. Men till skillnad från DNA kan RNA vara en förnybar resurs under rätt förhållanden. För att utnyttja den fördelen behövde Schaffter och hans kollegor först visa att RNA-kretsar, som celler teoretiskt skulle kunna producera, kunde fungera lika bra som den DNA-baserade sorten.
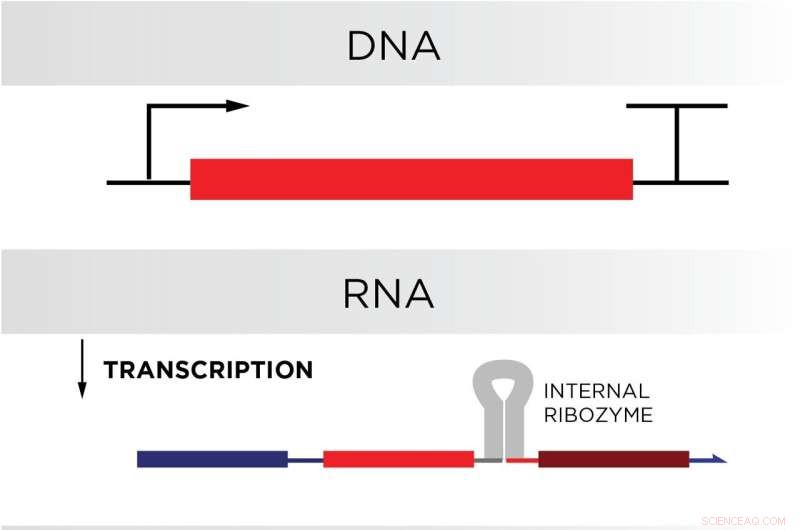
RNA:s kant över DNA härrör från en naturlig cellulär process som kallas transkription, där proteiner producerar RNA på en kontinuerlig basis med hjälp av en cells DNA som mall. Om DNA:t i en cells genom kodade för kretskomponenterna i en biologisk dator, skulle cellen producera datorkomponenterna kontinuerligt.
I den biologiska beräkningsprocessen kan enstaka strängar av nukleinsyror i en krets lätt hamna bundna till andra strängar i samma krets, en oönskad effekt som förhindrar kretskomponenter från att binda till sina avsedda mål. Utformningen av dessa kretsar innebär ofta att olika komponenter blir naturliga passningar för varandra.
För att förhindra oönskad bindning syntetiseras DNA-sekvenser som ingår i datorer som kallas strängförskjutningskretsar vanligtvis (i maskiner snarare än celler) separat och i en dubbelsträngad form. Med varje kemisk bas på varje sträng bunden till en bas på den andra, fungerar denna dubbelsträng som en låst grind som bara skulle låsas upp om målsekvensen kom och tog platsen för en av strängarna.
Schaffter och Elizabeth Strychalski, ledare för NIST:s Cellular Engineering Group och medförfattare till studien, försökte efterlikna denna "låsta grind"-funktion i deras RNA-krets, med tanke på att celler i slutändan skulle behöva producera dessa låsta grindar själva. För att förbereda celler för framgång skrev forskarna sekvenserna så att ena halvan av strängarna kunde binda jämnt med den andra halvan. Bindande på detta sätt skulle RNA-sekvenser vikas på sig själva som en korvbulle, vilket säkerställer att de är i ett låst tillstånd.
Men för att fungera korrekt skulle portarna behöva vara två kemiskt bundna men distinkta trådar, mer som en hamburgerbulle eller smörgås än en korvbulle. Teamet fick den dubbelsträngade designen i sina grindar genom att koda in en RNA-sträcka som kallas ett ribozym nära grindarnas vikningspunkt. Detta speciella ribozym – taget från arvsmassan av ett hepatitvirus – skulle bryta av sig själv efter att RNA-strängen den var inbäddad i veckad och skapat två separata strängar.
Författarna testade om deras kretsar kunde utföra grundläggande logiska operationer, som att bara låsa upp sina grindar under specifika scenarier, som om en av två specifika RNA-sekvenser var närvarande eller bara om båda var samtidigt. De byggde och undersökte också kretsar gjorda av flera grindar som utförde olika logiska operationer i serie. Först när dessa kretsar stötte på den rätta kombinationen av sekvenser, låstes deras grindar upp en efter en som dominobrickor.
Experimenten involverade att exponera olika kretsar för bitar av RNA - av vilka några kretsarna var designade för att fästa vid - och mäta uteffekten från kretsarna. I det här fallet var utsignalen i slutet av varje krets en fluorescerande reportermolekyl som skulle lysa upp när den sista grinden låstes upp.
Forskarna spårade också hastigheten med vilken grindarna låstes upp när kretsarna bearbetade indata och jämförde deras mätningar med förutsägelser från datormodeller.
"För mig behövde dessa fungera i ett provrör lika prediktivt som DNA-beräkningar. Det fina med DNA-kretsar är för det mesta, du kan bara skriva ut en sekvens på ett papper, och det kommer att fungera som du vill," sa Schaffter. "Det viktiga här är att vi fann att RNA-kretsarna var mycket förutsägbara och programmerbara, mycket mer än jag trodde att de skulle vara, faktiskt."
Likheterna i prestanda mellan DNA- och RNA-kretsar kan tyda på att det kan vara fördelaktigt att byta till den senare, eftersom RNA kan transkriberas för att fylla på en krets komponenter. Och många befintliga DNA-kretsar som forskare redan har utvecklat för att utföra olika uppgifter skulle teoretiskt kunna bytas ut mot RNA-versioner och bete sig på samma sätt. För att vara säker, men författarna till studien måste driva tekniken ytterligare.
I denna studie visade författarna att transkriberbara kretsar fungerar, men de har inte producerat dem med det verkliga cellulära maskineriet för transkription ännu. Istället syntetiserade maskiner nukleinsyrorna genom en process liknande den som användes för att producera DNA för forskning. Att ta nästa steg skulle kräva att DNA infogas i genomet av en organism, där det skulle fungera som en ritning för RNA-kretskomponenter.
"Vi är intresserade av att lägga dessa i bakterier härnäst. Vi vill veta:Kan vi paketera kretsdesign i genetiskt material med vår strategi? Kan vi få samma typ av prestanda och beteende när kretsarna är inuti celler?" sa Schaffter. "Vi har potential att." + Utforska vidare