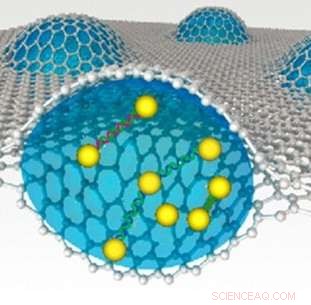
Detta schema över en vätskecell av grafen visar flera vätskefickor som innehåller enstaka nanopartiklar, dimerer sammansatta av dsDNA-bryggor i olika längder, och trimers.
(Phys.org) —Hösten är vanligtvis inte en så bra tid för stora specialeffektfilmer eftersom sommarens storfilmer har bleknat och de för semesterperioden ännu inte har öppnat. Hösten är oftare tid för eftertänksamma filmer om små ämnen, vilket gör den perfekt för avtäckningen av en ny film producerad av forskare vid U.S. Department of Energy (DOE) Lawrence Berkeley National Laboratory (Berkeley Lab). Genom en kombination av transmissionselektronmikroskopi (TEM) och deras egen unika grafenvätskecell, forskarna har registrerat den tredimensionella rörelsen av DNA kopplat till guld nanokristaller. Det är första gången TEM används för dynamisk 3D-avbildning av så kallade mjuka material.
"Vår demonstration av dynamisk 3D-avbildning går utöver TEM:s konventionella användning för att se platt, torra prover och öppnar många spännande möjligheter för att studera dynamiken i biologiska makromolekylära sammansättningar och artificiella nanostrukturer, " säger fysiker Alex Zettl, en av ledarna för denna forskning. "Dessa resultat möjliggjordes av vår nya grafenvätskecell, som kan möta utmaningarna med att använda TEM för att avbilda mjuka material."
Zettl, som har gemensamma möten med Berkeley Labs materialvetenskapsavdelning och UC Berkeleys fysikavdelning där han leder Center of Integrated Nanomechanical Systems, är en av medförfattarna till en tidning i NANO bokstäver beskriver denna forskning. Uppsatsen har titeln "3D Motion of DNA-Au Nanoconjugates in Graphene Liquid Cell Electron Microscopy."
Paul Alivisatos, Berkeley Lab Director och UC Berkeleys Samsung Distinguished Chair in Nanoscience and Nanotechnology, är motsvarande författare. Andra författare är Qian Chen, Jessica Smith, Jungwon Park, Kwanpyo Kim, Davy Ho och Haider Rasool.
Termen "mjuka material" innehåller en mängd olika saker, inklusive DNA, proteiner och andra biologiska föreningar, plast, terapeutiska läkemedel, flexibel elektronik, och vissa typer av solceller. Trots deras allestädes närvarande närvaro i våra dagliga liv, mjuka material ställer många frågor eftersom studiet av deras dynamik på nanoskala, speciellt biologiska system, har varit en utmaning. TEM, där en elektronstråle snarare än ljus används för belysning och förstoring, ger upplösningen för sådana studier men kan bara användas i högvakuum eftersom molekyler i luften stör elektronstrålen. Eftersom vätskor avdunstar i högvakuum, prover av mjuka material, som har beskrivits som "mycket viskösa vätskor, " måste förslutas hermetiskt i speciella solida behållare (kallade celler) med ett visningsfönster innan det avbildas med TEM.
Förr, flytande celler hade kiselbaserade visningsfönster vars tjocklek begränsade upplösningen och störde det naturliga tillståndet hos de mjuka materialen. Zettl och Alivisatos och deras respektive forskargrupper övervann dessa begränsningar med utvecklingen av en flytande cell baserad på ett grafenmembran som endast är en atomtjock. Denna utveckling har gjorts i nära samarbete med forskare vid National Centre for Electron Microscopy (NCEM), som ligger vid Berkeley Lab.
"Våra grafenvätskeceller förde den rumsliga upplösningen av TEM-avbildning i flytande fas till atomskalan men fokuserade fortfarande på tillväxtbanor för metalliska nanokristaller, " säger huvudförfattaren Qian Chen, en postdoktor i Alivisatos forskargrupp. "Nu har vi anammat tekniken för att avbilda 3D-dynamiken hos mjuka material, börjar med dubbelsträngad (dsDNA) kopplad till guld nanokristaller och uppnådde nanometerupplösning."
För att skapa cellen, två motsatta grafenark är bundna till varandra genom sin van der Waals-attraktion. Detta bildar en förseglad nanoskala kammare och skapar inuti kammaren en stabil vattenlösningsficka cirka 100 nanometer i höjd och en mikron i diameter. Det enatomtjocka grafenmembranet i cellerna är i huvudsak transparent för TEM-elektronstrålen, minimerar den oönskade förlusten av avbildningselektroner och ger överlägsen kontrast och upplösning jämfört med kiselbaserade fönster. De vattenhaltiga fickorna tillåter upp till två minuters kontinuerlig avbildning av mjuka materialprover exponerade för en 200 kilo volt avbildande elektronstråle. Under denna tid, mjuka materialprover kan rotera fritt.
Efter att ha visat att deras grafenvätskecell kan försegla en vattenhaltig provlösning mot ett TEM-högvakuum, Berkeley-forskarna använde det för att studera de typer av guld-dsDNA nanokonjugat som har använts i stor utsträckning som dynamiska plasmoniska sonder.
"Närvaron av dubbelsträngade DNA-molekyler innefattar de stora utmaningarna med att studera dynamiken hos biologiska prover med flytande fas TEM, " säger Alivisatos. "De högkontrastiga guldnanokristallerna underlättar spårning av våra exemplar."
Alivisatos- och Zettl-grupperna kunde observera dimerer, par guld nanopartiklar, bundna av ett enda stycke dsDNA, och trimers, tre guld nanopartiklar, ansluten till en linjär konfiguration med två enstaka delar av dsDNA. Från en serie 2D-projicerade TEM-bilder som tagits medan proverna roterade, forskarna skulle rekonstruera 3D-konfigurationen och rörelserna hos proverna när de utvecklades över tiden.
"Denna information skulle vara otillgänglig med konventionella TEM-tekniker, " säger Chen.
Utvecklingen av vätskecellstekniken för in situ TEM, ursprungligen rapporterad i tidskriften Vetenskap under 2012.