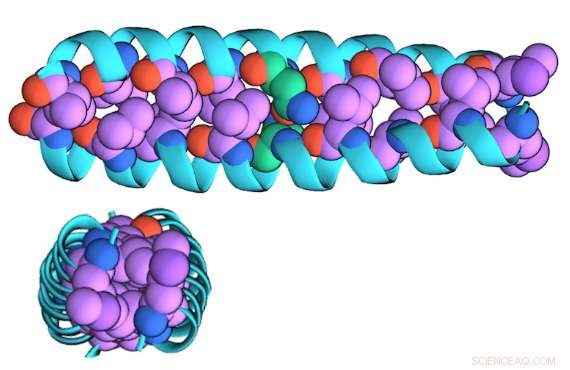
En illustration av den "dragkedjade" dimerformen av GCN4-p1 sett från sidan (överst) och ovanför (botten)
Forskare från University of Pennsylvania har tagit steg mot en ny metod för gensekvensering av en sträng av DNA:s baser läses när de träs genom ett nanoskopiskt hål.
I en ny studie, de har visat att denna teknik också kan tillämpas på proteiner som ett sätt att lära sig mer om deras struktur.
Befintliga metoder för denna typ av analys är arbetsintensiva, typiskt medför insamling av stora mängder av proteinet. De kräver också ofta modifiering av proteinet, begränsar dessa metoders användbarhet för att förstå proteinets beteende i dess naturliga tillstånd.
Penn-forskarnas translokationsteknik gör det möjligt att studera enskilda proteiner utan att modifiera dem. Prover tagna från en enskild individ kan analyseras på detta sätt, öppna ansökningar för sjukdomsdiagnostik och forskning.
Studien leddes av Marija Drndić, en professor vid School of Arts &Sciences Institutionen för fysik och astronomi; David Niedzwiecki, en postdoktor i hennes labb; och Jeffery G. Saven, en professor vid Penn Arts &Sciences Institutionen för kemi.
Det publicerades i tidskriften ACS Nano .
Penn-teamets teknik härrör från Drndićs arbete med nanopore-gensekvensering, som syftar till att särskilja baserna i en DNA-sträng med olika procent av öppningen som de blockerar när de passerar genom en nanoskopisk por. Olika silhuetter tillåter olika mängder av en jonisk vätska att passera igenom. Förändringen i jonflöde mäts av elektronik som omger poren; topparna och dalarna för den signalen kan korreleras till varje bas.
Medan forskare arbetar för att öka noggrannheten i dessa avläsningar till användbara nivåer, Drndić och hennes kollegor har experimenterat med att tillämpa tekniken på andra biologiska molekyler och strukturer i nanoskala.
Samarbetar med Savens grupp, de satte sig för att testa sina porer på ännu knepigare biologiska molekyler.
"Det finns många proteiner som är mycket mindre och svårare att manipulera än en DNA-sträng som vi skulle vilja studera, ", sa Saven. "Vi är intresserade av att lära oss om strukturen hos ett givet protein, till exempel om det existerar som en monomer, eller kombinerat med en annan kopia till en dimer, eller ett aggregat av flera kopior som kallas en oligomer."
Detektion är också ofta en begränsning.
"Det finns inga sätt att amplifiera peptider och proteiner som det finns för DNA, " sade Drndić. "Om du vill studera proteiner från en viss källa, du har fastnat med mycket små prover. Med denna metod, dock, du kan bara samla in mängden data du behöver och antalet proteiner du vill passera genom porerna och sedan studera dem en i taget eftersom de finns naturligt i kroppen."
Genom att använda Drndić-gruppens kiselnitrid-nanoporer, som kan borras till anpassade diametrar, forskargruppen försökte testa sin teknik på GCN4-p1, ett protein valt eftersom det innehåller ett gemensamt strukturellt motiv som finns i transkriptionsfaktorer och intracellulära receptorer.
"Dimerversionen är 'zippad' ihop, " Niedzwiecki sa, "Det är en "lindad spole" av interfolierade spiraler som är ungefär cylindriska. Monomerversionen är uppackad och är troligen inte spiralformad; den är förmodligen mer som en sträng."
Forskarna lade olika proportioner av zippade och uppackade versioner av proteinet i en jonisk vätska och förde dem genom porerna. Även om det inte går att se skillnaden mellan enskilda proteiner, forskarna kunde utföra denna analys på populationer av molekylen.
"Dimer- och monomerformen av proteinet blockerar ett annat antal joner, så vi ser en annan strömminskning när de går genom poren, " sade Niedzwiecki. "Men vi får en rad värden för båda, eftersom inte varje molekylär translokationshändelse är densamma."
Att avgöra om ett specifikt prov av dessa typer av proteiner aggregeras eller inte skulle kunna användas för att bättre förstå sjukdomsförloppet.
"Många forskare, sa Saven, "har observerat dessa långa härvor av aggregerade peptider och proteiner i sjukdomar som Alzheimers och Parkinsons, men det finns allt fler bevis som tyder på att dessa härvor uppstår i efterhand, att det som verkligen orsakar problemet är mindre proteinsammansättningar. Att ta reda på vad dessa sammansättningar är och hur stora de är är för närvarande väldigt svårt att göra, så det här kan vara ett sätt att lösa det problemet."