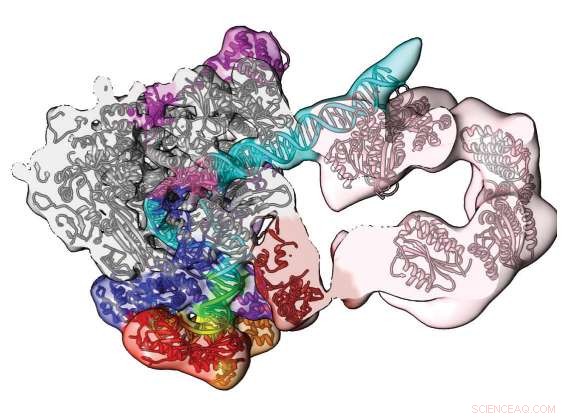
Superdatorer och kryo-elektronmikroskopi avslöjar denna del av det mänskliga pre-initieringskomplexet. Den öppna konformationstäthetskartan och modellen visar DNA -sökvägen (blå/grön) och dess engagemang av transkriptionsfaktorkomponenten TFIIH (rosa). Omtryckt med tillstånd från Macmillan Publishers Ltd:Han, Y. et al. Nära atomupplösning visualisering av mänsklig transkription promotor öppning. Natur 533, 359–365 (2016).
Det låter som något ur Borg i Star Trek. Robotar i nanostorlek monteras själv för att bilda biologiska maskiner som gör det som håller en vid liv. Och ändå pågår något sådant här.
Varje cell i vår kropp - vare sig det är kött och blod, hjärnan och allt däremellan - har identiskt DNA, den vridna trappan av nukleinsyror som är unikt kodad för varje organism. Komplexa sammansättningar som liknar molekylära maskiner tar bitar av DNA som kallas gener och gör en hjärncell vid behov, istället för, säga, en bencell. Dessa molekylära maskiner är så komplexa, ändå så liten, att forskare idag bara börjar förstå sin struktur och funktion med hjälp av de senaste mikroskopen och superdatorer. Biologiska molekylära maskiner kan lägga grunden för att utveckla botemedel mot sjukdomar som cancer. Hur liten kan man se, och vad hittar man?
Kryo-elektronmikroskopi i kombination med superdatorsimuleringar har skapat den bästa modellen än så länge, med detaljer på nära atomnivå, av en vital molekylär maskin, det mänskliga pre-initieringskomplexet (PIC). Ett vetenskapsteam från Northwestern University, Berkeley National Laboratory, Georgia State University, och UC Berkeley publicerade sina resultat på PIC maj 2016 i tidskriften Natur .
"För första gången, strukturer har beskrivits av de komplexa grupperna av molekyler som öppnar upp mänskligt DNA, "säger studieförfattaren Ivaylo Ivanov, docent i kemi vid Georgia State University. Ivanov ledde beräkningsarbetet som modellerade atomerna hos de olika proteinerna som fungerar som kuggar i PIC -molekylmaskinen.
PIC hittar gener som är associerade med att tillverka ett specifikt protein, såsom en antikropp eller ett enzym. Där drar PIC isär de två DNA -strängarna och matar den kodande strängen till arbetshästenzymet RNA -polymeras II. Detta startar transkription, där DNA -bitar kopieras av RNA -polymeras II till en enda sträng av messenger -RNA. RNA tar sig till "proteinfabriker" i cellen som kallas ribosomer som tar dem som order för vilket protein som ska tillverkas. Om DNA är som ritningen för ett nytt hus, RNA är instruktioner till 'entreprenörerna' vid ribosomarbetsstationen. De tillverkade proteinerna är som naglarna, trä, plåster, och nästan allt annat i huset.
Experimentet började med bilder noggrant tagna av PIC. De gjordes av en grupp som leds av studieförfattaren Eva Nogales, en professor vid institutionen för molekylär och cellulär biologi vid UC Berkeley och även senior fakultetsvetare vid Lawrence Berkeley National Laboratory och Howard Hughes Medical Investigator.
Nogales grupp använde kryo-elektronmikroskopi (kryo-EM), en stigande stjärna inom labtekniker. De frös kryogeniskt mänsklig PIC bunden till DNA. Frysningen höll den i en kemiskt aktiv, nära naturlig miljö. Därefter zappade de den med elektronstrålar. Tack vare de senaste framstegen inom direkt elektrondetektorteknik, cryo-EM kan nu avbilda med nära atomupplösning stora och komplicerade biologiska strukturer som har visat sig vara för svåra att kristallisera. Gå-till-tekniken, Röntgenkristallografi, kräver kristalliserade prover, och cryo-EM undviker detta hårda steg.
Över 1,4 miljoner cryo-EM "frysramar" av PIC bearbetades med hjälp av superdatorer vid National Energy Research for Scientific Computing Center för att sålla bort bakgrundsljud och rekonstruerade tredimensionella densitetskartor som visar detaljer i form av molekylen som aldrig hade varit sett förut.
"Cryo-EM genomgår en stor expansion liksom all datorprogramvara som används för att generera både densitetskartor och också för att tolka dem som vi har gjort i den här studien, "Sade Nogales." Det gör att vi kan få högre upplösning av fler strukturer i olika tillstånd så att vi inte bara kan beskriva en bild av hur de ser ut, men flera bilder som visar hur de rör sig. Vi ser ingen kontinuum, men vi ser ögonblicksbilder genom handlingsprocessen. "
Studieforskare byggde sedan en exakt modell som gjorde fysisk känsla för densitetskartorna för PIC med XSEDE, eXtream Science and Engineering Discovery Environment, finansierad av National Science Foundation. XSEDE tillåter forskare att interaktivt dela datorresurser, data och expertis via ett enda virtuellt system. Ivaylo Ivanovs team har kört över fyra miljoner kärntimmar simuleringar på Stampede -superdatorn vid Texas Advanced Computing Center för att modellera komplexa molekylära maskiner, inklusive de för denna studie. Ivanovs bredare molekylära maskinarbete inkluderar också en XSEDE -tilldelning på 1,7 miljoner kärntimmar på Comet -superdatorn vid San Diego Supercomputing Center.
"Jag har använt XSEDE -resurser i mer än 12 år nu, "Sa Ivanov." Utan tillgången på XSEDE -resurser, all vår forskning skulle ha varit mycket mer begränsad när det gäller de system som vi kan ta itu med. För oss, XSEDE har varit absolut nödvändigt. "
Målet med alla dessa beräkningsinsatser är att producera atommodeller som berättar hela historien om strukturen och funktionen av molekylernas proteinkomplex. För att komma dit tog Ivanovs team de tolv komponenterna i PIC-församlingen och skapade homologimodeller för varje komponent som stod för deras aminosyrasekvenser och deras relation till liknande kända protein-3D-strukturer.
Därefter approximerade de den experimentella densiteten Nogales team hittade på ett rutnät. "Vi kan använda en metod som kallas molekylär dynamik flexibel montering, "förklarade Ivanov, "där du i huvudsak kör en molekylär dynamiksimulering. Och du använder den experimentella densiteten för att förspänna atomerna i molekylär dynamiksimulering för att flytta in i de tätare områdena på EM -kartan. Det är processen med flexibel anpassning till EM -kartan."
De förädlade modellen med Phoenix kristallografiska förädlingspaket. "Det är en gratis teknik som gör att vi kan placera sidokedjor och förbättra modellen så att vi kan fånga alla detaljer som finns på densitetskartan, "Sa Ivanov.
XSEDE var "absolut nödvändigt" för denna modellering, sa Ivanov. "När vi inkluderar vatten och motjoner utöver PIC -komplexet i en simuleringsbox för molekylär dynamik, vi får simuleringssystemets storlek på över en miljon atomer. Man kan inte köra det på en arbetsstation eller ens på ett blygsamt kluster. För det behöver vi verkligen gå till tusen kärnor. I detta fall, vi gick upp till två tusen och fyrtioåtta kärnor. Och för det behövde vi tillgång till Stampede, "Sa Ivanov.
En av insikterna i studien är en arbetsmodell för hur PIC öppnar den annars stabila DNA -dubbla helixen för transkription. Nogales förklarade att man kunde tänka sig en sladd gjord av två trådar vridna runt varandra. Håll ena änden mycket hårt. Ta tag i den andra och vrid den i motsatt riktning av gängningen för att lossa sladden. Det är i princip hur de levande maskinerna som håller oss vid liv gör det.
"DNA måste öppnas och flyttas in i det polymeras aktiva stället för att koda för den första RNA -nukleotiden, "förklarade Nogales." Förinitieringskomplexet håller DNA:s två strängar mycket tätt samman i ena änden, så att de inte kan röra sig och de inte kan öppna. På andra sidan av PIC finns en maskin som använder energi för att driva DNA, vrid den i motsatt riktning i vilken de två trådarna är gängade. Och när detta händer, mellan de två sidorna, trådarna öppnas, sa Nogales.
Denna studie löste strukturen för den molekylära maskinen som fungerar som de vridande fingrarna, transkriptionsfaktorkomponenten TFIIH. "TFIIH har en translokasundenhet, vars roll är att samtidigt skjuta DNA mot det aktiva stället för polymeraset och varva ner DNA:t. Genom det kombinerade trycket och avkopplingen, effektivt separerar du de två delarna av DNA:t, "Sa Ivanov.
Båda forskarna sa att de precis har börjat få en atomär förståelse för transkription, avgörande för genuttryck och i slutändan sjukdom. "Många sjukdomstillstånd uppstår eftersom det finns fel i hur mycket en viss gen läses och hur mycket ett visst protein med en viss aktivitet i cellen är närvarande, "Sade Nogales." Dessa sjukdomstillstånd kan bero på överproduktion av proteinet, eller omvänt inte tillräckligt. Det är mycket viktigt att förstå den molekylära processen som reglerar denna produktion så att vi kan förstå sjukdomstillståndet. "
"Detta arbete illustrerar väl två allmänna principer som kommer att driva vetenskapen under de närmaste åren, "kommenterade Peter Preusch, programansvarig med National Institutes of Health (NIH). "Den ena är tillämpningen av hybridmetoder-kombinationer av biofysiska metoder inklusive röntgenkristallografi och cryoEM tillsammans med beräkningsmetoder i stor skala för att integrera information om större molekylkomplex. Två, Det finns krav på att teamvetenskap ska dra expertis från flera utredare för att lösa problem som inte kan hanteras av ett enda labb som arbetar ensam. "Peter Preusch är chef för biofysik, Avdelningen för cellbiologi och biofysik, National Institute of General Medical Sciences, NIH.
Även om detta grundläggande arbete inte direkt producerar botemedel, det lägger grunden för att hjälpa till att utveckla dem i framtiden, sa Ivanov. "För att förstå sjukdom, vi måste förstå hur dessa komplex fungerar i första hand ... Ett samarbete mellan beräkningsmodeller och experimentella strukturbiologer kan vara mycket fruktbart i framtiden. "
Studien i maj 2016 Nature Articles (DOI:10.1038/nature17970), "Visualisering av nära atomupplösning av öppning av mänsklig transkriptionspromotor, "författades av Yuan He, Lawrence Berkeley National Laboratory och nu vid Northwestern University; Chunli Yan och Ivaylo Ivanov, Georgia State University; Jie Fang, Carla Inouye, Robert Tjian, Eva Nogales, UC Berkeley. Finansiering kom från National Institute of General Medical Sciences (NIH) och National Science Foundation.