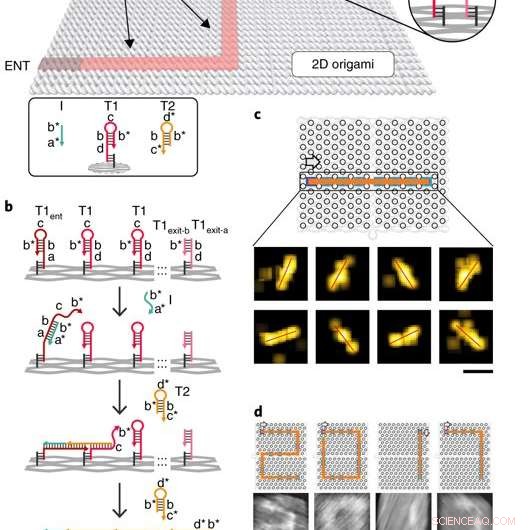
Implementering av DNA-navigatorn med en molekyl. a) Schematisk illustration av PSEC-systemet. En acyklisk sammankopplad graf (dvs ett träd) är konstruerad på ett rektangulärt DNA-origamisubstrat på 100 x 70 nm^2. T1 och T2 är två typer av bränslen som driver PSEC på trädet. Initiator I användes för att utlösa initieringen av PSEC från ingångspunkten ENT. b) spridningsmekanism för PSEC-systemet. Initiator I känner igen och öppnar T1ent-hårnålen vid vertex ÖNH. Den öppnade hårnålen fångar sedan och öppnar en T2 hårnål från omgivningen för att starta kaskaden. c) En rak linje banad av PSEC och visualiserad med DNA-PAINT. d) Fyra asfalterade siffror av "2017" avbildade med atomkraftsmikroskopi (AFM). T1-hårnålarna vid hörnpunkterna modifierades för att förhindra oönskade spann. Pilarna anger utbredningsriktningen. Kreditera: Naturmaterial , doi:10.1038/s41563-018-0205-3.
Området intelligent nanorobotik är baserat på det stora löftet om molekylära enheter med informationsbehandlingskapacitet. I en ny studie som stöder trenden med DNA-baserade informationsbärare, forskare har konstruerat ett DNA-navigatorsystem som kan utföra en molekyl, parallell, djupgående sökoperationer på en tvådimensionell origamiplattform.
Pathfinding-operationer med DNA-navigatorer använder sig av en lokaliserad strängbyteskaskadprocess som initieras vid en unik triggerplats på origamiplattformen. Automatisk progression längs banor möjliggörs av DNA-hårnålar som innehåller en universell genomgångssekvens. Genom design, varje enmolekylär navigator kan självständigt utforska vilken som helst av de möjliga vägarna genom ett rotat träd med 10 vertex som konstruerats i studien. Labyrinterna motsvarade ett träd med en ingång vid roten och en utgång genom ett av löven. Studien utförd av Jie Chao och medarbetare resulterade i att utforska alla vägar som DNA-navigatorerna tog för att extrahera en specifik lösningsväg som kopplade samman ett givet par av start- och slutpunkt i labyrinten. Som ett resultat, Lösningsbanan lades enkelt på origamiplattformen och illustrerades med en molekylavbildning. Tillvägagångssättet är nu publicerat i Naturmaterial , detaljer om realiseringen av molekylära material med inbäddade biomolekylära beräkningsfunktioner för att fungera på nivån av en enda molekyl med potential att konstruera intelligenta nanorobotar för framtida tillämpningar inom industri och medicin.
Sofistikerade molekylära verktyg användes tidigare för att skapa molekylära maskiner som omvandlar kemikalier, fotonisk eller elektrisk energi till roterande eller linjära rörelser på nanoskala. Till exempel, Brownsk rörelse på nanoskala kan kontrolleras omvandlas till riktade rörelser inom DNA-baserade nanomaskiner med hjälp av DNA-hybridiseringsreaktioner. Sådana DNA-baserade maskiner fungerar autonomt genom att följa ett inbäddat "molekylärt program" fördesignat som en kaskadreaktion manuellt utlöst via en extern stimulans för varje steg i operationen.
Fokus för området har successivt skiftat till att aktualisera DNA-baserade logiska kretsar med användning av aptamerer och DNAzymer för att designa molekylära logiska grindar. Till exempel, under 2006, Stojanovic och hans medarbetare integrerade mer än 100 DNA-logikportar för att konstruera en automatisering som heter MAYA-II för att spela en omgång Tic-Tac-Toe. Tidigare studier har visat ett enzymfritt datorsystem baserat på hybridiseringskedjereaktioner (HCR) för att skapa logiska grindar och logiska kretsar för mer robust och effektiv prestanda än de ursprungliga systemen.
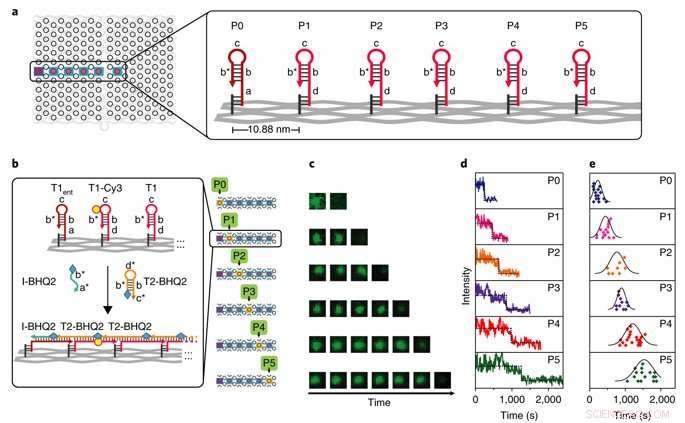
Enkelmolekylkarakterisering av PSEC-kinetik. a) En rak linje i mitten av DNA-origami användes som testbädd. Vertex P0 är startpunkten och hörn P1-P5 är mellanliggande punkter. b) Detaljer om utformningen av sex parallella tester för att mäta kinetiken med tidsupplöst TIRF. T2 märkt med BHQ2 användes för att släcka T1 märkt med Cy3, montera en kinetisk profil vid varje steg. Det illustrerade exemplet visar släckningsmekanismen. c) Exempel på TIRF-bilder visar att fluorescensen förändras med tiden över de sex parallella testerna från P0 till P5. d) Typiska enkelmolekylära fluorescensspår som används för att övervaka släckningshändelser som inträffar vid hörn P0 till P5. e) Spridningsdiagram som visar statistisk analys av längdfördelningen per väg. Kreditera: Naturmaterial , doi:10.1038/s41563-018-0205-3.
I den aktuella studien av Chao et al, samma grundläggande princip för HCR-reaktionsschemat användes i ett annat beräkningssammanhang för att utveckla ett DNA-navigatorsystem med en molekyl. Plattformen utforskade alla möjliga vägar genom en trädgraf designad på en origamistruktur som en enkelt sammankopplad labyrint utan cykliska stigar. Sådana DNA-origamistrukturer är informationsbärande nanostrukturer av naturen med väldefinierad nanoskala geometri. Labyrinten kunde utforskas genom proximal strängbyteskaskad (PSEC) baserad på hybridiseringskedjereaktioner. Forskarna visade att ett system med ett stort antal enkelmolekylära DNA-navigatorer tillsammans kunde utföra parallell djup-först-sökning (PDFS) på trädet för att effektivt utföra labyrintlösning inom 2-D origami. Inledningsvis genomförde forskarna studier för att testa PSEC-designen.
Systemet för proximal strängbyteskaskad (PSEC) (arbetsprincipen för DNA-navigatorn) underlättades på ett rektangulärt origamisubstrat gjord av tre komponenter, som inkluderade den fysiska implementeringen av en trädgraf, fulla strängar och en initiatorsträng. Lediga ytor utan häftförlängningar motsvarade väggar i labyrinten, förhindrar utbredning av strängbyteskaskaden. The entrance and exit were defined and denoted as ENT and EXIT respectively. In the second component, two types of DNA hairpins, T1 and T2, were used as fuels to drive the PSEC on the tree graph. The two hairpins coexisted metastably in solution to hybridize and fuel the PSEC process with free energy .
Genom design, information only propagated through the network in the presence of an initiator (Initiator I). Upon addition of initiator I, PSEC was conducted and observed using atomic force microscopy (AFM). To visualize an established formation, the scientists enabled DNA-navigator-based formation of the number 2017 as a proof-of-principle. Another technique known as DNA-PAINT was employed as a single-molecule, super-resolution imaging technique to reveal molecular features at the nanoscale to further substantiate the PSEC-based path paving process. The on-origami PSEC was highly specific, without intra- or inter-origami crosstalk.
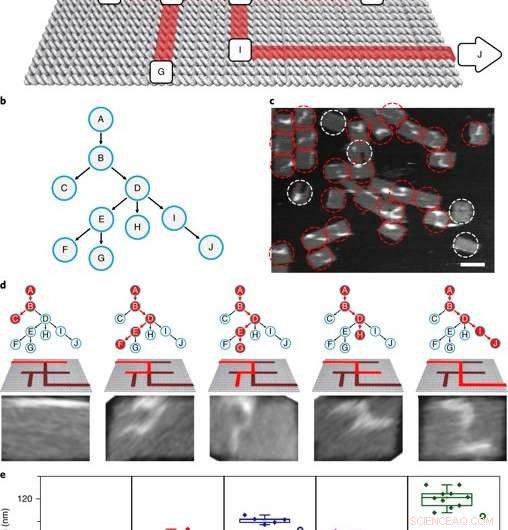
The PSEC-driven graph traversal on a maze. a) A maze design with 10 vertices. Arrows indicate the entrance vertex A and exit vertex J. b) The maze is equivalent to a rooted tree with 10 vertices. The entrance vertex A corresponds to the root of the tree. c) An AFM image showed the result of a transversal experiment generating all possible paths. In this DNA computing implementation of a PDFS algorithm, a vast number of PSEC events simultaneously occurred to realize the graph traversal on the maze. PSEC ending at the exit or deadends were highlighted in red circles. Invalid structures were highlighted with white circles. d) Typical paths found in the mixture seen from left to right. Only P ABDIJ was the correct solution to the maze. e) Scatter plots showing the statistical analysis of the length distribution for each path. Kreditera: Naturmaterial , doi:10.1038/s41563-018-0205-3.
Kinetics of the process were investigated at the single-molecule level in the study using time-resolved total internal reflection fluorescence microscopy (TIRF) in a setup with a prescribed starting point (P0) and five intermediate steps (P1-P5). Fluorescence in the setup was quenched using fluorescence resonance energy transfer (FRET), and the cascade was observed in real time by recording fluorescence signals continuously. The average speed of propagation was recorded to be 2.46 nm per minute, propagation across the straight line (54.4 nm) took approximately 22 minutes.
The scientists then constructed the main model maze with 10 vertices that included an entrance vertex A and an exit vertex J, three junctions (B, D, E) an intermediate vertex (I) and four dead ends (C, F, G, H). Each path of the maze that was equivalent to a tree with 10 vertices was investigated using DNA investigators starting at root A. The PSEC reactions produced a mixture of various paths on the maze, confirmed with AFM. Each individual PSEC could progress on one of the five possible paths. Statistical analysis of the length distribution showed that the measured paths coincided well with the values predicted.
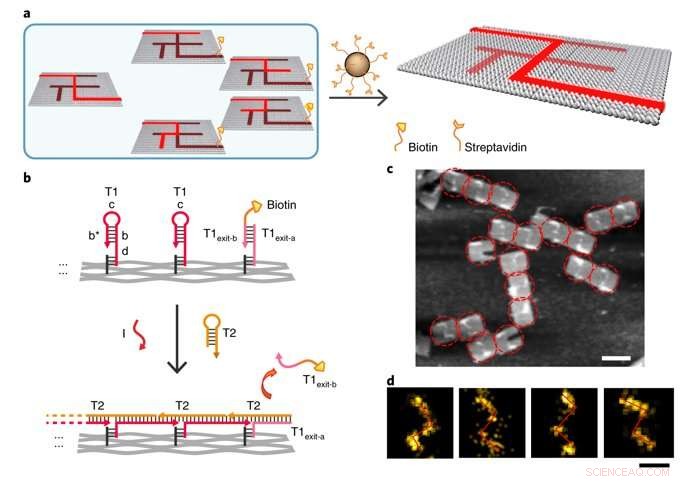
Single-molecule DNA navigators for maze-solving. a) Schematic illustration of magnetic bead-based selection. Exit vertex J is labelled with biotin to enable differentiation between the correct and wrong paths. Only if the PSEC reaches the correct exit J, would the biotin-modified T1exit-B strand be released. All wrong paths could be captured and removed by the Streptavidin-modified magmatic beads therefore. b) Details of the release of biotin modification at the exit J by the PSEC. c) AFM characterization of the correct solution after selection. The remaining structures all showed the correct solution path PABDIJ. d) Single-molecule and class-averaged DNA-PAINT characterization of the correct solution after selection. Kreditera: Naturmaterial , doi:10.1038/s41563-018-0205-3.
To prevent the navigators from propagating through a wrong path with dead ends, the scientists designed a streptavidin-biotin tag-based method to selectively eliminate inaccurate path navigation. Only the correct path (P ABDIJ ) was followed in the maze therefore. The computational context used in the study allowed exploration of paths through tree graphs defined on the origami. The autonomous path explored by the DNA navigators proceeded unidirectionally and irreversibly, turning at junctions and corners on the origami platform as they were designed to. The design enabled parallel depth first search (PDFS) allowing each DNA navigator to individually explore any one of the paths through the given graph at a defined speed, greater than that previously achieved.
The main advantage of the described biomolecular computer schemes in comparison to conventional electronic computing is they can be interfaced directly with biologically relevant processes. Som ett resultat, the scientists envision translational biomedical sensing and decision-making platforms with DNA origami and single-molecule diagnostics using decision trees. Future applications will also include simple sensors or those coupled to a molecular actuator to trigger downstream molecular cascades.
© 2018 Science X Network