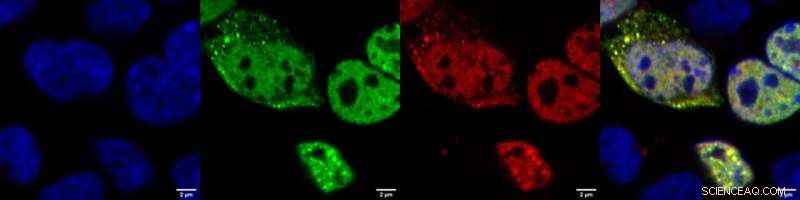
Zooma in på enstaka odlade däggdjursceller i vilka TDP-43 har inducerats att aggregera. I detta system producerar cellerna TDP-43 smält till en grön fluorescerande molekyl, för att kunna detektera om proteinet bildar olösliga granuler (gröna fluorescerande prickar). RNA-sonden är märkt med en röd fluorescerande tagg. Den gula färgen, som ges av överlappningen mellan det gröna i TDP-43 och det röda i RNA-sonden, betyder att sonden kan söka och hitta sitt proteinmål i levande celler, vilket tyder på att den kan användas som ett detektionsverktyg för att spåra utvecklingen av TDP-43-aggregation vid sjukdom.Blå:kärnor; grön:TDP-43; röd:RNA-sond; gul:TDP-43+RNA-sond. Kredit:IIT-Istituto Italiano di Tecnologia
Ett team av forskare från IIT-Istituto Italiano di Tecnologia (Italienska tekniska institutet) har i silico designat "molekylära prober" som kan spåra utvecklingen av ett protein som inte beter sig i olika neurodegenerativa sjukdomar, såsom amyotrofisk lateralskleros (ALS) och Fronto-Temporal Demens (FTD). Proberna kan användas för att studera beteendet hos målproteinet i en cell och testades i samarbete med Sapienza University of Rome, Center for Genomic Regulation n Barcelona, University of Edinburgh och Kings College London. Forskningsstudien har publicerats i Nature Communications.
Skapat av gruppen "RNA Systems Biology" vid IIT i Genua, proberna består av datordesignade RNA-molekyler som binder till ett neurodegenerationsassocierat protein som heter TDP-43. Detta protein finns i många fall av amyotrofisk lateralskleros (ALS) och fronto-temporal demens (FTD), där det aggregeras och skapar olösliga proteinblobbar i neurala celler, vilket förändrar deras metabolism och funktion.
Forskargruppen inspirerades av proteinets naturliga interaktioner med RNA-molekyler för att designa molekylära prober, som kallas "aptamerer", bokstavligen molekyler gjorda för att passa ett enda mål. Deras huvudsakliga mål var att få fram ett nytt tillvägagångssätt för att spåra aggregationen av neurodegenerationsassocierade proteiner i de allra första stegen av processen.
"Med våra egna algoritmer designade vi RNA-aptamerer specifika för TDP-43 och använde dem tillsammans med avancerade mikroskopitekniker för att följa proteinövergången mot dess aggregerade former", förklarar Gian Gaetano Tartaglia, huvudutredare för RNA System Biology Lab. "Vi kan identifiera TDP-43-aggregat så små som 10 nanometer, vilket, såvitt vi vet, är den bästa upplösningen som uppnåtts hittills när vi visualiserar proteinaggregat."
Zooma in på enstaka odlade däggdjursceller i vilka TDP-43 har inducerats att aggregera. I detta system producerar cellerna TDP-43 smält till en grön fluorescerande molekyl, för att kunna detektera om proteinet bildar olösliga granuler (grönt fluorescerande) prickar). RNA-sonden är märkt med en röd fluorescerande tagg. Den gula färgen, som ges av överlappningen mellan det gröna i TDP-43 och det röda i RNA-sonden, betyder att sonden kan söka och hitta sitt proteinmål i levande celler, vilket tyder på att den kan användas som ett detektionsverktyg för att spåra utvecklingen av TDP-43-aggregation vid sjukdom. Blå:kärnor; grön:TDP-43; röd:RNA-sond; gul:TDP-43+RNA-sond. Kredit:IIT-Istituto Italiano di Tecnologia
Dessa aptamerer skulle kunna användas för att studera, på molekylär nivå, fenomenet med onormal proteinaggregation som är typiskt för flera neurodegenerativa sjukdomar och skulle därför bana väg för utvecklingen av tidig diagnosverktyg för dessa sjukdomar.
"Vi visade att RNA-aptamererna också kan användas för att spåra TDP-43 i levande celler och i realtid, för att detektera alla former av proteinet, från den fysiologiskt lösliga till det olösliga tillståndet, passera genom aggregat av mellanstorlekar som inte går att upptäcka enligt standard. tillvägagångssätt", tillägger Elsa Zacco, ledande forskare i projektet. + Utforska vidare