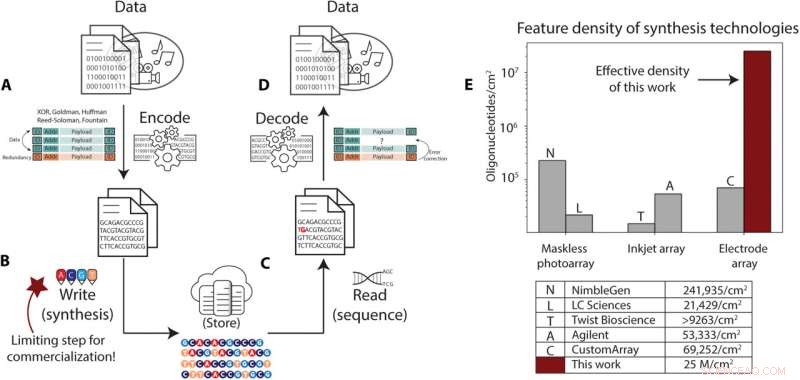
DNA-datalagring kräver högre syntesgenomströmning än vad som är möjligt med nuvarande tekniker. (A till D) Översikt av pipeline för DNA-datalagring. (A) Digitala data kodas från sin binära representation till sekvenser av DNA-baser, med en identifierare som korrelerar dem med ett dataobjekt, adresseringsinformation som används för att ordna om data vid läsning och redundant information som används för felkorrigering. (B) Dessa sekvenser syntetiseras till DNA-oligonukleotider och lagras. (C) Vid hämtningstidpunkten väljs DNA-molekylerna ut och kopieras via PCR eller andra metoder och sekvenseras tillbaka till elektroniska representationer av baserna i dessa sekvenser. (D) Avkodningsprocessen tar denna bullriga och ibland ofullständiga uppsättning av sekvensläsningar, korrigerar för fel och saknade sekvenser och avkodar informationen för att återställa data. (E) Sammanfattning av de kommersiella syntesprocesserna och motsvarande uppskattade oligonukleotiddensiteter, som rapporterats i litteraturen eller av företagen själva. Vår elektrokemiska metoddensitet är markerad i mörkrött. Kredit:Science Advances , 10.1126/sciadv.abi6714
Genetiker kan lagra data i syntetiskt DNA som ett medium för långtidslagring på grund av dess densitet, lätthet att kopiera, livslängd och hållbarhet. Forskningen inom området hade nyligen gått framåt med nya kodningsalgoritmer, automatisering, bevarande och sekvensering. Ändå är det mest utmanande hindret vid implementering av DNA-lagring skrivkapaciteten, vilket kan begränsa datalagringskapaciteten. I en ny rapport utvecklade Bichlien H. Nguyen och ett team av forskare inom Microsoft Research och datavetenskap och ingenjörskonst vid University of Washington, Seattle, USA, den första DNA-lagringsskrivaren i nanoskala. Teamet hade för avsikt att skala DNA-skrivtätheten till 25 x 10 6 sekvenser per kvadratcentimeter, en förbättrad lagringskapacitet jämfört med befintliga DNA-syntesmatriser. Forskarna skrev och avkodade framgångsrikt ett meddelande i DNA för att etablera ett praktiskt system för DNA-datalagring. Resultaten publiceras nu i Science Advances .
Långsiktiga DNA-arkiv
Den nuvarande takten för datagenerering överstiger befintlig lagringskapacitet, DNA är en lovande lösning på detta problem med en förväntad praktisk densitet på mer än 60 petabyte per kubikcentimeter. Materialet är hållbart under en rad förhållanden, relevant och lätt att kopiera, med löfte om att vara mer hållbart eller grönare än kommersiella medier. Under processen kan digital data i form av sekvenser av bitar kodas i sekvenser av de fyra naturliga DNA-baserna - guanin, adenin, tiamin och cytosin, även om ytterligare baser också är möjliga. Teamet kan sedan skriva sekvenserna till molekylär form via de novo DNA-oligonukleotidsyntes för att skapa specifika molekyler baserade på en uppsättning upprepade kemiska steg. De resulterande oligonukleotiderna kan bevaras och lagras efter syntes. För att komma åt data kan DNA-lagringen amplifieras med användning av polymeraskedjereaktioner och sekvenseras för att återföra DNA-bassekvenserna till den digitala domänen, sedan kan DNA-bassekvenserna avkodas för att återställa den ursprungliga sekvensen av bitar.
Översikt över 650-nm array med en pitch på 2 μm. (A) Finita elementanalys av anodsyragenerering och diffusion vid en 650 nm-diameter elektrod med en 200-nm brunn visas med en tvärsnittsvy längs y =x-planet och (B) uppifrån och ned på z =0 plan. Färgerna blått och gult representerar regioner med relativt låga respektive höga syrakoncentrationer. (C) En översikt över DNA-syntesen i nanoskala med svepelektronmikroskopbilder av 650-nm elektroduppsättningen och förstorad bild av en elektrod. (D) En fluorescerande bild där brunnen som omger varje aktiverad anod är mönstrad med AAA-fluorescein. Det tecknade diagrammet visar vilka elektroder i layouten som aktiverades. (E) Illustration av brunnarna mönstrade med AAA-fluorescein och AAA-AquaPhluor och (F) motsvarande bildöverlagring av de två fluoroforerna i änden av DNA syntetiserat på samma 650-nm elektroduppsättning. Kredit:Science Advances , 10.1126/sciadv.abi6714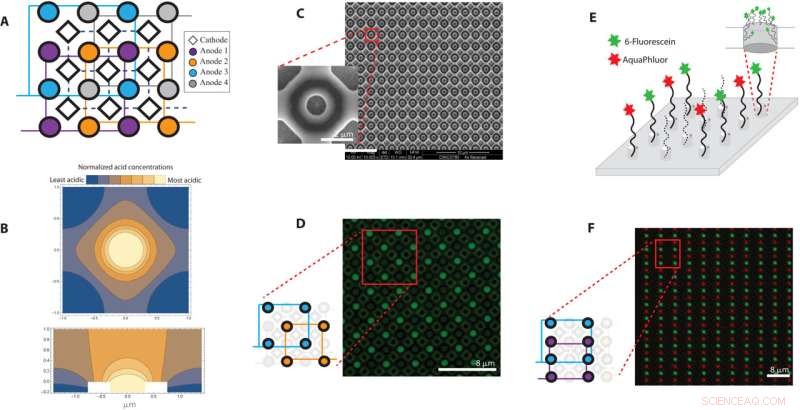
I denna studie har Nguyen et al. producerade en elektroduppsättning som visade oberoende elektrodspecifik kontroll av DNA-syntes med elektrodstorlekar och stigningar för att etablera syntestäthet på 25 miljoner oligonukleotider per cm 2 . Detta värde uppskattas som den elektrodtäthet som krävs för att uppnå minimimålet på kilobyte per sekund av datalagring i DNA. Teamet drev det senaste inom elektronisk-kemisk kontroll och gav experimentella bevis för den skrivbandbredd som krävs för lagring av DNA-data.
Teamet introducerade en proof-of-concept molekylär styrenhet i form av en liten DNA-lagringsskrivmekanism på ett chip. Chipet kunde tätt packa DNA-syntes med 3 storleksordningar högre än tidigare för att uppnå större DNA-skrivkapacitet. För att lagra information i DNA i den skala som krävs för kommersiellt bruk krävdes två avgörande processer. Först fick teamet översätta digitala bitar (ettor och nollor) till strängar av syntetiskt DNA som representerade bitar med kodningsprogram och en DNA-syntes. Sedan måste de kunna läsa och avkoda informationen tillbaka till sina bitar för att återställa den informationen till digital form igen med en DNA-sekvenserare och avkodningsprogram.
Utvecklar elektrokemiska arrayer för funktioner i nanoskala
Under den traditionella syntesen av DNA-kedjor använder forskare en flerstegsmetod känd som fosforamiditkemi, där en DNA-kedja kan odlas sekventiellt genom tillägg av DNA-baser. Varje DNA-bas innehåller en blockerande grupp för att förhindra flera tillägg av DNA-baser till den växande kedjan. Vid anslutning till en DNA-kedja kan syra levereras i uppställningen för att klyva den blockerande gruppen och prima DNA-kedjan för att lägga till nästa bas. Under elektrokemisk DNA-syntes innehåller varje punkt i arrayen en elektrod och när en spänning appliceras genereras syra vid arbetselektroden (anod) för att deblockera de växande DNA-kedjorna, medan en ekvivalent bas genereras vid motelektroden (katoden) . Teamet förhindrade syradiffusion i uppställningen genom att designa en elektroduppsättning, där varje arbetselektrod runt vilken syrabildning inträffade under DNA-syntes sänktes i en brunn och omgavs av fyra vanliga motelektroder, dvs katoder som drev basbildning, för att begränsa syran till specifika regioner. Nguyen et al. verifierade effektiviteten av designen med hjälp av finita elementanalys. Under experimenten, när den presenterades i tillräcklig koncentration, avblockerade syran de ytbundna nukleotiderna för att tillåta nästa nukleotid att kopplas. Med hjälp av uppsättningen av chips som innehåller funktionsfläckar för att begränsa syror, utvecklade de elektrokemiska arrayer med fyra individuella elektroder för att reglera DNA-syntes. Teamet utförde sedan experiment med två fluorescerande märkta baser i grönt och rött. Som bevis på konceptet visade de enhetens kapacitet att skriva data genom att syntetisera fyra unika DNA-strängar, var och en 100 baser långa med ett kodat meddelande, utan fel.
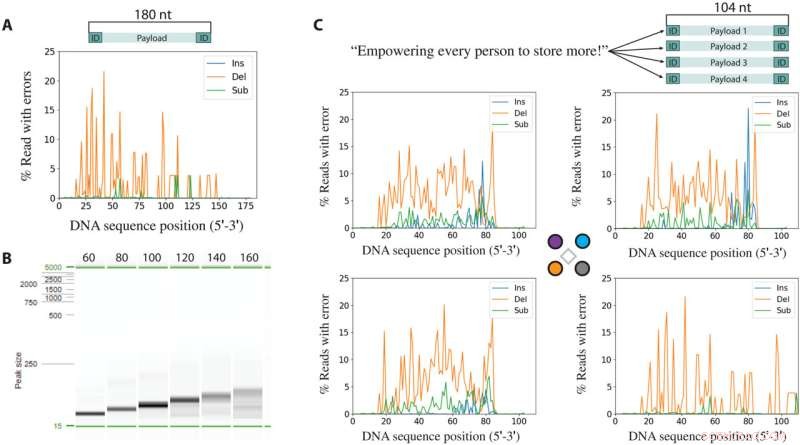
Fel som härrör från syntes följt av sekvensering. (A) Insertioner (Ins), deletioner (Del) och substitutioner (Sub) per position för en syntetiserad och PCR-amplifierad 180-bassekvens. (B) Elektroforesbild av syntesprodukter efter PCR-amplifiering. (C) Meddelande kodat i 64 byte uppdelat i fyra unika sekvenser om 104 baser (överst). Insertioner, deletioner och substitutioner per lokus av var och en av de fyra sekvenserna i multiplexsynteskörningen. I varje felanalysgraf kommer de terminala 20 baserna vid både 3'- och 5'-ändarna från primrarna som används i PCR och är inte representativa för de syntetiserade felen. Kredit:Science Advances , 10.1126/sciadv.abi6714
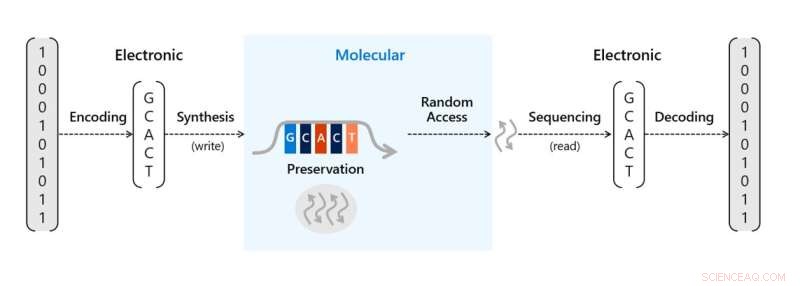
Skalning av DNA-datalagring med elektrodbrunnar i nanoskala. Liten DNA-lagringsskrivmekanism på ett chip. Kredit:Microsoft Research Blog, Science Advances , 10.1126/sciadv.abi6714
Outlook:Syntetisera korta oligonukleotider på elektroduppsättningen för datalagring
Med hjälp av inställningen, Nguyen et al. visade också rumsligt kontrollerad syntes av korta oligonukleotider på elektroduppsättningen för att bedöma den maximala längden av DNA som kunde bildas. Forskarna skapade en enda DNA-sekvens med 180 nukleotider och PCR-amplifierade produkter av olika längd från hela oligonukleotidernas längd. När amplikonen blev längre verkade de förväntade PCR-produkterna svagare och mindre väldefinierade, medan kortare amplikoner visade starkare och mer väldefinierade band som tyder på högre syntesfel. Baserat på resultaten valde forskarna en sekvenslängd som motsvarar 100 baser för att underlätta reningen för att ge en praktisk demonstration av lagring av DNA-data utan ytterligare optimering. På detta sätt banade proof-of-concept-metoden som demonstreras i detta arbete av Bichlien H. Nguyen och kollegor vägen framåt för att generera storskaliga och unika DNA-sekvenser parallellt för datalagring. Arbetet överträffade tidigare rapporter om täta syntetiska DNA-sekvenser för att ge en första experimentell indikation för att uppnå den skrivbandbredd som krävs för datalagring i nanoskala funktionsstorlekar. Forskarna förväntar sig omedelbara tillämpningar av enheterna inom informationsteknologi och förutser deras praktiska tillämpningar inom materialvetenskap, syntetisk biologi och storskaliga molekylärbiologiska analyser. + Utforska vidare
© 2021 Science X Network