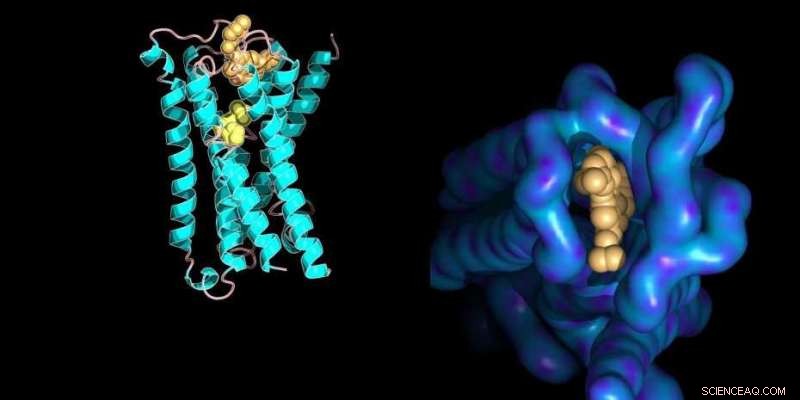
Molekylär modell av en proteinmembranreceptor bunden till dess små molekylligander med strukturella detaljer om det allosteriska bindningsstället (höger). Kredit:Ilaria Piazza / ETH Zürich / PDB databaspost 4MQT
Forskare från ETH Zürich har använt ett nytt tillvägagångssätt för att upptäcka tidigare okända interaktioner mellan proteiner och små metaboliska molekyler i bakterieceller. Tekniken kan också användas för att testa effekten av mediciner.
Vetenskapen har nu en ny "omics" - en interaktomik, som handlar om interaktioner mellan proteiner och små molekyler. Efter att tidigare ha fokuserat på genomik eller proteomik, systembiologer kan nu lägga till protein-metabolit interaktomik till mixen. Genomics handlar om systematisk analys av alla en organisms gener, medan proteomik handlar om hela uppsättningen av proteiner inom en biologisk enhet.
Med protein-metabolit interaktomik, gruppen ledd av Paola Picotto, professor i Molekylär systembiologi vid ETH Zürich, har nu lagt till ytterligare en nivå till den befintliga "omics". Forskarna publicerade nyligen en studie i tidskriften Cell, där de systematiskt analyserade och kvantifierade interaktionerna mellan alla proteiner och metaboliter (små metabola molekyler) på nivån av hela proteomet för första gången, etablera relationerna dem emellan.
Olika klippplatser
Forskarna har visat hur många av de proteiner och enzymer som finns i en E. coli-bakteriecell interagerar med metaboliter. Att göra så, de använde en metod som kallas begränsad proteolys (LiP), kombinerat med masspektrometrisk analys.
Forskarna extraherade cellvätska, som innehåller proteiner, från bakteriecellerna. De tillsatte sedan en metabolit till varje prov och lät den interagera med proteinerna. Till sist, de skär proteinerna i mindre bitar (peptider) med hjälp av "molekylära saxar". Totalt, forskarna testade 20 olika metaboliter och deras interaktioner med proteiner på detta sätt.
När ett protein interagerar med en metabolit, om det sätter sig på proteinets aktiva plats eller fäster till en annan plats, proteinstrukturen förändras. Den "molekylära saxen" klippte den sedan på olika platser från den ursprungliga strukturen, vilket resulterar i en annan uppsättning peptider.
Med hjälp av masspektrometern, forskarna mätte alla delar som fanns i provet och matade in data som erhölls till en dator för att rekonstruera de strukturella skillnaderna och förändringarna, och var i proteinet dessa är lokaliserade.
Hundratals nya interaktioner upptäckts
Kunskap om protein-metabolit-interaktomen (interaktionerna mellan proteiner och metaboliter och de molekylära (signal-) nätverken som tillhör dem), var tidigare mycket begränsad jämfört med vad man visste om interaktioner mellan olika proteiner eller mellan proteiner och DNA eller RNA. Studien har nu kraftigt ökat denna kunskap.
Genom att använda detta tillvägagångssätt med E. coli, Picotto och hennes team upptäckte ungefär 1, 650 olika protein-metabolitinteraktioner, varav över 1, 400 var tidigare okända. Tusentals bindningsställen på proteiner som metaboliter kan fästa vid kom också fram. "Även om metabolismen av E. coli och associerade molekyler redan är mycket välkänd, vi lyckades upptäckta många nya interaktioner och motsvarande bindningsställen, " säger Picotti. Detta bevisar metodens enorma potential:"Datan som vi producerar med den här tekniken kommer att hjälpa till att identifiera nya regleringsmekanismer, okända enzymer och nya metabola reaktioner i cellen."
Strukturförändringar reglerar aktiviteten
I deras studie, forskarna visar också att små metaboliska molekyler föredrar att binda till (och därmed reglera) proteiner vars koncentration är mer eller mindre konstant över tiden. Detta tyder på att bindningen av metaboliter till proteiner och förändringar i proteinkoncentrationer är två komplementära vägar genom vilka celler reglerar proteinaktivitet.
Proteiner kan aktiveras eller inaktiveras relativt snabbt genom en strukturell förändring som medieras av metaboliter. "Denna typ av strukturförändring kan vändas snabbare, " förklarar Picotto. Ur cellens perspektiv, detta är ofta vettigt, eftersom att använda vägen som involverar en förändring i koncentrationen innebär att cellen måste demontera eller återuppbygga proteiner, vilket kostar mer tid, energi och resurser.
Picotti och hennes kollegor har också kunnat visa att många enzymer är mindre kräsna än man tidigare trott:de kan tydligen binda till och kemiskt förändra flera olika metaboliter. Tills nu, det antogs att enzymer mestadels var specifika för ett fåtal mycket lika molekyler.
Läkemedelsindustrin är mycket intresserad av det nya tillvägagångssättet, eftersom det kan användas för att testa interaktionen mellan läkemedel och cellulära proteiner och identifiera målen för ett läkemedel. Forskare kan undersöka vilka proteiner och vilka platser detta läkemedel binder till, hur det förändrar deras struktur och därmed påverkar deras aktivitet. Detta skulle underlätta och påskynda tester och utveckling av nya läkemedel.