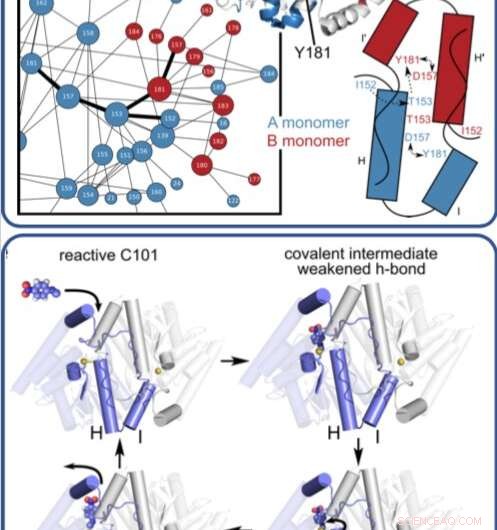
Denna illustration visar hur ett enzym rör sig och förändras när det katalyserar komplexa reaktioner och bryter ner organiska föreningar. Kreditera: Förfaranden från National Academy of Sciences
I ett tidsupplöst röntgenförsök, forskare avslöjade, i atomupplösning och i realtid, det tidigare okända sättet att ett mikrobiellt enzym bryter ner organiska föreningar.
Laget, ledd av Mark Wilson vid University of Nebraska Lincoln (UNL) och Henry van den Bedem vid Department of Energy's SLAC National Accelerator Laboratory (nu vid Atomwise Inc.), publicerade sina fynd förra veckan i Förfaranden från National Academy of Sciences . Vad de lärde sig om detta enzym, vars struktur liknar en som är inblandad i neurodegenerativa sjukdomar som Parkinsons, kan leda till en bättre förståelse för hur antibiotika bryts ner av mikrober och till utveckling av effektivare läkemedel.
Tidigare, forskarna använde SLACs Stanford Synchrotron Radiation Lightsource (SSRL) för att erhålla enzymets struktur vid mycket låga temperaturer med hjälp av röntgenkristallografi. I den här studien, Medhanjali Dasgupta, en UNL -doktorand som var studiens första författare, använde Linac Coherent Light Source (LCLS), SLAC:s röntgenlaser, att se enzymet och dess substrat i kristallen röra sig och förändras när det gick igenom en fullständig katalytisk cykel vid rumstemperatur.
Forskarna använde speciell programvara, designad av van den Bedem, som är mycket känslig för att identifiera proteinrörelse från röntgenkristallografidata för att tolka resultaten, avslöjar aldrig tidigare setta rörelser som spelar en nyckelroll för att katalysera komplexa reaktioner, som att bryta ner antibiotika. Nästa, forskarna hoppas kunna använda LCLS för att erhålla rumstemperaturstrukturer av andra enzymer för att få en bättre titt på hur de rörelser som sker inom dem hjälper till att gå längs reaktioner.