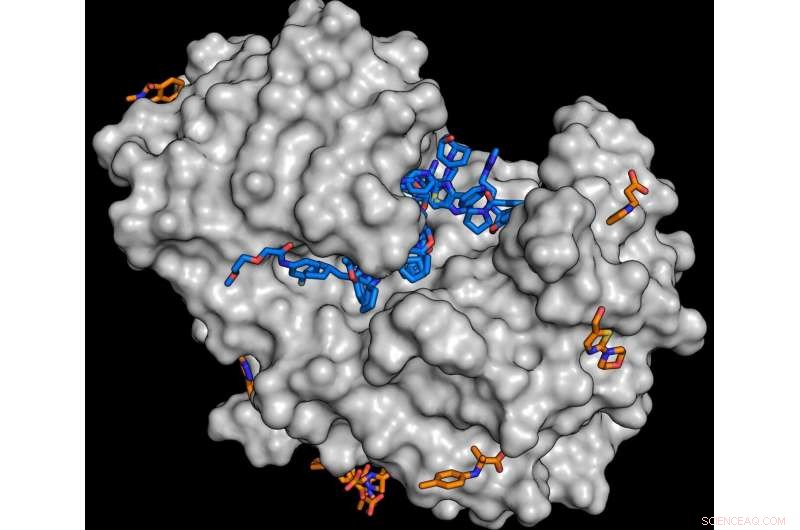
För studien, enzymet endotiapepsin (grått) kombinerades med molekyler från fragmentbiblioteket. Analysen visar att många ämnen kan docka till enzymet (blå och orange molekyler). Varje hittat ämne är en potentiell utgångspunkt för utvecklingen av större molekyler. Upphovsman:J. Wollenhaupt/HZB
För att påskynda den systematiska utvecklingen av läkemedel, MX-teamet vid Helmholtz-Zentrum Berlin (HZB) och Drug Design Group vid University of Marburg har etablerat ett nytt ämnesbibliotek. Den består av 1103 organiska molekyler som kan användas som byggstenar för nya läkemedel. MX -teamet har nu validerat detta bibliotek i samarbete med FragMAX -gruppen på MAX IV. HZB:s substansbibliotek är tillgängligt för forskning över hela världen och spelar också en roll i sökandet efter ämnen som är aktiva mot SARS-CoV-19.
För att läkemedel ska vara effektiva, de måste vanligtvis docka till proteiner i en organism. Som en nyckel i ett lås, en del av läkemedelsmolekylen måste passa in i urtag eller hålrum i målproteinet. Sedan flera år tillbaka, teamet vid avdelningen för makromolekylär kristallografi (MX) vid HZB under ledning av Dr Manfred Weiss tillsammans med Drug Design Group under ledning av professor Gerhard Klebe (University of Marburg) har därför arbetat med att bygga upp så kallade fragmentbibliotek. Dessa består av små organiska molekyler (fragment) med vilka de funktionellt viktiga hålrummen på ytan av proteiner kan sonderas och kartläggas. Proteinkristaller mättas med fragmenten och analyseras sedan med kraftfullt röntgenljus. Detta gör att tredimensionell strukturell information kan erhållas vid nivåer av atomupplösning. Bland annat, det är möjligt att ta reda på hur väl ett specifikt molekylfragment ansluter sig till målproteinet. Utvecklingen av dessa substansbibliotek skedde som en del av det gemensamma Frag4Lead -forskningsprojektet och finansierades av det tyska federala ministeriet för utbildning och forskning (BMBF).
MX-teamet (MX står för makromolekylär kristallografi) har nu publicerat designen av ett kemiskt varierat fragmentbibliotek som kallas "F2X-Universal" -biblioteket, som består av 1, 103 föreningar. Ett representativt urval av 96 föreningar har extraherats från detta bibliotek, som kallas F2X Entry Screen. Under publiceringen av biblioteket, detta urval har nu testats och validerats framgångsrikt av HZB:s MX-team vid MAX IV-röntgenkällan i Lund, Sverige och på BESSY II.
I studien, HZB- och MAX IV -teamen verifierade effektiviteten hos F2X Entry -biblioteket genom att screena endothiapepsin och Aar2/RnaseH -proteinkomplexet som målenzymer. I nästa steg, MX -teamet kommer att använda hela det universella biblioteket.
"För den aktuella studien, fragmentscreeningsexperterna på HZB — BESSY II arbetade mycket nära FragMAX -projektteamet vid MAX IV ”, sa Dr. Uwe Müller från MX -teamet på HZB som hjälpte till att sätta upp de tre MX -strålarna på BESSY II samt BioMAX -strålen vid MAX IV. "Detta gjorde det möjligt för båda parter att vidareutveckla sina egna teknikplattformar och använda dem för att avbilda funktionella ytor hos olika proteiner. Detta kommer att vara en utmärkt grund för framtida samarbete mellan MAX IV och HZB."