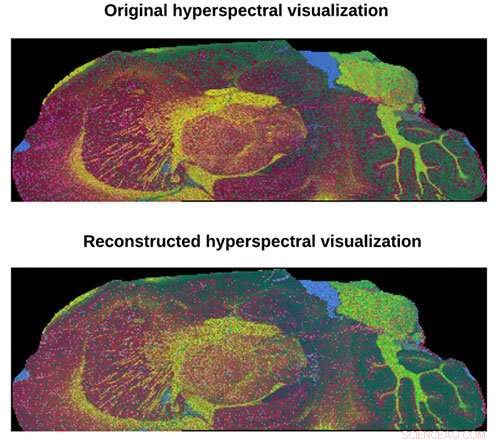
Topp, hyperspektral visualisering med data från ett standard 9-timmarsexperiment jämfört med hyperspektral visualisering med data från ett föreslaget 1-timmarsexperiment. Kredit:Beckman Institute for Advanced Science and Technology
En ny datorbaserad masspektrometrisk avbildningsmetod gör det möjligt för forskare att uppnå hög massupplösning och hög rumslig upplösning för biologiska prover samtidigt som de tillhandahåller datamängder exponentiellt snabbare.
Forskare vid Beckman Institute for Advanced Science and Technology utvecklade en subrymdmasspektrometrisk avbildningsmetod som accelererar datainsamlingshastigheten – utan att offra kvaliteten – genom att utforma en modellbaserad rekonstruktionsstrategi.
Tekniken, som utvecklades med hjälp av djurmodeller, kan få viktiga konsekvenser för många applikationer, inklusive analytisk kemi och kliniska studier, med resultat tillgängliga på en bråkdel av tiden. Den kan också detektera ett brett utbud av biomolekyler - från små molekyler som signalsubstanser och aminosyror till större molekyler som lipider eller peptider.
Uppsatsen "Accelerating Fourier Transform-Ion Cyclotron Resonance Mass Spectrometry Imaging Using a Subspace Approach" publicerades i Journal of the American Society of Mass Spectrometry .
"Fourier transform-jon cyklotronresonans är ett riktigt kraftfullt instrument, ger den högsta massupplösningen, sa Yuxuan Richard Xie, en bioingenjörsstudent vid University of Illinois Urbana-Champaign, som är första författare på tidningen. "Men en nackdel med FT-ICR är att den är väldigt långsam. Så i huvudsak, om människor vill uppnå en viss massupplösning, de måste vänta dagar för att skaffa datamängder. Vår beräkningsmetod påskyndar denna förvärvsprocess, potentiellt från en dag till kanske en till två timmar – i princip en tiofaldig ökning av datainsamlingshastigheten."
"Vår metod förändrar hur vi skaffar data, " sa Xie. "Istället för att skaffa masspektra per pixel, Tekniken känner igen redundansen i högdimensionell bilddata och använder en lågdimensionell delrumsmodell för att utnyttja denna redundans för att rekonstruera multispektrala bilder från endast en delmängd av datan."
Xie samarbetade med Fan Lam, en biträdande professor i bioteknik, och Jonathan V. Sweedler, James R. Eiszner-familjens begåvade ordförande i kemi och chef för School of Chemical Sciences, som är medansvariga utredare på tidningen. Daniel Castro, en doktorand i molekylär och integrativ fysiologi, också bidragit.
"Vi har använt subspace-modeller i vårt MRI- och MR-spektroskopiska avbildningsarbete under lång tid, " Sa Lam. "Det är verkligen trevligt att se att det också har stora möjligheter för en annan biokemisk avbildningsmodalitet."
"Förmågan att skaffa förbättrad kemisk information och var kemikalierna finns i ett komplext prov som en del av en hjärna blir möjlig för vår neurokemiska forskning, " sa Sweedler.
Konceptet för subspace imaging var banbrytande av Zhi-Pei Liang, en professor i el- och datateknik och heltidsanställd vid Beckman-fakulteten, som är en världsledande expert inom MR och MRSI.
Forskningen fortsätter när forskare försöker tillämpa tekniken på 3D-avbildning. "(Tillvägagångssättet) kan ha en mycket större inverkan för forskarvärlden för 3D-avbildning av större områden, som hjärnan, " sa Xie. "För om vi gör 50 skivor på FT-ICR, det skulle ta veckor nu, men (med denna teknik) kan vi uppnå anständig täckning kanske inom några dagar.
"Jag tror att datoravbildning, speciellt det datadrivna tillvägagångssättet, är som en ny lysande stjärna. Det blir mer och mer kraftfullt, och vi borde definitivt använda några av dessa tekniker för kemisk analys av vävnad genom masspektrometri avbildning."