Kredit:Kanazawa University
Forskare vid Kanazawa University rapporterar i Journal of Physical Chemistry Letters hur man simulerar 3D atomkraftmikroskopbilder av system som inte är i jämvikt som involverar biomolekyler. Tillvägagångssättet använder sig av en berömd ekvation från termodynamiken som är tillämplig på situationer utan jämvikt.
Tredimensionell atomkraftsmikroskopi (3D-AFM) är en teknik som används för att undersöka fördelningen av lösningsmedelsmolekyler vid fasta-vätskegränssnitt. Initialt användes metoden för att studera situationer där lösningsmedlet är vatten, metoden används nu även för andra molekyler. En ny utveckling är att använda 3D-AFM för att lösa organiseringen av biopolymerer som kromosomer eller proteiner i celler. På grund av komplexiteten hos sådana system behövs dock simuleringar av 3D-AFM-avbildningsprocessen för att hjälpa till med dess tolkning. Simuleringsmetoder som hittills utvecklats har antagit att det sonderade systemet är i jämvikt under AFM-skanningscykeln. Detta begränsar deras giltighet till situationer där lösningsmedelsmolekylerna rör sig mycket snabbare än skanningssonden. Takeshi Fukuma från Kanazawa University och kollegor har nu utvecklat en 3D-AFM-simuleringsmetod som fungerar för system som inte är i jämvikt, tillämpbar på situationer där molekylär rörelse sker på tidsskalor som är jämförbara med eller större än den för AFM-sonderingscykeln.
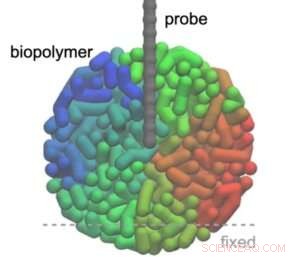
Den grundläggande principen för AFM är att göra en mycket liten spets, fäst vid en cantilever, för att skanna ett provs yta. Spetsens svar på höjdskillnader i den skannade ytan ger strukturell information om provet. I 3D-AFM görs spetsen för att penetrera provet, och kraften som spetsen upplever är resultatet av interaktioner med närliggande (delar av) molekyler. För en given horisontell (xy) position av spetsen fångas kraftens F beroende av spetsens vertikala (z) position när den penetrerar in i provet i en kraft-avståndskurva (F mot z). Genom att kombinera alla kraft-avståndskurvor erhållna under xy-skanningen ger 3D-AFM-bilden.
Fukuma och kollegor övervägde situationen där en AFM-spets sonderar en globulär biopolymer och modellerade både spetsen och molekylen som pärlor förbundna med fjädrar (2 000 pärlor för molekylen, 50 pärlor för spetsen). De beräknade kraft-avståndskurvorna genom att använda den så kallade Jarzynski-jämlikheten, en ekvation som relaterar den fria energiskillnaden mellan två tillstånd i ett system till det arbete (proportionellt mot kraften) som krävs för att gå från ett tillstånd till det andra. Viktigt är att jämlikheten gäller för icke-jämviktssituationer.
Forskarna kunde visa att simuleringarna återgav biopolymerens inre struktur, med vissa fiberegenskaper som tydligt kunde observeras. De tittade också på hur scanningshastigheten påverkar simuleringsresultaten och fann att det finns ett optimalt hastighetsområde för den vertikala (z) scanningen. Slutligen simulerade Fukuma och kollegor 3D-AFM-bilder av cytoskelettfibrer för vilka det finns experimentellt erhållna 3D-AFM-bilder och fann att simuleringarna stämmer väl överens med experimentet. Forskarna drog därför slutsatsen att deras metod "är tillämpbar på olika fibrer i celler som DNA och så vidare genom att ändra parametrar som styvhet, vilket ger en viktig teoretisk bas för sådana experimentella mätningar." + Utforska vidare