Att ta reda på vad som får vissa proteiner att lysa kräver förståelse för kemi. Kredit:eLife - tidskriften, CC BY-SA
Artificiell intelligens har förändrat sättet vetenskap görs på genom att tillåta forskare att analysera de enorma mängder data som moderna vetenskapliga instrument genererar. Den kan hitta en nål i en miljon höstackar av information och med hjälp av djupinlärning kan den lära sig av själva data. AI påskyndar framstegen inom genjakt, medicin, läkemedelsdesign och skapandet av organiska föreningar.
Deep learning använder algoritmer, ofta neurala nätverk som tränas på stora mängder data, för att extrahera information från ny data. Det skiljer sig mycket från traditionell datoranvändning med sina steg-för-steg-instruktioner. Den lär sig snarare av data. Deep learning är mycket mindre transparent än traditionell datorprogrammering, vilket lämnar viktiga frågor – vad har systemet lärt sig, vad vet det?
Som kemiprofessor gillar jag att designa prov som har minst en svår fråga som tänjer elevernas kunskaper för att fastställa om de kan kombinera olika idéer och syntetisera nya idéer och koncept. Vi har tagit fram en sådan fråga till AI-förespråkarnas affischbarn, AlphaFold, som har löst problemet med proteinveckning.
Proteinvikning
Proteiner finns i alla levande organismer. De förser cellerna med struktur, katalyserar reaktioner, transporterar små molekyler, smälter mat och gör mycket mer. De är uppbyggda av långa kedjor av aminosyror som pärlor på ett snöre. Men för att ett protein ska göra sitt jobb i cellen måste det vrida sig och böjas till en komplex tredimensionell struktur, en process som kallas proteinveckning. Felveckade proteiner kan leda till sjukdomar.
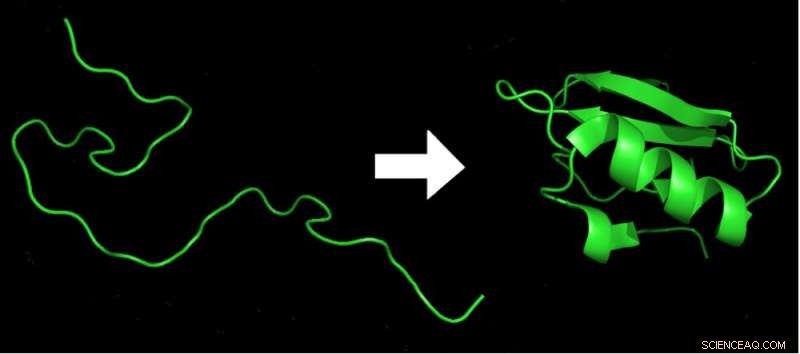
Inom millisekunder efter att en aminosyrakedja (vänster) gått ut från ribosomen viks den till 3D-formen med lägst energi (höger), vilket krävs för proteinets funktion. Kredit:Marc Zimmer, CC BY-ND
I sitt kemi-Nobel-acceptanstal 1972 postulerade Christiaan Anfinsen att det borde vara möjligt att beräkna den tredimensionella strukturen av ett protein från sekvensen av dess byggstenar, aminosyrorna.
Precis som bokstävernas ordning och avstånd i den här artikeln ger det mening och budskap, så avgör ordningen på aminosyrorna proteinets identitet och form, vilket resulterar i dess funktion.
På grund av den inneboende flexibiliteten hos aminosyrabyggstenarna kan ett typiskt protein anta uppskattningsvis 10 till 300 olika former. Detta är ett enormt antal, fler än antalet atomer i universum. Men inom en millisekund kommer varje protein i en organism att vikas till sin alldeles egna specifika form - det lägsta energiarrangemanget av alla kemiska bindningar som proteinet består av. Byt bara en aminosyra i de hundratals aminosyror som vanligtvis finns i ett protein och det kan vikas fel och inte längre fungera.
AlphaFold
I 50 år har datavetare försökt lösa problemet med proteinveckning – med liten framgång. Sedan 2016 initierade DeepMind, ett AI-dotterbolag till Googles moderbolag Alphabet, sitt AlphaFold-program. Den använde proteindatabanken som sin träningsuppsättning, som innehåller de experimentellt bestämda strukturerna av mer än 150 000 proteiner.
Neuroner som uttrycker fluorescerande proteiner avslöjar hjärnstrukturerna hos två fruktfluglarver. Kredit:Wen Lu och Vladimir I. Gelfand, Feinberg School of Medicine, Northwestern University
På mindre än fem år hade AlphaFold problemet med proteinveckning – åtminstone den mest användbara delen av det, nämligen att bestämma proteinstrukturen utifrån dess aminosyrasekvens. AlphaFold förklarar inte hur proteinerna viker sig så snabbt och exakt. Det var en stor vinst för AI, eftersom det inte bara samlade på sig enorm vetenskaplig prestige, det var också ett stort vetenskapligt framsteg som kunde påverka allas liv.
Idag, tack vare program som AlphaFold2 och RoseTTAFold, kan forskare som jag bestämma den tredimensionella strukturen hos proteiner från sekvensen av aminosyror som utgör proteinet – utan kostnad – på en timme eller två. Innan AlphaFold2 var vi tvungna att kristallisera proteinerna och lösa strukturerna med hjälp av röntgenkristallografi, en process som tog månader och kostade tiotusentals dollar per struktur.
Vi har nu också tillgång till AlphaFold Protein Structure Database, där Deepmind har deponerat 3D-strukturerna för nästan alla proteiner som finns i människor, möss och mer än 20 andra arter. Hittills har de löst mer än en miljon strukturer och planerar att lägga till ytterligare 100 miljoner strukturer bara i år. Kunskapen om proteiner har skjutit i höjden. Strukturen för hälften av alla kända proteiner kommer sannolikt att dokumenteras i slutet av 2022, bland dem många nya unika strukturer förknippade med nya användbara funktioner.
Tänker som en kemist
AlphaFold2 var inte designad för att förutsäga hur proteiner skulle interagera med varandra, men den har ändå kunnat modellera hur enskilda proteiner kombineras för att bilda stora komplexa enheter som består av flera proteiner. Vi hade en utmanande fråga för AlphaFold – hade dess strukturella träningsuppsättning lärt den lite kemi? Kan det avgöra om aminosyror skulle reagera med varandra - en sällsynt men viktig händelse?
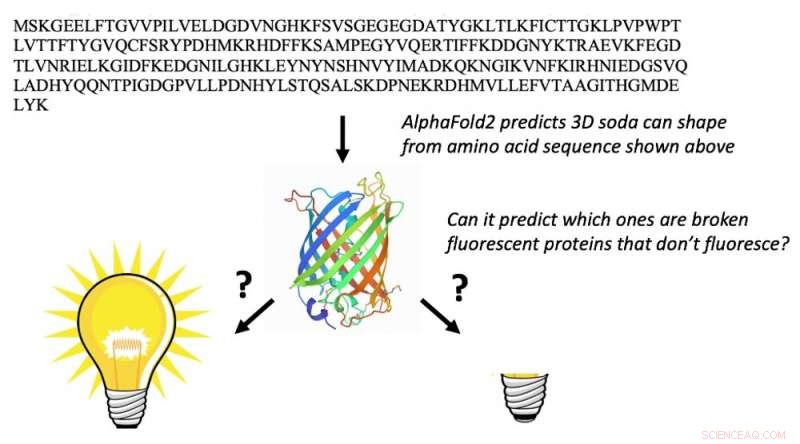
AlphaFold2 kan ta aminosyrasekvensen av fluorescerande proteiner (bokstäver längst upp) och förutsäga deras 3D-fatformer (mitten). Detta är inte förvånande. Det som är helt oväntat är att det också kan förutsäga vilka fluorescerande proteiner som är "trasiga" och inte kan fluorescera. Kredit:Marc Zimmer, CC BY-ND
Jag är en kemist som är intresserad av fluorescerande proteiner. Dessa är proteiner som finns i hundratals marina organismer som maneter och koraller. Deras glöd kan användas för att belysa och studera sjukdomar.
Det finns 578 fluorescerande proteiner i proteindatabanken, varav 10 är "trasiga" och fluorescerar inte. Proteiner attackerar sällan sig själva, en process som kallas autokatalytisk posttranslationsmodifiering, och det är mycket svårt att förutsäga vilka proteiner som kommer att reagera med sig själva och vilka som inte kommer att göra det.
Endast en kemist med en betydande mängd fluorescerande proteinkunskap skulle kunna använda aminosyrasekvensen för att hitta de fluorescerande proteiner som har rätt aminosyrasekvens för att genomgå de kemiska omvandlingar som krävs för att göra dem fluorescerande. När vi presenterade AlphaFold2 med sekvenserna av 44 fluorescerande proteiner som inte finns i proteindatabanken, vek den de fixerade fluorescerande proteinerna annorlunda än de trasiga.
Resultatet förbluffade oss:AlphaFold2 hade lärt sig lite kemi. Den hade räknat ut vilka aminosyror i fluorescerande proteiner som gör kemin som får dem att lysa. Vi misstänker att proteindatabankens träningsuppsättning och flera sekvensanpassningar gör det möjligt för AlphaFold2 att "tänka" som kemister och leta efter aminosyrorna som krävs för att reagera med varandra för att göra proteinet fluorescerande.
Ett hopfällbart program som lär sig lite kemi från sin träningsuppsättning har också bredare konsekvenser. Genom att ställa de rätta frågorna, vad mer kan man vinna på andra algoritmer för djupinlärning? Kan ansiktsigenkänningsalgoritmer hitta dolda markörer för sjukdomar? Kan algoritmer utformade för att förutsäga utgiftsmönster bland konsumenter också hitta en benägenhet för mindre stöld eller bedrägeri? Och viktigast av allt, är denna förmåga – och liknande språng i förmåga i andra AI-system – önskvärd? + Utforska vidare
Den här artikeln är återpublicerad från The Conversation under en Creative Commons-licens. Läs originalartikeln. 