Lipider är en klass av biomolekyler som spelar en viktig roll i många cellulära processer. Analyser som försöker karakterisera alla lipider i ett prov – så kallade lipidomics – är avgörande för att studera komplexa biologiska system.
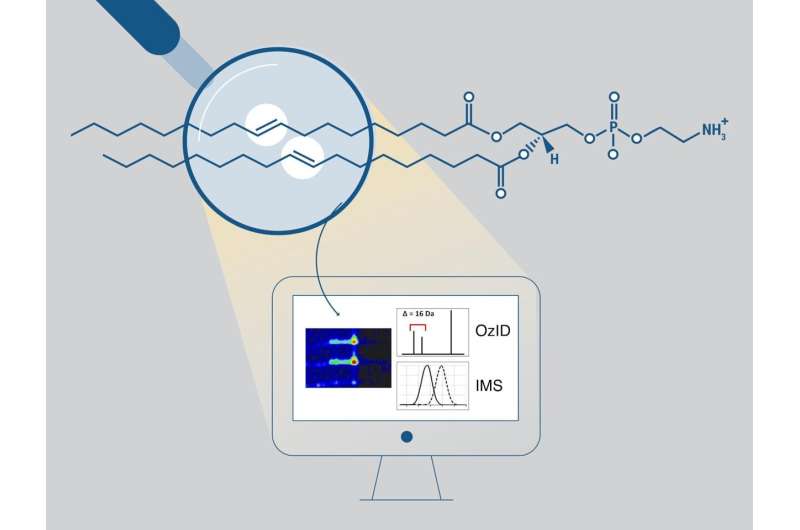
En viktig utmaning inom lipidomics är att koppla samman olika strukturer av lipider med deras biologiska funktioner. Positionerna för dubbelbindningarna inom fettsyrakedjorna är särskilt viktiga. Detta beror på att de kan påverka de fysiska egenskaperna hos cellulära membran och modulera cellsignaleringsvägar.
Denna information mäts inte rutinmässigt i lipidomikstudier eftersom den kräver en komplicerad experimentell uppställning som producerar komplexa data. Således utvecklade forskare vid Pacific Northwest National Laboratory (PNNL) ett strömlinjeformat arbetsflöde för att bestämma positionerna för dubbelbindningar. Detta arbetsflöde använder både automatisering och maskininlärning.
Deras nya metod, LipidOz, effektiviserar dataanalysen för att bestämma positionerna för dubbelbindningar. Genom att ta itu med denna nyckeldel av analysen av lipider, erbjuder LipidOz forskare en mer effektiv och korrekt metod för lipidkarakterisering. Studien är publicerad i tidskriften Communications Chemistry .
Den entydiga identifieringen av lipider kompliceras av närvaron av molekylära delar som har samma kemiska formel men olika fysiska konfigurationer. Specifikt inkluderar skillnaderna i dessa molekylära delar fettacylkedjelängden, stereospecifikt numrerad (sn) position och position/stereokemi för dubbelbindningar.
Konventionella analyser kan bestämma fettacylkedjelängderna, antalet dubbelbindningar och - i vissa fall - sn-positionen men inte positionerna för kol-kol-dubbelbindningar. Positionerna för dessa dubbelbindningar kan bestämmas med större säkerhet med hjälp av en gasfasoxidationsreaktion som kallas ozoninducerad dissociation (OzID), som producerar karakteristiska fragment.
Analysen av data som erhålls från denna reaktion är dock komplex och repetitiv, och det saknas stöd för mjukvaruverktyg. Python-verktyget med öppen källkod, LipidOz, bestämmer och tilldelar automatiskt dubbelbindningspositionerna för lipider med en kombination av traditionell automatisering och metoder för djupinlärning. Ny forskning visar denna förmåga för vanliga lipidblandningar och komplexa lipidextrakt, vilket möjliggör praktisk tillämpning av OzID för framtida lipidomikstudier.
Mer information: Dylan H. Ross et al, LipidOz möjliggör automatiserad belysning av lipid-kol-kol-dubbelbindningspositioner från ozoninducerade dissociationsmasspektrometridata, Communications Chemistry (2023). DOI:10.1038/s42004-023-00867-9
Journalinformation: Kommunikationskemi
Tillhandahålls av Pacific Northwest National Laboratory