Figur 1. Övergripande schema för Deep DDDI och förutsägelse av matbeståndsdelar som minskar in vivo-koncentrationen av godkända läkemedel. Kredit:Korea Advanced Institute of Science and Technology
Läkemedelsinteraktioner, inklusive interaktioner mellan läkemedel och läkemedel (DDI) och interaktioner mellan läkemedel och livsmedelsbeståndsdelar (DFIs), kan utlösa oväntade farmakologiska effekter, inklusive biverkningar av läkemedel (ADE), med orsaksmekanismer ofta okända. Dock, nuvarande förutsägelsemetoder ger inte tillräckliga detaljer utöver risken för DDI-förekomst, eller kräver detaljerad läkemedelsinformation som ofta inte är tillgänglig för DDI-förutsägelse.
För att ta itu med detta problem, Dr Jae Yong Ryu, Biträdande professor Hyun Uk Kim och framstående professor Sang Yup Lee, alla från Institutionen för kemi- och biomolekylär teknik vid Korea Advanced Institute of Science and Technology (KAIST), utvecklat ett beräkningsramverk, heter DeepDDI, som exakt förutsäger 86 DDI-typer för ett givet läkemedelspar. Forskningsresultaten publicerades online i Proceedings of the National Academy of Sciences ( PNAS ) den 16 april, 2018, som har titeln "Djup inlärning förbättrar förutsägelse av interaktioner mellan droger och droger."
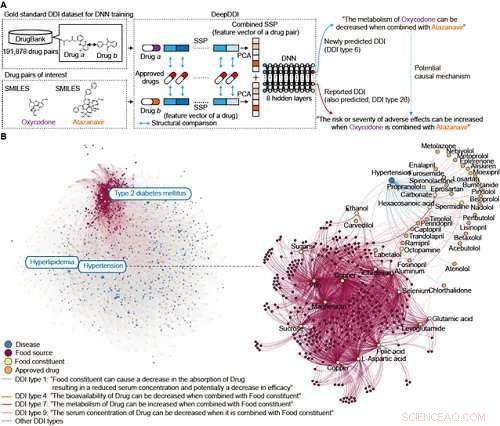
DeepDDI tar strukturell information och namn på två läkemedel i par som input, och förutsäger relevanta DDI-typer för det ingående läkemedelsparet. DeepDDI använder djupa neurala nätverk för att förutsäga 86 DDI-typer med en genomsnittlig noggrannhet på 92,4 % med hjälp av DrugBanks guldstandard DDI-datauppsättning som täcker 192, 284 DDI bidragit med 191, 878 drogpar. Mycket viktigt, DDI-typer som förutsägs av DeepDDI genereras i form av människoläsbara meningar som utdata, som beskriver förändringar i farmakologiska effekter och/eller risken för ADE till följd av interaktionen mellan två läkemedel i par. Till exempel, DeepDDI output meningar som beskriver potentiella interaktioner mellan oxikodon (opioid smärtstillande medicin) och atazanavir (antiretroviral medicin) genererades enligt följande:"Oxykodons metabolism kan minskas när det kombineras med Atazanavir"; och "Risken eller svårighetsgraden av biverkningar kan öka när Oxycodone kombineras med Atazanavir". Genom att göra det här, DeepDDI kan ge mer specifik information om läkemedelsinteraktioner utöver risken för förekomst av DDI eller ADE som vanligtvis rapporterats hittills.
DeepDDI användes först för att förutsäga DDI-typer av 2, 329, 561 läkemedelspar från alla möjliga kombinationer av 2, 159 godkända läkemedel, från vilka DDI-typer av 487, 632 drogpar förutspåddes nyligen. Också, DeepDDI kan användas för att föreslå vilket läkemedel eller livsmedel som ska undvikas under medicinering för att minimera risken för biverkningar av läkemedel eller optimera läkemedlets effektivitet. För detta ändamål, DeepDDI användes för att föreslå potentiella orsaksmekanismer för de rapporterade ADEs av 9, 284 drogpar, och även förutsäga alternativa drogkandidater för 62, 707 läkemedelspar har negativa hälsoeffekter för att bara behålla de gynnsamma effekterna. Vidare, DeepDDI applicerades på 3, 288, 157 par av drog-mat beståndsdelar (2, 159 godkända läkemedel och 1, 523 välkarakteriserade livsmedelsbeståndsdelar) för att förutsäga DFI. Effekterna av 256 livsmedelsbeståndsdelar på farmakologiska effekter av interagerande läkemedel och bioaktiviteter av 149 livsmedelsbeståndsdelar förutspåddes också slutligen. Alla dessa förutsägelseresultat kan vara användbara om en individ tar mediciner för en specifik (kronisk) sjukdom som högt blodtryck eller diabetes mellitus typ 2.
Den framstående professor Sang Yup Lee sa, "Vi har utvecklat en plattformsteknologi DeepDDI som kommer att möjliggöra precisionsmedicin under den fjärde industriella revolutionens era. DeepDDI kan tjäna till att tillhandahålla viktig information om läkemedelsförskrivning och kostförslag samtidigt som vi tar vissa läkemedel för att maximera hälsofördelarna och i slutändan hjälpa till att upprätthålla ett hälsosamt liv i detta åldrande samhälle."