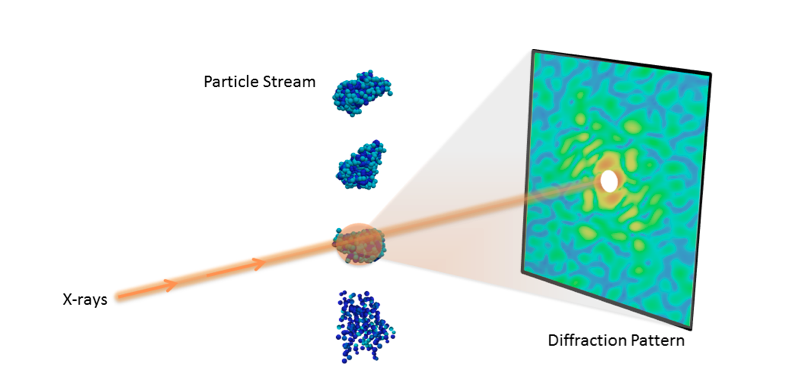
Experimentell installation för ett experiment med en partikel diffraktion. Upphovsman:Peter Zwart, Berkeley Lab
Att förstå 3D -molekylstrukturen för viktiga nanoobjekt som proteiner och virus är avgörande för biologi och medicin. Med de senaste framstegen inom röntgenteknik, forskare kan nu samla diffraktionsbilder från enskilda partiklar, slutligen låta forskare visualisera molekyler vid rumstemperatur.
Dock, att bestämma 3D-struktur från dessa enkelpartikeldiffraktionsexperiment är ett betydande hinder. Till exempel, nuvarande datainsamlingshastigheter är mycket begränsande, vanligtvis resulterar i färre än 10 användbara ögonblicksbilder per minut, begränsa mängden funktioner som kan lösas. Dessutom, bilderna är ofta mycket korrumperade av brus och andra experimentella artefakter, gör det svårt att tolka uppgifterna korrekt.
För att möta dessa utmaningar, ett team av forskare från Lawrence Berkeley National Laboratory (Berkeley Lab) har utvecklat ett nytt algoritmiskt ramverk som kallas multi-tiered iterative phasing (M-TIP) som använder avancerade matematiska tekniker för att bestämma 3D-molekylstruktur från mycket glesa uppsättningar av bullriga, enpartikeldata. Detta tillvägagångssätt tillåter i huvudsak forskare att extrahera mer information från experiment med begränsad data. Tillämpade matematiker Jeffrey Donatelli och James Sethian, och fysiska biovetaren Peter Zwart introducerade detta ramverk genom att utöka en algoritm som de ursprungligen utvecklade för att lösa rekonstruktionen från ett relaterat röntgenspridningsexperiment, kallas fluktuationsröntgenspridning. Ett papper som beskriver M-TIP-ramverket publicerades 26 juni i Förfaranden från National Academy of Sciences .
"Detta tillvägagångssätt har potential att revolutionera området, "säger Zwart." Med tanke på att det är svårt att få många bra data, tillvägagångssätt som minskar mängden data som behövs för att lyckas avbilda 3D -nanoobjekt kommer sannolikt att få ett varmt välkomnande. "
Donatelli, Sethian och Zwart är alla en del av CAMERA (The Center for Advanced Mathematics for Energy Research Applications), vars uppdrag är att skapa den toppmoderna matematiken som krävs för att hantera data från många av DOE:s mest avancerade vetenskapliga anläggningar. CAMERA finansieras gemensamt av programmen Advanced Scientific Computing Research och Basic Energy Sciences i DOE:s Office of Science.
Enstaka partikeldiffraktion
Den senaste tillkomsten av röntgenfrielektronlasrar (XFEL) har möjliggjort flera nya experimentella tekniker för att studera biomolekyler som var omöjliga för traditionella ljuskällor. En sådan teknik är enpartikeldiffraktion, som samlar ett stort antal röntgendiffraktionsbilder med bara en enda partikel i strålen. Genom att utnyttja XFEL:s extrema kraft, forskare kan samla mätbara signaler även från de minsta partiklarna.
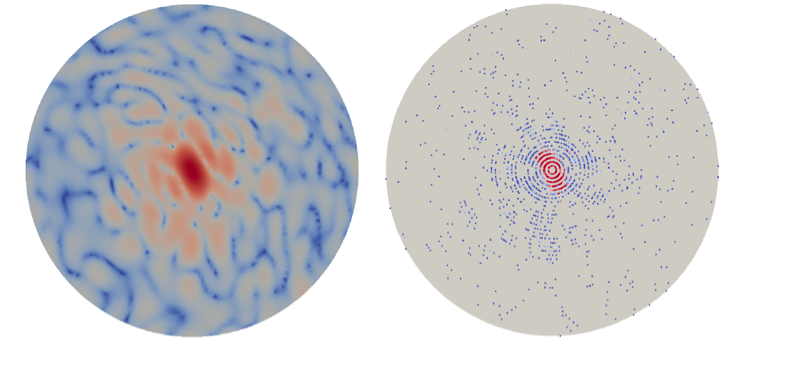
Ett exempel på en ren enpartikeldiffraktionsbild (vänster) och samma diffraktionsbild efter brusförorening (höger). Upphovsman:Peter Zwart, Berkeley Lab
En stor fördel med denna enkelpartikel-diffraktionsteknik är förmågan att studera hur olika kopior av en molekyl varierar eller förändras i form. Eftersom varje bild kommer från en enda partikel, dessa variationer kan fångas i experimentet, i kontrast till traditionella avbildningsmetoder som kristallografi eller röntgenspridning med liten vinkel, där forskare bara kan mäta ett genomsnitt över alla olika tillstånd i molekylprovet.
Dock, att bestämma 3D-strukturen utifrån enpartikeldiffraktionsdata är utmanande. Att börja, när varje partikel avbildas, dess orientering är okänd och måste återställas för att korrekt kunna kombinera data till en 3D -diffraktionsvolym. Detta problem förvärras om molekylen kan anta olika former, vilket kräver ytterligare klassificering av bilderna. Vidare, fasinformation spelas inte in i diffraktionsbilder och måste återställas för att slutföra rekonstruktionen. Till sist, även med kraftfulla XFEL, antalet spridda fotoner är mycket litet, resulterar i extremt bullriga bilder, som kan förorenas ytterligare av systematiska bakgrunds- och detektoravläsningsproblem.
Tidigare tillvägagångssätt är baserade på att lösa återuppbyggnadsproblemet i separata steg, där varje enskilt problem tas upp separat. Tyvärr, en nackdel med dessa seriella tillvägagångssätt är att de inte enkelt utnyttjar tidigare kända funktioner om hur molekylen ser ut. Dessutom, varje fel som begås i ett steg sprids till nästa, vilket resulterar i en ytterligare ökning av felen. Denna "fel snöboll" försämrar slutligen kvaliteten på rekonstruktionen som erhölls i det sista steget.
Bästa av båda världar
Istället för att lösa beräkningsproblemen i separata steg, teamets M-TIP-algoritm löser alla delar av problemet samtidigt. Detta tillvägagångssätt utnyttjar tidigare information om strukturen för att kraftigt minska problemets frihetsgrader i alla steg, och följaktligen minska den information som krävs för att uppnå en 3D-rekonstruktion.
"Standard black-box optimeringstekniker kan införliva förkunskaper i rekonstruktionen men kasta bort hela problemstrukturen, att lösa det i helt separata seriella delsteg utnyttjar problemets struktur men kastar bort nästan all tidigare information om hur lösningen kan se ut, "Sa Donatelli." M-TIP utnyttjar det bästa från båda världarna genom att utnyttja problemets struktur för att dela upp beräkningen i flera hanterbara bitar och sedan iterativt förfina över alla dessa bitar för att komma fram till en lösning som överensstämmer med båda data och eventuella strukturella begränsningar. "
Med denna teknik, laget kunde bestämma 3D -struktur från extremt låga bildantal från simulerade data, så lågt som 6 till 24 bilder för brusfria data och 192 bilder från mycket förorenad data.
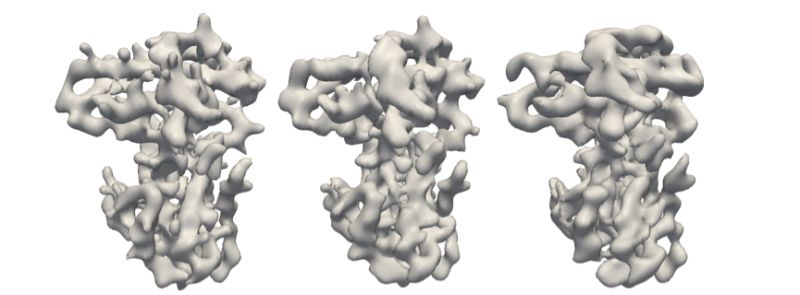
Ursprungligt retinoblastomprotein (vänster) och rekonstruktioner med M-TIP-algoritmen med 24 rena bilder (mitten) och 192 brusiga bilder (höger), som visas i figur 2. Kredit:Peter Zwart, Berkeley Lab
Bryter ny mark
Detta arbete är en del av ett nytt samarbetsinitiativ mellan SLAC National Accelerator Laboratory, KAMERA, National Energy Research Scientific Computing Center (NERSC) och Los Alamos National Laboratory som en del av DOE:s Exascale Computing Project (ECP). Målet med projektet är att utveckla de beräkningsverktyg som är nödvändiga för att utföra dataanalys i realtid från experiment som utförs vid SLAC:s Linac Coherent Light Source (LCLS). Med uppgraderingar av strållinjen, LCLS-II planerar att generera flera terabyte data per sekund, som, till exempel, kommer att göra det möjligt för forskare att kraftigt expandera på nuvarande experiment med enpartikel. Att analysera all denna data i realtid kommer att kräva nya algoritmer och stora datormaskiner. M-TIP-algoritmen kommer att fungera som en del av denna process.
"Det här är några av de mest utmanande problemen inom beräknad datavetenskap, " säger Sethian. "För att ta itu med dem, vi måste utnyttja en rad tekniker, inklusive framväxande exascale datorarkitekturer, sofistikerade höghastighetsnät, och de mest avancerade matematiska algoritmerna som finns. Att föra samman CAMERA-forskare med exascale-applikationsprojekt har öppnat dörren till att bygga verktyg för att närma sig några akuta problem inom biologi och materialvetenskap."
Forskarna noterar att detta bara är de första stegen. För att metoden ska vara klar att distribueras, andra hinder måste övervinnas.
"Experimentell vetenskap är rörig, "säger Zwart." Det finns ytterligare experimentella effekter som måste beaktas för att vi ska få bästa möjliga resultat. "
"Lyckligtvis, M-TIP är en mycket modulär teknik, "tillägger Donatelli, "så, det är väl lämpat att modellera många av dessa ytterligare effekter utan att behöva ändra det kärnalgoritmiska ramverket. "
Teamet arbetar för närvarande med att studera dessa effekter som en del av Single Particle Initiative, en stor, multiinstitutionellt samarbete för att ta itu med teoretiska och praktiska frågor inom X-FEL-baserad bildning av en enda molekyl, slutligen leder till att det vetenskapliga samfundet får de verktyg som behövs för att bryta ny mark inom biologi, medicin och energivetenskap.