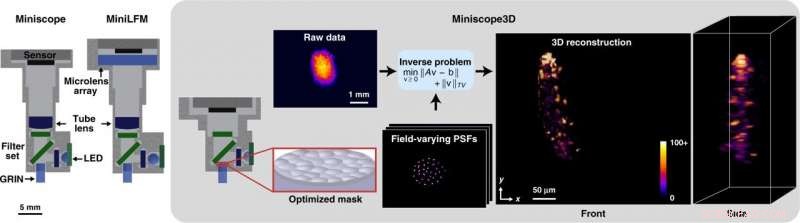
Miniscope3D systemöversikt. Jämfört med tidigare Miniscope- och MiniLFM-designer, vår Miniscope3D är lättare och mer kompakt. Vi tar bort Miniscopes rörlins och placerar en 55 μm tjock optimerad fasmask vid bländarstoppet (Fourier-planet) på GRIN-objektivet. En gles uppsättning (64 per djup) av kalibreringspunktspridningsfunktioner (PSF) fångas upp genom att skanna en 2,5 μm grön fluorescerande pärla genom hela volymen. Vi använder denna datamängd för att förberäkna en effektiv framåtmodell som exakt fångar fältvarierande aberrationer. Framåtmodellen används sedan för att iterativt lösa ett omvänt problem för att rekonstruera 3D-volymer från 2D-mätningar i engångsbilder. 3D-rekonstruktionen här är av en fritt simmande fluorescensmärkt tardigrad. Kredit:Light:Science &Applications, doi:10.1038/s41377-020-00403-7
Ett miniatyrfluorescensmikroskop som väger mindre samtidigt som det erbjuder hög upplösning jämfört med befintliga enheter kommer att ha en rad tillämpningar inom systembiologi. Befintliga miniatyrfluorescensmikroskop är en standardteknik inom biovetenskap, men de erbjuder bara tvådimensionell (2D) information. I en ny rapport nu på Nature Light:Science &Applications , Kyrollos Yanny, Nick Antipa och ett team av forskare i Joint Graduate Program in Bioengineering, Elektroteknik och datavetenskap vid University of California, Berkeley och Universite libre de Bruxelles Belgien, utvecklat ett single-shot 3-D fluorescensmikroskop. De konstruerade den nya enheten känd som Miniscope3D genom att ersätta rörlinsen på ett konventionellt 2-D miniskop med en optimerad multifokal fasmask vid objektivets bländarstopp. Använda enheten, Yanny och Antipa et al. optiskt registrerad neural aktivitet hos djur som rör sig fritt och vid långvariga in situ-avbildningstillämpningar i inkubatorer och i lab-on-a-chip-enheter.
Miniatyr fluorescensavbildning och tekniska innovationer
Miniatyrfluorescensmikroskop är viktiga inom systembiologin för optiska registreringar av neural aktivitet hos fritt rörliga djur, långsiktig in situ-avbildning i inkubatorer och medicintekniska produkter. Sådana mikroskop är också kända som "miniskop" och är gjorda av 3-D-tryckta delar, även om den erbjuder enbart 2D-fluorescensavbildning. Enkelbildsmetoder kan möjliggöra snabbare tagningshastigheter och en tidsmässig upplösning som begränsas av kamerans bildhastighet. Till exempel, ett tidigare utvecklat miniatyrljusfältsmikroskop (MiniLFM) kan bearbeta neural aktivitet med en optimerad algoritm. I det här arbetet, Yanny et al. utvecklat ett 3-D miniskop för att uppnå högre upplösning med lägre vikt jämfört med befintliga tekniker. Teamet testade de mikroskopiska kapaciteterna genom att avbilda fluorescerande upplösningsmål samt fritt simmande biologiska prover och mushjärnvävnad. De validerade de rekonstruerade resultaten i jämförelse med tvåfotonmikroskopi för att förstå gränserna för den nya tekniken.
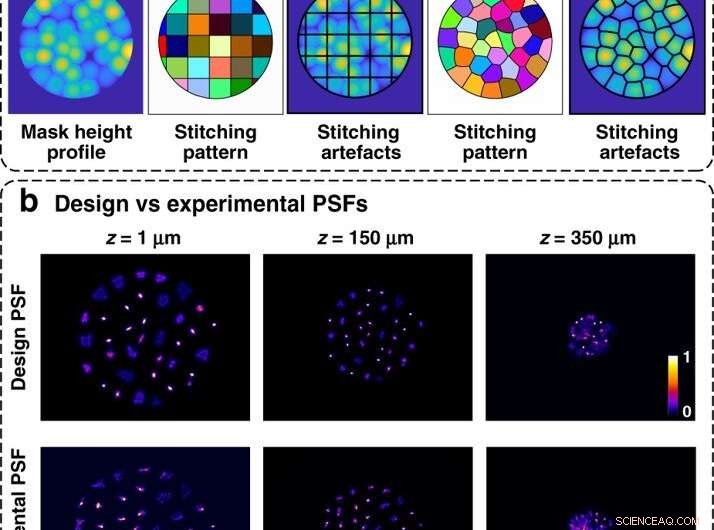
Fasmasktillverkning med nanoscribe. (a) Rektangulära sömmar leder till sömmar (svarta linjer) som går genom de många mikrolinserna, medan adaptiv sömnad placerar sömmarna vid gränserna för mikrolinserna för att mildra artefakter. (b) Jämförelse mellan designade och experimentella polyesterstapelfibrer på några få provdjup, visar god överensstämmelse, med lätt försämring vid kanten av volymen. Kredit:Light:Science &Applications, doi:10.1038/s41377-020-00403-7
För att uppnå högkvalitativ bildbehandling i en liten, låg vikt enhet, Yanny et al. placerade fasmasken (där ljus som passerar genom masken kommer att genomgå en fasförskjutning proportionell mot materialets tjocklek) i Fourier-utrymmet för att minska beräkningsbördan och förbättra kompaktheten. De lade till 3D-funktioner till 2D-miniskopet till priset av en liten förlust i sidoupplösning och lägre signal-brusförhållande. Algoritmen förenade den optiska teorin med komprimerad avkänning för att tillverka de optimerade fasmaskerna. Tekniken underlättade en ny miniatyr 3-D mikroskoparkitektur med högre upplösning, design med öppen källkod, tillverkning av högre kvalitet och ett effektivt kalibreringsschema eller rekonstruktionsalgoritm.
Karakterisera beräkningsmikroskopet och undersöka mushjärnan
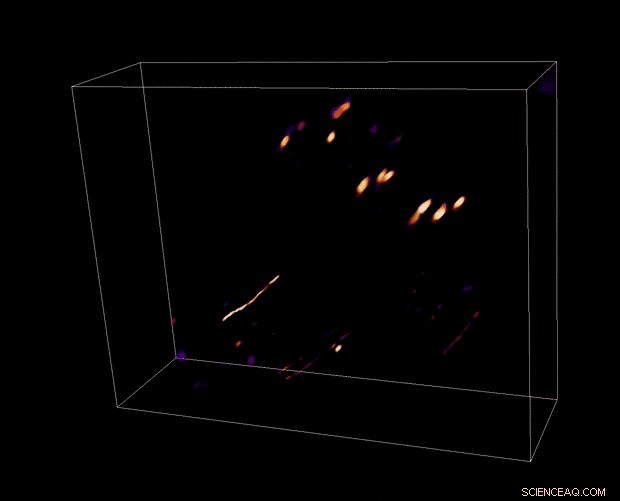
Teamet testade prestandan hos beräkningsmikroskopet med hjälp av prover av ökande komplexitet för att fånga 3-D dynamiska inspelningar. De mätte den laterala upplösningen på olika djup genom att avbilda ett fluorescerande upplösningsmål. De validerade sedan riktigheten av sina resultat med hjälp av tvåfotonmikroskopi. Till exempel, Miniscope3D kunde exakt återställa alla rekonstruerade bilder av det 3-D fluorescerande pärlprovet efter bearbetning. De visade potentialen för metoden med hjälp av neurobiologiska prover där grönt fluorescerande proteinmärkta regioner uttryckte glesa populationer av neuroner i hela provet. De rekonstruerade bilderna som erhölls från olika delar av hippocampus visade dendriter som löpte över ytan tillsammans med individuella cellkroppar. När Yanny et al. nästa undersökte dynamiska prover av frisim, grönfärgade tardigrader (även känd som vattenbjörnar), de rekonstruerade bilderna visade effektiviteten hos Miniscope3D-avbildning för att spåra fritt rörliga biologiska organismer med hög upplösning i rum-tid.
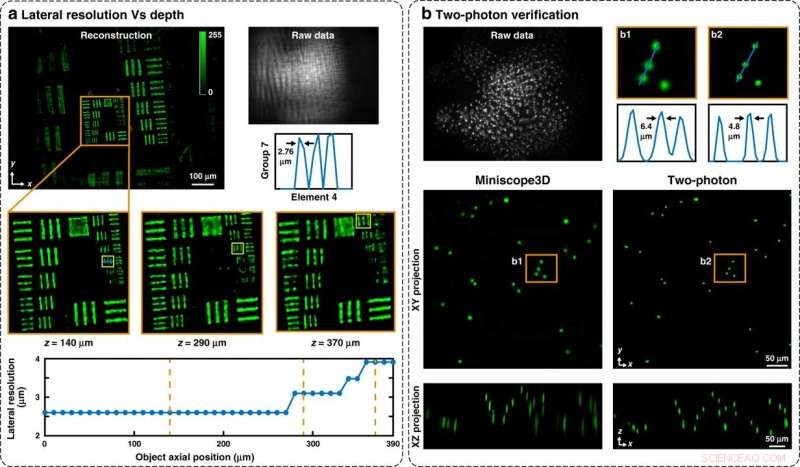
Experimentell karaktärisering. (a) Rekonstruktioner av ett fluorescerande USAF-mål vid olika axiella positioner för att bestämma den djupberoende laterala upplösningen. Vi återställer en upplösning på 2,76 μm över de flesta av djupintervallet på 390 μm, med det värsta fallet på 3,9 μm (streckade orangea linjer markerar de infällda platserna, och gula rutor i infällningarna indikerar de minst lösta grupperna). Observera att upplösningsmålet har diskreta upplösningsnivåer som resulterar i hopp i data och att upplösningen här hänvisar till gapet mellan staplarna, inte linjeparets bredd. (b) Rekonstruktion av ett 160 μm tjockt prov av 4,8 μm fluorescerande pärlor jämfört med en tvåfoton 3D-skanningsbild (maximal intensitetsprojektioner i yx- och zx-planen visas). Vårt system upptäcker samma funktioner, med en något större lateral fläckstorlek. Kredit:Light:Science &Applications, doi:10.1038/s41377-020-00403-7
Rekonstruktion av GFP-märkta neuroner i 300 µm tjock optiskt rensad mushjärna som visar enstaka neuronupplösning och tydligt upplösta dendriter som löper över volymen axiellt. Kredit:Light:Science &Applications, doi:10.1038/s41377-020-00403-7
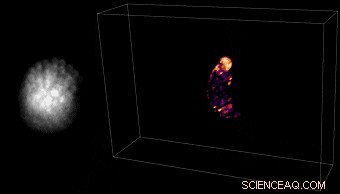
3D-rekonstruktion av fritt simmande tardigrader. (Vänster) Rådata. (Höger) Rekonstruktion av fritt rörliga SYBR-grönfärgade tardigrader. Kredit:Kredit:Light:Science &Applications, doi:10.1038/s41377-020-00403-7
Applikationer och tillgänglighet för enheten
De flesta tillämpningar av Miniscope3D kommer att likna 3D-mikroskopi och MiniLFM (miniatyrljusfältsmikroskopi), som anses vara guldstandarden för enkelbilds 3-D fluorescensavbildning i miniatyr. Jämfört med MiniLFM, dock, den nya Miniscope3D-metoden erbjöd flera förbättringar inklusive multifokala linser, bästa fall lateral upplösning och en 10-faldig ökning av den användbara mätvolymen. Den förbättrade prestandan kom i ett hårdvarupaket som var mindre än MiniLFM med lättare vikt för att fritt kunna observera rörliga organismer. Metoden möjliggjorde ytterligare experimentell rekonstruktion med eller utan spridning för mushjärnvävnad vid enstaka neuronupplösning. Teamet kommer att optimera befintliga gränser för enheten inklusive spridning, för ytterligare ansökningar.
Genom att bygga på en populär miniskopplattform med öppen källkod, Yanny et al. tillhandahållit tillgänglighet för Miniscope3D-designen. På det här sättet, Kyrollos Yanny, Nick Antipa och kollegor tillhandahöll en 3D-prototyp som en möjlighet att uppgradera de 2D-miniskop som för närvarande används i 450 laboratorier. De experimentella resultaten stämde väl överens med den teoretiska designen och analysen för att fungera som ett användbart ramverk för skräddarsydda enkelbilds 3-D-system.
© 2020 Science X Network