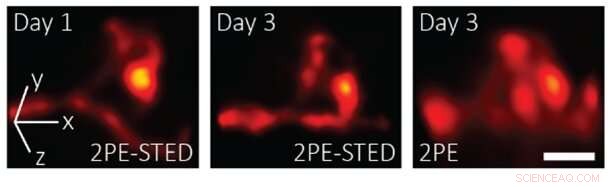
Forskare utvecklade ett 3D-2PE-STED-system som kan avbilda dendritiska ryggraden djupt inne i hjärnan på en levande mus. Deras system visade subtila förändringar som inträffade mellan dag 1 och 3 (vänster bilder). Dessa förändringar är svåra att särskilja med enbart tvåfotonmikroskopi (höger). Kredit:Joerg Bewersdorf, Yale School of Medicine
Forskare har utvecklat en ny mikroskopiteknik som kan ta 3-D superupplösta bilder av subcellulära strukturer från cirka 100 mikron djupt inuti biologisk vävnad, inklusive hjärnan. Genom att ge forskare en djupare inblick i hjärnan, metoden kan hjälpa till att avslöja subtila förändringar som inträffar i neuroner över tid, under inlärning, eller som ett resultat av sjukdom.
Det nya tillvägagångssättet är en förlängning av stimulerad emission depletion (STED) mikroskopi, en banbrytande teknik som uppnår upplösning i nanoskala genom att övervinna den traditionella diffraktionsgränsen för optiska mikroskop. Stefan Hell vann Nobelpriset i kemi 2014 för att ha utvecklat denna superupplösningsteknik.
I Optica , forskarna beskriver hur de använde sitt nya STED-mikroskop för att avbilda, i superupplösning, 3D-strukturen av dendritiska ryggraden djupt inne i hjärnan på en levande mus. Dendriska ryggraden är små utsprång på neuronernas dendritiska grenar, som tar emot synaptiska input från närliggande neuroner. De spelar en avgörande roll i neuronal aktivitet.
"Vårt mikroskop är det första instrumentet i världen för att uppnå 3D STED-superupplösning djupt inne i ett levande djur, ", sade ledaren för forskargruppen Joerg Bewersdorf från Yale School of Medicine. "Sådana framsteg inom djupvävnadsavbildningsteknologi kommer att tillåta forskare att direkt visualisera subcellulära strukturer och dynamik i sin inhemska vävnadsmiljö, ", sa Bewersdorf. "Förmågan att studera cellulärt beteende på detta sätt är avgörande för att få en heltäckande förståelse av biologiska fenomen för biomedicinsk forskning såväl som för läkemedelsutveckling."
Går djupare
Konventionell STED-mikroskopi används oftast för att avbilda odlade cellprover. Att använda tekniken för att avbilda tjock vävnad eller levande djur är mycket mer utmanande, speciellt när superupplösningsfördelarna med STED utvidgas till den tredje dimensionen för 3-D-STED. Denna begränsning uppstår eftersom tjock och optiskt tät vävnad hindrar ljus från att tränga in djupt och från att fokusera ordentligt, vilket försämrar superupplösningsförmågan hos STED-mikroskopet.
För att övervinna denna utmaning, forskarna kombinerade STED-mikroskopi med tvåfotonexcitation (2PE) och adaptiv optik. "2PE möjliggör avbildning djupare i vävnaden genom att använda nära-infraröda våglängder snarare än synligt ljus, " sa Mary Grace M. Velasco, tidningens första författare. "Infrarött ljus är mindre känsligt för spridning och, därför, är bättre i stånd att penetrera djupt in i vävnaden."
Forskarna lade också till adaptiv optik till sitt system. "Användningen av adaptiv optik korrigerar förvrängningar av ljusets form, dvs. de optiska aberrationerna, som uppstår vid bildtagning i och genom vävnad, " sade Velasco. "Under bildbehandling, det adaptiva elementet modifierar ljusvågfronten på exakt motsatt sätt som vävnaden i provet gör. Avvikelserna från det adaptiva elementet, därför, eliminera aberrationerna från vävnaden, skapa idealiska bildförhållanden som gör att STEDs superupplösningsförmåga kan återställas i alla tre dimensioner."
Att se förändringar i hjärnan
Forskarna testade sin 3-D-2PE-STED-teknik genom att först avbilda välkarakteriserade strukturer i odlade celler på ett täckglas. Jämfört med att använda enbart 2PE, 3-D-2PE-STED upplösta volymer mer än 10 gånger mindre. De visade också att deras mikroskop kunde lösa fördelningen av DNA i kärnan av mushudceller mycket bättre än ett konventionellt tvåfotonmikroskop.
Efter dessa tester, forskarna använde sitt 3-D-2PE-STED-mikroskop för att avbilda hjärnan på en levande mus. De zoomade in på en del av en dendrit och löste 3D-strukturen hos enskilda ryggar. De avbildade sedan samma område två dagar senare och visade att ryggradens struktur verkligen hade förändrats under denna tid. Forskarna observerade inga förändringar i nervcellernas struktur i deras bilder eller i mössens beteende som skulle tyda på skador från avbildningen. Dock, de planerar att studera detta vidare.
"Dendritiska ryggar är så små att utan superupplösning är det svårt att visualisera deras exakta 3D-form, än mindre förändringar av denna form över tid, ", sa Velasco. "3-D-2PE-STED ger nu möjlighet att observera dessa förändringar och att göra det inte bara i de ytliga lagren av hjärnan, men också djupare inuti, där fler av de intressanta kopplingarna sker."