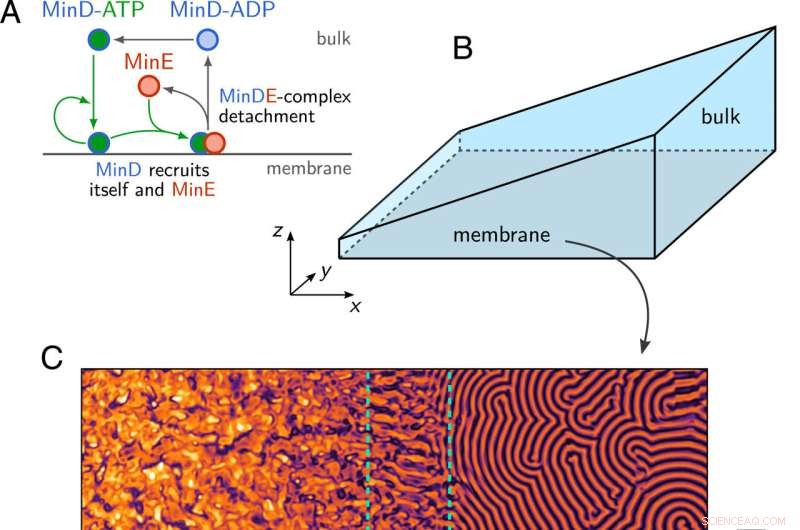
(A) Schematisk illustration av Min-proteinreaktionsnätverket. (B) Kilgeometri med en membranyta i bottenplanet (z =0) och bulkhöjden H(x) som ökar linjärt längs x-riktningen. (C) Ögonblicksbild av membrandensiteten för MinD, erhållen genom att numeriskt simulera Min-dynamikens ekv. 1–3 i geometrin som visas i B. Man observerar regioner med kaotiska mönster, stående vågor (SW, streckad grön kontur) och resande vågor (TW) längs membranet och på olika bulkhöjder. Kredit:Proceedings of the National Academy of Sciences (2022). DOI:10.1073/pnas.2206888119
För många processer som är viktiga för livet, såsom celldelning, cellmigration och utveckling av organ, är den spatialt och tidsmässigt korrekta bildningen av biologiska mönster avgörande. För att förstå dessa processer består huvuduppgiften inte i att förklara hur mönster bildas ur ett homogent initialtillstånd, utan i att förklara hur enkla mönster förändras till allt mer komplexa. Att belysa mekanismerna för denna komplexa självorganisering på olika rumsliga och tidsmässiga skalor är en nyckelutmaning för vetenskapen.
Så kallade "grovkorniga"-tekniker tillåter att sådana flerskaliga system förenklas, så att de kan beskrivas med en reducerad modell i stora längd- och tidsskalor. "Priset man betalar för grovkornighet är dock att viktig information om mönstren i små skalor — som mönstertypen — går förlorad. Men saken är den att dessa mönster spelar en avgörande roll i biologiska system. För att ge ett exempel , de kontrollerar viktiga cellulära processer", förklarar Laeschkir Würthner, medlem av teamet ledd av LMU-fysikern Prof. Erwin Frey och huvudförfattare till en ny studie publicerad i Proceedings of the National Academy of Sciences som övervinner denna fråga.
I samarbete med professor Cees Dekkers forskargrupp (TU Delft) har Freys team utvecklat ett nytt grovkornigt tillvägagångssätt för så kallade massbevarande reaktions-diffusionssystem, där storskalig analys av de totala densiteterna av de inblandade partiklarna möjliggör förutsägelse av mönster i små skalor.
Forskarna illustrerade potentialen i deras tillvägagångssätt med Min-proteinsystemet, en paradigmatisk modell för biologisk mönsterbildning. Bakterien E. coli använder olika Min-proteiner som cirkulerar i en cell för att bestämma var celldelningen äger rum. En avgörande faktor här är att de inblandade proteinerna förekommer med olika frekvenser beroende på deras placering i cellen och kemiskt tillstånd – det vill säga de har en mängd olika densiteter.
"Vi har nu lyckats minska komplexiteten i det här systemet genom att utveckla en teori som enbart baseras på proteinernas totala täthet, så att vi helt kan spegla dynamiken i mönsterbildning", säger Frey. "Detta är en enorm minskning. De numeriska beräkningarna är nu klara på några minuter istället för månader."
Forskarna kunde experimentellt bekräfta teoretiska förutsägelser av modellen, enligt vilken fördelningen av proteinerna beror på omgivningens geometri. De gjorde detta genom att rekonstruera Min-proteinsystemet i en in vitro-flödescell, med resultaten som visade samma proteinmönster som avslöjades i simuleringen.
"Sådan rekonstruktion av information i en liten skala från reducerad dynamik på makroskopisk nivå öppnar nya vägar för en bättre förståelse av komplexa flerskaliga system, som förekommer i ett brett spektrum av fysiska system", säger Frey. + Utforska vidare