I mikroskopins ständigt utvecklande område har de senaste åren sett anmärkningsvärda framsteg i både hårdvara och algoritmer, vilket driver vår förmåga att utforska livets oändliga underverk. Men resan mot tredimensionell strukturerad belysningsmikroskopi (3DSIM) har hämmats av utmaningar som härrör från hastigheten och invecklad polarisationsmodulering.
Gå in i 3DSIM-systemet för höghastighetsmodulering "DMD-3DSIM", som kombinerar digital skärm med superupplöst bildbehandling, vilket gör att forskare kan se cellulära strukturer i oöverträffad detalj.
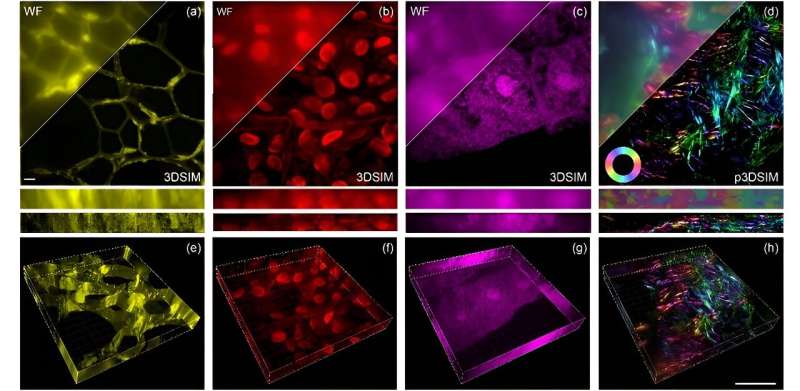
Som rapporterats i Advanced Photonics Nexus , utvecklade professor Peng Xis team vid Peking University denna innovativa uppställning kring en digital mikrospegelenhet (DMD) och en elektrooptisk modulator (EOM). Den hanterar upplösningsutmaningar genom att avsevärt förbättra både lateral (sida-till-sida) och axiell (topp-till-botten) upplösning, för en rumslig 3D-upplösning som enligt uppgift är dubbelt så stor som den som uppnås med traditionella bredfältstekniker.
Rent praktiskt betyder detta att DMD-3DSIM kan fånga intrikata detaljer av subcellulära strukturer, såsom kärnporkomplexet, mikrotubuli, aktinfilament och mitokondrier i djurceller. Systemets tillämpning utökades till att studera mycket spridande växtcellsultrastrukturer, såsom cellväggar i oleanderblad och ihåliga strukturer i svartalgblad. Även i en musnjurskiva avslöjade systemet en uttalad polarisationseffekt i aktinfilament.
En öppen inkörsport till upptäckt
Det som gör DMD-3DSIM ännu mer spännande är ett engagemang för öppen vetenskap. Xis team har gjort alla hårdvarukomponenter och kontrollmekanismer öppet tillgängliga på GitHub, vilket främjar samarbete och uppmuntrar det vetenskapliga samfundet att bygga vidare på denna teknik.
DMD-3DSIM-tekniken underlättar inte bara betydande biologiska upptäckter utan lägger också grunden för nästa generation av 3DSIM. I applikationer som involverar levande cellavbildning, lovar framsteg inom ljusare och mer fotostabila färgämnen, deoising-algoritmer och djupinlärningsmodeller baserade på neurala nätverk att förbättra avbildningstiden, informationshämtning och realtidsåterställning av 3DSIM-bilder från brusiga data. Genom att kombinera öppenhet för hårdvara och mjukvara hoppas forskarna kunna bana väg för framtiden för multidimensionell bildbehandling.
Mer information: Yaning Li et al, Höghastighets autopolarisationssynkroniseringsmodulering tredimensionell strukturerad belysningsmikroskopi, Advanced Photonics Nexus (2023). DOI:10.1117/1.APN.3.1.016001
Tillhandahålls av SPIE