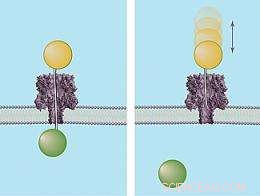
Grafik som visar hur "isfiske"-metoden bestämmer avståndet över ett membran nanopore. Båda bilderna visar DNA-strängar av kända längder toppade av ett polymerlock (orange sfär) som drivs genom nanoporen. Om DNA-strängen är tillräckligt lång för att helt tvärsöver kanalen (vänster), det kommer att "haka" en cirkulerande polymer (grön sfär) på andra sidan av membranet och definiera nanoporens längd. Om inte tillräckligt länge, DNA-sonden kommer att studsa ut ur poren (höger). Kredit:J. Robertson, NIST
(PhysOrg.com) -- Genom att använda ett par exotiska tekniker inklusive en molekylär skala version av isfiske, ett team av forskare som arbetar vid National Institute of Standards and Technology har utvecklat metoder för att exakt mäta längden på "nanoporer, ” de minimala kanalerna som finns i cellmembranen. De "molekylära linjalerna" som de beskriver i en nyligen publicerad artikel* skulle kunna fungera som ett sätt att kalibrera skräddarsydda nanoporer - vars diameter i genomsnitt är nästan 10, 000 gånger mindre än ett människohår – för en mängd olika tillämpningar som snabb DNA-analys.
Studier vid NIST och andra forskningsinstitutioner har visat att en enda por i nanometerskala i ett tunt membran kan användas som ett "miniatyranalyslaboratorium" för att upptäcka och karakterisera enskilda biologiska molekyler som DNA eller toxiner när de passerar genom eller blockerar passagen. . Ett sådant system skulle potentiellt kunna passa på en enda mikrochipenhet, för en mängd olika applikationer. Dock, att göra minilabbet praktiskt kräver en noggrann definition av dimensionerna och strukturella egenskaperna hos nanoporen.
I nya experiment, forskare från NIST och University of Maryland byggde först ett membran - ett dubbelskiktsark av lipidmolekyler - liknande det som finns i djurceller. De "borrade" en por i den med ett protein** utformat specifikt för att penetrera cellmembran. När spänning appliceras över membranväggen, laddade molekyler som enkelsträngat DNA tvingas in i nanoporen. När molekylen passerar in i kanalen, jonströmflödet reduceras under en tid som är proportionell mot kedjans storlek, så att dess längd lätt kan härledas.
Om en kedja är tillräckligt lång för att nå den smalaste delen av nanoporen – känd som klämpunkten – kommer kraften från det elektriska fältet bakom den att driva molekylen vidare genom resten av kanalen. Utnyttja denna egenskap, NIST/Maryland-teamet utvecklade en DNA-sondmetod för att mäta avstånden från öppningarna på varje sida av membranet till nyppunkten, och i sin tur, hela längden av nanoporen genom att lägga de två måtten tillsammans. Sonderna består av DNA-strängar av kända längder toppade i ena änden av en polymersfär. Sfären hindrar sonden från att helt röra sig genom nanoporen samtidigt som den lämnar DNA-kedjan hängande från den fri att sträcka sig in i kanalen. Om kedjan når klämpunkten, kraften som normalt skulle driva en fri DNA-kedja förbi korsningen håller istället sonden på plats (eftersom polymersfären "låser" den i andra änden) och definierar avståndet till nyppunkten. Om kedjan är kortare än avståndet till klämpunkten, det kommer att studsas ut ur nanoporen, berätta för forskare att en längre kedja behövs för att mäta avståndet till gapet.
NIST/Maryland-forskarna utvecklade också ett andra sätt att mäta längden på nanoporen för att bekräfta resultaten av metoden "single lollipop". I detta system, polymermolekyler tillåts cirkulera fritt i lösningen som finns på insidan av membranet. Polymertäckta DNA-sonder av olika längd tvingas en i taget in i nanoporen från den motsatta sidan. Om änden av en sonds kedja är tillräckligt lång för att helt tvärs över kanalen, den kommer att ta tag i en fri polymermolekyl i lösning. Detta definierar längden på kanalen.
Dessutom, denna "isfiske"-metod ger insikt i nanoporens struktur. När DNA-kedjan slingrar sig igenom, förändringar i elektrisk spänning motsvarar kanalens ändrade form. Denna information kan användas för att effektivt kartlägga passagen.