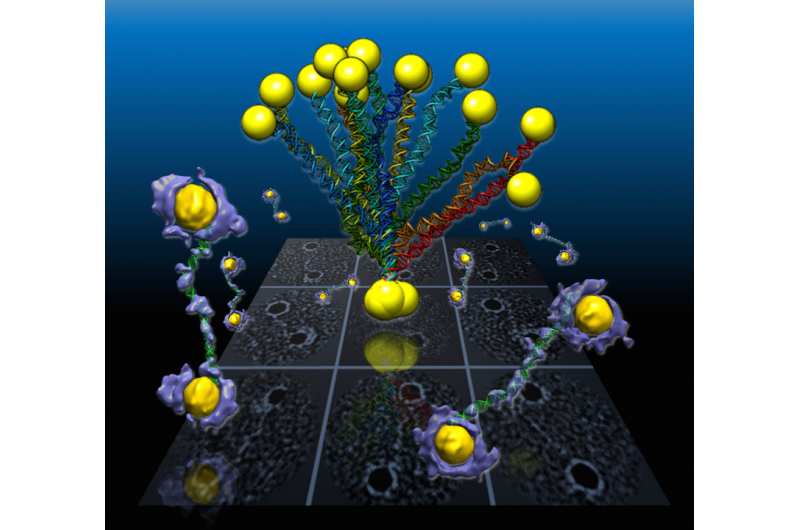
I en Berkeley Lab-ledd studie, flexibla dubbelhelix-DNA-segment kopplade till guldnanopartiklar avslöjas från 3D-densitetskartorna (lila och gula) som rekonstruerats från individuella prover med hjälp av en Berkeley Lab-utvecklad teknik som kallas individuell-partikelelektrontomografi eller IPET. Projektioner av strukturerna visas i bakgrundsrutnätet. Kredit:Berkeley Lab
Ett internationellt team som arbetar vid Department of Energys Lawrence Berkeley National Laboratory (Berkeley Lab) har tagit de första högupplösta 3D-bilderna från individuella dubbelhelix-DNA-segment fästa i vardera änden av guldnanopartiklar. Bilderna beskriver den flexibla strukturen av DNA-segmenten, som visas som hopprep i nanoskala.
Denna unika bildförmåga, banbrytande av Berkeley Lab-forskare, kan hjälpa till med användningen av DNA-segment som byggstenar för molekylära enheter som fungerar som läkemedelsleveranssystem i nanoskala, markörer för biologisk forskning, och komponenter för datorminnen och elektroniska enheter. Det kan också leda till bilder av viktiga sjukdomsrelevanta proteiner som har visat sig svårfångade för andra avbildningstekniker, och av sammansättningsprocessen som bildar DNA från separata, enskilda strängar.
Formerna på de lindade DNA-strängarna, som var inklämda mellan polygonformade guldnanopartiklar, rekonstruerades i 3D med hjälp av en banbrytande elektronmikroskopteknik i kombination med en proteinfärgningsprocess och sofistikerad programvara som gav strukturella detaljer i en skala av cirka 2 nanometer, eller två miljarddelar av en meter.
"Vi hade ingen aning om hur dubbelsträngat DNA skulle se ut mellan nanogoldpartiklarna, " sa gänget "Gary" Ren, en Berkeley Lab-forskare som ledde forskningen. "Detta är första gången för att direkt visualisera ett individuellt dubbelsträngat DNA-segment i 3-D, " sa han. Resultaten publicerades i den 30 mars upplagan av Naturkommunikation .

Gang Ren (stående) och Lei Zhang deltog i en studie vid Berkeley Labs Molecular Foundry som producerade 3-D-reproduktioner av individuella prover av dubbelhelix-DNA-segment fästa på guldnanopartiklar. Kredit:Roy Kaltschmidt/Berkeley Lab
Metoden som utvecklats av detta team, kallas individuell partikelelektrontomografi (IPET), hade tidigare fångat 3D-strukturen av ett enda protein som spelar en nyckelroll i människans kolesterolmetabolism. Genom att ta 2D-bilder av samma objekt från olika vinklar, tekniken gör det möjligt för forskare att sammanställa en 3D-bild av det objektet. Teamet har också använt tekniken för att avslöja fluktuationen hos ett annat välkänt flexibelt protein, humant immunglobulin 1, som spelar en roll i vårt immunförsvar.
För denna senaste studie av DNA-nanostrukturer, Ren använde en elektronstrålestudieteknik som kallas kryo-elektronmikroskopi (cryo-EM) för att undersöka frusna DNA-nanoguldprover, och använde IPET för att rekonstruera 3D-bilder från prover färgade med tungmetallsalter. Teamet använde också molekylära simuleringsverktyg för att testa de naturliga formvariationerna, kallas "konformationer, "i proverna, och jämförde dessa simulerade former med observationer.
Ren förklarade att den naturligt flexibla dynamiken hos prover, som en man som viftar med armarna, kan inte fullständigt detaljeras med någon metod som använder ett genomsnitt av många observationer.
Ett populärt sätt att se de strukturella detaljerna i nanoskala av ömtåliga biologiska prover är att forma dem till kristaller och zappa dem med röntgenstrålar, även om detta inte bevarar deras naturliga form och DNA-nanoguldproverna i denna studie är otroligt utmanande att kristallisera. Andra vanliga forskningstekniker kan kräva en samling av tusentals nästan identiska föremål, ses med ett elektronmikroskop, att kompilera en singel, genomsnittlig 3D-struktur. Men den här 3D-bilden kanske inte visar de naturliga formfluktuationerna för ett givet objekt.
Proverna i det senaste experimentet bildades från individuella polygon guld nanostrukturer, mäter cirka 5 nanometer tvärs över, kopplade till enkla DNA-segmentsträngar med 84 baspar. Baspar är grundläggande kemiska byggstenar som ger DNA dess struktur. Varje enskilt DNA-segment och guld-nanopartikel kneps naturligt ihop med en partner för att bilda det dubbelsträngade DNA-segmentet med en guldpartikel i vardera änden.
Proverna snabbfrystes för att bevara sin struktur för studier med kryo-EM-avbildning, och avståndet mellan de två guldpartiklarna i enskilda prover varierade från 20-30 nanometer baserat på olika former som observerats i DNA-segmenten. Forskare använde ett kryo-elektronmikroskop vid Berkeley Labs Molecular Foundry för denna studie.
De samlade en serie lutande bilder av de färgade föremålen, och rekonstruerade 14 elektrondensitetskartor som detaljerade strukturen för enskilda prover med hjälp av IPET-tekniken. De samlade ett dussin konformationer för proverna och fann att DNA-formvariationerna överensstämde med de som mättes i de snabbfrysta cryo-EM-proverna. Formerna överensstämde också med prover som studerats med andra elektronbaserade avbildnings- och röntgenspridningsmetoder, och med datorsimuleringar.
Medan 3D-rekonstruktionerna visar provernas grundläggande nanoskalastruktur, Ren sa att nästa steg blir att arbeta för att förbättra upplösningen till subnanometerskalan.
"Även i detta nuvarande tillstånd börjar vi se 3D-strukturer med 1- till 2-nanometers upplösning, ", sa han. "Genom bättre instrumentering och förbättrade beräkningsalgoritmer, det skulle vara lovande att driva upplösningen till att visualisera en enda DNA-helix i ett enskilt protein."
Tekniken, han sa, har redan väckt intresse bland några framstående läkemedelsföretag och nanoteknikforskare, och hans vetenskapsteam har redan dussintals relaterade forskningsprojekt i pipelinen.
I framtida studier, forskare kan försöka förbättra bildupplösningen för komplexa strukturer som innehåller fler DNA-segment som en sorts "DNA-origami, " sa Ren. Forskare hoppas kunna bygga och bättre karakterisera molekylära enheter i nanoskala med hjälp av DNA-segment som kan, till exempel, lagra och leverera läkemedel till riktade områden i kroppen.
"DNA är lätt att programmera, syntetisera och replikera, så det kan användas som ett speciellt material för att snabbt självmontera till nanostrukturer och för att styra driften av enheter i molekylär skala, ", sa han. "Vår nuvarande studie är bara ett bevis på konceptet för att avbilda dessa typer av molekylära enheters strukturer."