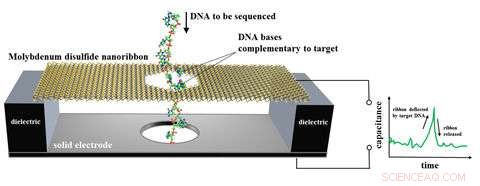
NIST:s föreslagna design för en DNA-sekvenserare baserad på en elektronisk rörelsesensor. Ett nanoskala band av molybdendisulfid hängs över en metallelektrod och nedsänks i vatten. Enkelsträngat DNA, som innehåller en kedja av baser (bitar av genetisk kod), träs genom ett hål i bandet, som böjer sig endast när en DNA-bas parar sig med och sedan separeras från en komplementär bas fäst vid hålet. Membranrörelsen detekteras som en elektrisk signal. Numeriska simuleringar och teoretiska uppskattningar visar att membranet skulle vara 79 till 86 procent exakt när det gäller att identifiera DNA-baser i en enda mätning med hastigheter upp till cirka 70 miljoner baser per sekund. Kredit:NIST
Forskare från National Institute of Standards and Technology (NIST) och samarbetspartners har föreslagit en design för den första DNA -sekvenseraren baserad på en elektronisk nanosensor som kan upptäcka små rörelser så små som en enda atom.
Den föreslagna enheten - en typ av kondensator, som lagrar elektrisk laddning - är ett litet band av molybdendisulfid suspenderat över en metallelektrod och nedsänkt i vatten. Bandet är 15,5 nanometer (nm, miljarddels meter) lång och 4,5 nm bred. Enkelsträngat DNA, som innehåller en kedja av baser (bitar av genetisk kod), träs genom ett 2,5 nm brett hål i det tunna bandet. Bandet böjer sig endast när en DNA-bas parar sig med och sedan separeras från en komplementär bas som är fäst vid hålet. Membranrörelsen detekteras som en elektrisk signal.
Som beskrivs i en ny tidning, NIST-teamet gjorde numeriska simuleringar och teoretiska uppskattningar för att visa att membranet skulle vara 79 till 86 procent exakt när det gäller att identifiera DNA-baser i en enda mätning med hastigheter upp till cirka 70 miljoner baser per sekund. Integrerade kretsar skulle detektera och mäta elektriska signaler och identifiera baser. Resultaten tyder på att en sådan enhet kan vara en snabb, exakt och kostnadseffektiv DNA-sekvenserare, enligt tidningen.
Konventionell sekvensering, utvecklades på 1970-talet, innebär att separera, kopiering, märkning och återmontering av bitar av DNA för att läsa den genetiska informationen. Nyare metoder inkluderar automatiserad sekvensering av många DNA-fragment på en gång - fortfarande kostsamma - och nya "nanopore-sekvensering"-koncept. Till exempel, samma NIST-grupp demonstrerade nyligen idén att sekvensera DNA genom att föra det genom en grafennanopor, och mäta hur grafens elektroniska egenskaper reagerar på belastning.
Det senaste NIST-förslaget bygger på en tunn film av molybdendisulfid - en stabil, skiktat material som leder elektricitet och används ofta som smörjmedel. Bland andra fördelar, detta material fastnar inte på DNA, vilket kan vara ett problem med grafen. NIST-teamet föreslår att metoden till och med kan fungera utan en nanopore - en enklare design - genom att skicka DNA över membranets kant.
"Det här tillvägagångssättet löser potentiellt problemet med att DNA fastnar på grafen om det sätts in på fel sätt, eftersom denna metod inte använder grafen, period, "NIST-teoretikern och huvudförfattaren Alex Smolyanitsky sa. "En annan stor skillnad är att istället för att förlita sig på egenskaperna hos grafen eller något speciellt material som används, vi läser av rörelser elektriskt på ett enklare sätt genom att bilda en kondensator. Detta gör vilket elektriskt ledande membran som helst lämpligt för applikationen."
Nanomaterialexperten Boris Yakobson från Rice University, en medförfattare på tidningen, föreslog kondensatoridén. Beräkningsstöd gavs av University of Groningen i Nederländerna.
DNA har fyra baser. För simuleringarna, cytosin (C), som naturligt går ihop med guanin (G), är fäst på insidan av poren. När en bit av DNA passerar genom poren, varje G i strängen fäster tillfälligt på det inbäddade C, dra i nanobandet och signalera elektroden. DNA-sekvensen bestäms genom att mäta hur och när elektriska blips varierar över tiden. För att upptäcka alla fyra baserna, fyra nanorband, var och en med olika bas fäst vid poren, kan staplas vertikalt för att skapa en integrerad DNA-sensor.
Molybdendisulfidbandet är tillräckligt flexibelt för att deformeras mätbart som svar på de krafter som krävs för att bryta upp ett DNA-par, men tillräckligt stel för att ha mindre pågående, meningslös rörelse än grafen, eventuellt reducera oönskat brus i sekvenssignalerna. Böjningen av bandet är ytterst liten, i storleksordningen en ångström, storleken på en väteatom. Dess dragkraft är i storleksordningen 50 piconewton, eller biljondelar av en newton, tillräckligt för att bryta upp de känsliga kemiska bindningarna mellan DNA -baser.
Forskare uppskattade hur enheten skulle fungera i en integrerad krets och fann att toppströmmarna genom kondensatorn var mätbara (50 till 70 pikoampere), även för de små studerade nanorbanden. Strömtopparna förväntas bli ännu större i fysiska system. Enhetens storlek kan justeras för att göra det ännu lättare att mäta sekvenssignaler.
NIST-författarna hoppas kunna bygga en fysisk version av enheten i framtiden. För praktiska tillämpningar, the chip-sized DNA sequencing microfluidic technology might be combined with electronics into a single device small enough to be handheld.