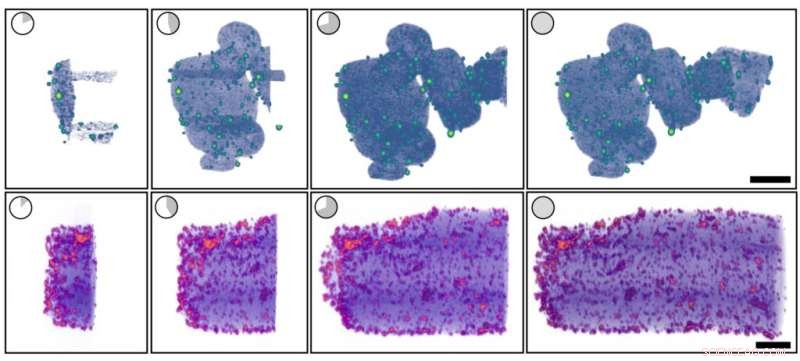
Denna rendering av platinananopartiklar på ett kolbärare visar hur tomviz tolkar mikroskopidata när de skapas, och löser sig från en skuggbild till en detaljerad rendering. Kredit:Jonathan Schwartz et al, Nature Communications (2022). DOI:10.1038/s41467-022-32046-0
Datachipdesigners, materialforskare, biologer och andra forskare har nu en oöverträffad nivå av tillgång till världen av nanoskalamaterial tack vare 3D-visualiseringsprogramvara som ansluts direkt till ett elektronmikroskop, vilket gör det möjligt för forskare att se och manipulera 3D-visualiseringar av nanomaterial i realtid .
Utvecklad av ett team av ingenjörer och mjukvaruutvecklare som leds av University of Michigan, är funktionerna inkluderade i en ny betaversion av tomviz, ett 3D-datavisualiseringsverktyg med öppen källkod som redan används av tiotusentals forskare. Den nya versionen återuppfinner visualiseringsprocessen, vilket gör det möjligt att gå från mikroskopprover till 3D-visualiseringar på några minuter istället för dagar.
Förutom att generera resultat snabbare gör de nya funktionerna det möjligt för forskare att se och manipulera 3D-visualiseringar under ett pågående experiment. Det kan dramatiskt påskynda forskningen inom områden som mikroprocessorer, elfordonsbatterier, lättviktsmaterial och många andra.
"Det har varit en långvarig dröm för halvledarindustrin, till exempel att kunna göra tomografi på en dag, och här har vi minskat det till mindre än en timme", säger Robert Hovden, biträdande professor i materialvetenskap och ingenjör vid U-M och motsvarande författare på tidningen, publicerad i Nature Communications . "Du kan börja tolka och göra vetenskap innan du ens är klar med ett experiment."
Hovden förklarar att den nya programvaran hämtar data direkt från ett elektronmikroskop när den skapas och visar resultat omedelbart, en grundläggande förändring från tidigare versioner av tomviz. Tidigare har forskare samlat in data från elektronmikroskopet, som tar hundratals tvådimensionella projektionsbilder av ett nanomaterial från flera olika vinklar. Därefter tog de projektionerna tillbaka till labbet för att tolka och förbereda dem innan de matades till tomviz, vilket skulle ta flera timmar att generera en 3D-visualisering av ett objekt. Hela processen tog dagar till en vecka, och ett problem med ett steg i processen innebar ofta att man började om.
Den nya versionen av tomviz gör all tolkning och bearbetning på plats. Forskare får en skuggig men användbar 3D-rendering inom några minuter, som gradvis förbättras till en detaljerad visualisering.
"När du arbetar i en osynlig värld som nanomaterial, vet du aldrig riktigt vad du kommer att hitta förrän du börjar se det," sa Hovden. "Så möjligheten att börja tolka och göra justeringar medan du fortfarande är i mikroskopet gör en enorm skillnad i forskningsprocessen."
Själva hastigheten i den nya processen kan också vara användbar inom industrin - halvledarchiptillverkare kan till exempel använda tomografi för att köra tester på nya chipdesigner och leta efter fel i tredimensionella kretsar i nanoskala som är alldeles för små för att se. Tidigare var tomografiprocessen för långsam för att köra de hundratals tester som krävdes i en kommersiell anläggning, men Hovden tror att tomviz kan ändra på det.
Hovden betonar att tomviz kan köras på en vanlig bärbar dator av konsumentklass. Den kan ansluta till nyare eller äldre modeller av elektronmikroskop. Och eftersom det är öppen källkod är själva programvaran tillgänglig för alla.
"Mjukvara med öppen källkod är ett utmärkt verktyg för att ge vetenskapen globalt makt. Vi gjorde kopplingen mellan tomviz och mikroskopet agnostisk för mikroskoptillverkaren," sa Hovden. "Och eftersom programvaran bara tittar på data från mikroskopet, bryr det sig inte om det mikroskopet är den senaste modellen på U-M eller en tjugo år gammal maskin."
För att utveckla de nya funktionerna, utgick UM-teamet från sitt mångåriga partnerskap med mjukvaruutvecklaren Kitware och anställde även ett team av forskare som arbetar i skärningspunkten mellan datavetenskap, materialvetenskap och mikroskopi.
I början av processen arbetade Hovden med Marcus Hanwell från Kitware och Brookhaven National Laboratory för att finslipa idén om en version av tomviz som skulle möjliggöra visualisering och experiment i realtid. Därefter samarbetade Hovden och Kitwares utvecklare med U-M materialvetenskap och ingenjörsforskare Jonathan Schwartz, mikroskopiforskaren Yi Jiang och maskininlärnings- och materialvetenskapsexperten Huihuo Zheng, båda från Argonne National Laboratory, för att bygga algoritmer som snabbt och exakt kan vända elektronmikroskopbilder till 3D-visualiseringar.
När algoritmerna var klara arbetade Cornell professor i tillämpad och teknisk fysik David Muller och Peter Ericus, en stabsforskare vid Berkeley Labs Molecular Foundry, tillsammans med Hovden för att designa ett användargränssnitt som skulle stödja de nya funktionerna.
Slutligen gick Hovden ihop med materialvetenskap och ingenjörsprofessor Nicholas Kotov, datavetare Jacob Pietryga, biogränssnittsforskaren Anastasiia Visheratina och kemiteknikforskaren Prashant Kumar, alla vid U-M, för att syntetisera en nanopartikel som kan användas för tester i verkligheten av de nya förmågorna, för att både säkerställa deras noggrannhet och visa upp sina förmågor. De slog sig ner på en nanopartikel formad som en helix, cirka 100 nanometer bred och 500 nanometer lång. Den nya versionen av tomviz fungerade som planerat; inom några minuter genererade den en bild som var skuggig men tillräckligt detaljerad för att forskarna skulle kunna urskilja viktiga detaljer som hur nanopartikeln vrids, känd som kiralitet. Cirka 30 minuter senare förvandlades skuggorna till en detaljerad, tredimensionell visualisering.
Källkoden för den nya betaversionen av tomviz är gratis tillgänglig för nedladdning på GitHub. Hovden tror att det kommer att öppna nya möjligheter för områden bortom materialrelaterad forskning; områden som biologi är också redo att dra nytta av tillgång till elektrontomografi i realtid. Han hoppas också att projektets tillvägagångssätt "programvara som vetenskap" kommer att stimulera ny innovation inom områdena vetenskap och mjukvaruutveckling.
"Vi har verkligen ett tvärvetenskapligt förhållningssätt till forskning i skärningspunkterna mellan datavetenskap, materialvetenskap, fysik, kemi," sa Hovden. "Det är en sak att skapa riktigt coola algoritmer som bara du och dina doktorander vet hur man använder. Det är en annan sak om du kan göra det möjligt för labb över hela världen att göra dessa toppmoderna saker."
Kitware-samarbetspartners i projektet var Chris Harris, Brainna Major, Patrick Avery, Utkarsh Ayachit, Berk Geveci, Alessandro Genova och Hanwell. Kotov är också Irving Langmuir Distinguished University Professor of Chemical Sciences and Engineering, Joseph B. och Florence V. Cejka Professor of Engineering, och professor i kemiteknik och makromolekylär vetenskap och teknik.
"Jag är exalterad över alla nya vetenskapliga upptäckter och 3D-visualiseringar som kommer att komma ut från materialvetenskapen och mikroskopigemenskapen med vårt nya ramverk för realtidstomografi," sa Schwartz. + Utforska vidare