
I en ny studie har forskare vid Institutionen för biokemi, Indian Institute of Science (IISc), använt en ny avbildningsteknik för att fastställa hur starkt intilliggande baser – byggstenarna i DNA – staplas ovanpå varandra i en enda sträng . Fynden öppnar upp möjligheter för att bygga komplexa DNA-nanoenheter och reda ut grundläggande aspekter av DNA-struktur.
Bakom den sömlösa driften av varje levande cell ligger DNA – det ärftliga fordonet som bär information för dess tillväxt, funktion och reproduktion. Varje DNA-sträng består vanligtvis av fyra nukleotidbaser - adenin (A), guanin (G), tymin (T) och cytosin (C). Baserna på en sträng parar sig med de på den motsatta strängen för att bilda dubbelsträngat DNA (A parar med T och G parar med C).
Två typer av interaktioner stabiliserar DNA:s dubbla helixstruktur. Basparning - interaktion mellan baser på motsatta strängar - är mer allmänt känd, medan basstapling - interaktion mellan baser i samma sträng - inte är särskilt väl studerad. Föreställ dig en dragkedja där basparning är som dragkedjan som håller ihop de två trådarna, medan basstapling fungerar som dragkedjans tänder, vilket säkerställer en tät och säker anslutning.
Bas-stacking-interaktioner är vanligtvis starkare än basparning, säger Mahipal Ganji, biträdande professor vid institutionen för biokemi, IISc, och motsvarande författare till artikeln publicerad i Nature Nanotechnology .
För att studera alla 16 möjliga basstaplingskombinationer använde forskarna DNA-PAINT (Point Accumulation in Nanoscale Topography). DNA-PAINT är en avbildningsteknik som fungerar utifrån principen att två artificiellt designade DNA-strängar - vardera slutar på olika bas - när de sätts samman i en buffertlösning vid rumstemperatur, kommer att binda och lossa till varandra slumpmässigt under en mycket kort tid .
Teamet märkte en av strängarna (imager-sträng) med en fluorofor som skulle avge ljus under bindning och testade staplingen av denna sträng ovanpå en annan dockad sträng. Bindningen och avbindningen av olika strängkombinationer (baserat på ändbaserna) togs som bilder under ett fluorescensmikroskop.
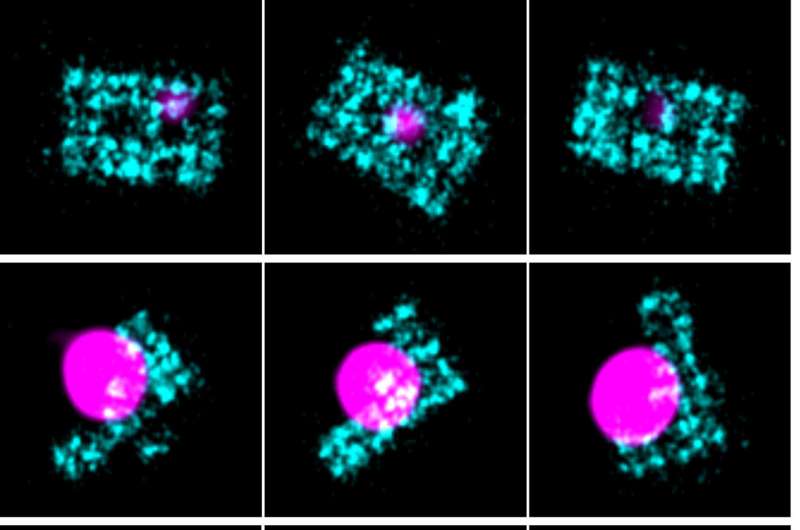
Tiden det tog för bindning och avbindning av strängarna visade sig öka om interaktionen mellan de staplade baserna var stark, förklarar Abhinav Banerjee, första författare och Ph.D. student vid institutionen för biokemi. Därför, med hjälp av data som erhållits från DNA-PAINT, byggde forskarna en modell som kopplade samman tidpunkten för bindning och avbindning med styrkan i interaktionen mellan de staplade baserna.
Med den här tekniken kunde teamet avslöja intressanta insikter om basstackning. Till exempel, att lägga till bara en basstaplingsinteraktion till till en DNA-sträng verkar öka dess stabilitet med upp till 250 gånger. De fann också att varje nukleotidpar hade sin egen unika staplingsstyrka. Denna information gjorde det möjligt för teamet att designa en mycket effektiv trearmad DNA-nanostruktur som potentiellt skulle kunna byggas in i ett polyederformat fordon för biomedicinska tillämpningar, som att rikta in sig på specifika sjukdomsmarkörer och leverera riktade terapier.
Forskarna arbetar också med att förbättra själva DNA-PAINT-tekniken. Banerjee säger att de med hjälp av staplingsinteraktioner planerar att designa nya sonder som skulle utöka de potentiella tillämpningarna av DNA-PAINT.
Dessutom har forskningen bredare tillämpningar bortom bildbehandling och nanoteknik, enligt forskarna. Ganji hoppas att dessa fynd kan användas för att studera grundläggande egenskaper hos enkel- och dubbelsträngat DNA som i sin tur kan belysa DNA-reparationsmekanismer, vars misslyckande leder till många sjukdomar inklusive cancer.
Mer information: Banerjee A, Anand M, Kalita S, Ganji M, Single-Molecule Analysis of DNA Base-Stacking Energetics Användning av mönstrade DNA-nanostrukturer, naturnanoteknologi (2023). www.nature.com/articles/s41565-023-01485-1
Journalinformation: Nanoteknik i naturen
Tillhandahålls av Indian Institute of Science