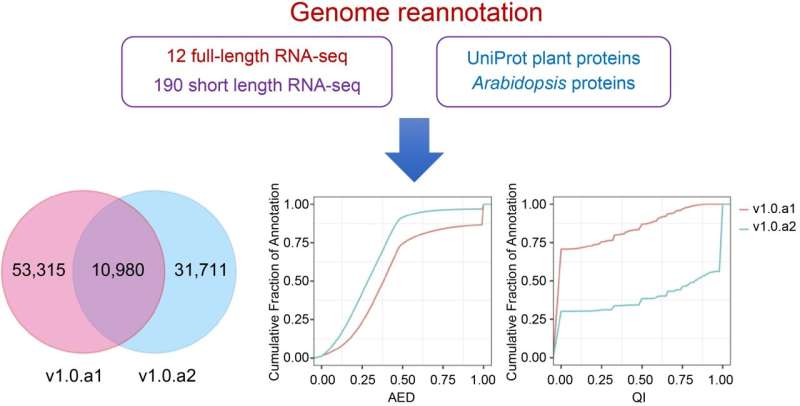
Ett forskarlag har avsevärt förbättrat annoteringen av sötpotatisgenomet "Taizhong 6", och introducerat en mer omfattande och detaljerad version, v1.0.a2. Den här uppdateringen använder 12 Nanopore RNA-bibliotek i full längd och 190 Illumina RNA-seq-bibliotek, vilket resulterar i identifiering av 360 nya gener, modifiering eller tillägg av 31 771 genmodeller och förfinad gennomenklatur.
Den förbättrade annoteringen inkluderar detaljerade miRNA-uttrycksprofiler, gynnar genfunktionella studier i sötpotatis och avancerade genomiska analyser över hela Convolvulaceae-familjen, vilket ger en viktig resurs för framtida jordbruks- och genetisk forskning.
Sötpotatis (Ipomoea batatas) är en viktig gröda i utvecklingsländer, som ger viktiga näringsämnen och bekämpar A-vitaminbrist i Afrika som en hexaploid art. Den första genomsekvensen, cultivar Taizhong 6, använde Illumina-sekvensering, senare förbättrad med tredje generationens sekvenseringsteknologier, vilket ledde till en högkvalitativ kromosomskalad genomsammansättning. Men det kvarstår utmaningar med att uppnå exakta genomannoteringar på grund av begränsningarna med kortläst sekvensering.
Nyligen har framsteg inom långläst sekvensering, som Oxford Nanopore Technologies, underlättat mer exakta anteckningar och möjliggjort djupare insikter i genstruktur och alternativ splitsning.
En studie publicerad i Tropical Plants den 21 mars 2024, bygger på dessa framsteg med hjälp av en förfinad annoteringspipeline som inkorporerade både Nanopore fullängds och omfattande RNA-seq-datauppsättningar, vilket förbättrar den nuvarande genomannoteringen för sötpotatis.
I den här studien förbättrade forskarna annoteringen av I. batata-genomet till version 1.0.a2, genom att använda ett omfattande tillvägagångssätt som integrerar Nanopore fullängdstranskriptom och Illumina RNA-seq-data över olika utvecklingsstadier och vävnader hos sötpotatis.
Deras metod använde BRAKER för initiala genförutsägelser, berikad av olika genomiska tips, följt av konsensusmodellgenerering genom EVidenceModeler (EVM). Noterbart är att den uppdaterade annoteringen nu innehåller 42 751 proteinkodande gener, vilket förbättrar modellen med 3' och 5' UTR och ökar det genomsnittliga exonantalet per gen.
Det är viktigt att denna revision lade till eller modifierade 31 771 genmodeller, som inkorporerade 8 736 alternativa splitsningsisoformer, och introducerade en ny gennomenklatur för tydligare referens. Denna mer detaljerade anteckning hjälper till med exakta genomiska studier och stöder avancerad funktionell genomik i sötpotatis.
Dessutom erbjuder integrering av miRNA-data och deras mål nya insikter i genreglering, särskilt under olika utvecklingsstadier av lagringsrötter, vilket förbättrar vår förståelse för sötpotatisbiologi och hjälper till med riktade avelsinsatser. De omfattande genfunktionsförutsägelserna utfördes med InterProScan och eggNOG-mapper, vilket ger en rikare anteckning som är avgörande för pågående forskning och avelsprogram fokuserade på att förbättra sötpotatissorter för globalt jordbruk.
Enligt studiens ledande forskare, professor Guopeng Zhu, "vår studie bidrar till en uppdaterad genomkommentar för sötpotatisgenomet, vilket avsevärt kommer att underlätta genfunktionella studier i sötpotatis och främja genomiska analyser över hela familjen Convolvulaceae."
Sammantaget underlättar detta förbättrade genomiska ramverk djupare funktionell genomik i sötpotatis och stödjer avancerade avelsprogram genom att integrera detaljerade miRNA-data och genfunktionsförutsägelser för att förbättra kultivaregenskaper.
Mer information: Bei Liang et al, Genomomnotering av sötpotatisen (Ipomoea batatas (L.) Lam.) med hjälp av omfattande Nanopore- och Illumina-baserade RNA-seq-datauppsättningar, Tropical Plants (2024). DOI:10.48130/tp-0024-0009
Tillhandahålls av Chinese Academy of Sciences