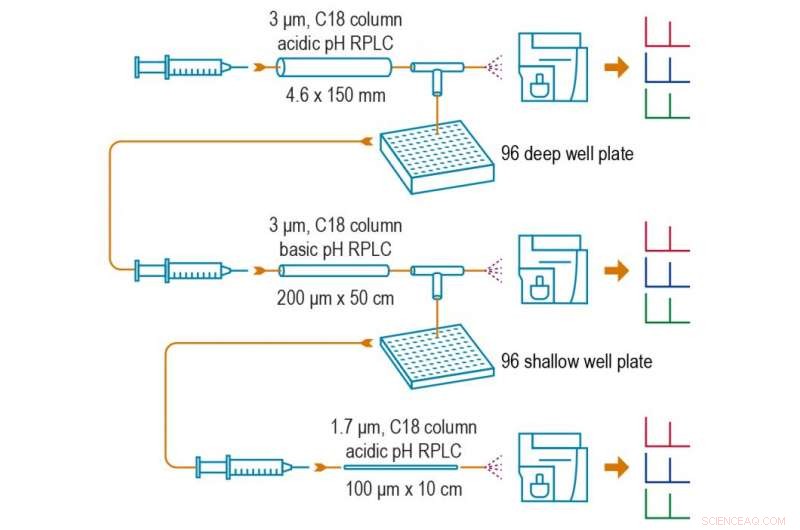
PNNL:s djupdykning SRM-metod erbjuder ett nytt sätt att förbereda och analysera blodprover, och gör det möjligt att upptäcka proteiner som finns i mycket låga koncentrationer – så lite som 10 pikogram per milliliter av ett blodprov. Metoden har en process i tre steg:1) Skär det initiala provet i 96 fraktioner och isolera fraktionen med proteinet av intresse, 2) Separera just den fraktionen i ytterligare 96 fraktioner och isolera återigen fraktionen som innehåller proteinet av intresse, och 3) Injicera det resulterande berikade provet i en masspektrometer för att analysera närvarande proteiner. Kredit:Pacific Northwest National Laboratory
Människor skulle kunna börja få livräddande behandling för cancer och andra sjukdomar mycket tidigare med hjälp av en ny metod att analysera blodproteiner.
En artikel av forskare vid Pacific Northwest National Laboratory beskriver ett nytt sätt att förbereda och analysera blodprover som avsevärt förbättrar forskarnas förmåga att upptäcka proteiner som finns i mycket låga koncentrationer - så lite som 10 pikogram per milliliter blod. Så liten, svårupptäckta koncentrationer är vanliga med sjukdomsindikerande proteiner, som också kallas biomarkörer.
Många forskare identifierar proteiner genom att använda antikroppar, Y-formade proteiner som tar tag i proteiner som de är speciellt utformade för att rikta in sig på. Men många proteiner har antingen inte matchande antikroppar eller så är antikropparna de har inte tillräckligt bra för att hitta proteiner när få av dem är närvarande.
Forskare har länge använt antikroppar på detta sätt, men masspektrometri ger ett attraktivt alternativ som är mer effektivt för att identifiera proteiner. För att analysera ett givet protein i blod eller andra biologiska prover, forskare använder en masspektrometrisk metod som kallas vald reaktionsövervakning, även känd som SRM. SRM letar specifikt efter unika peptider som indikerar närvaron av ett målprotein.
Under 2012, PNNL-forskare förbättrade standard SRM när de utvecklade en ny metod som heter PRISM-SRM, som separerar ett prov i 96 fraktioner och bestämmer vilken av dessa fraktioner som innehåller målproteiner. Detta gör det möjligt för forskare att köra ett berikat prov med mer riktade proteiner genom masspektrometern. Som ett resultat, den kan upptäcka proteiner som finns i koncentrationer mellan 1 nanogram och 100 pikogram per milliliter i blod. Känslighetsnivån på 100 pikogram uppstår när vanliga proteiner som inte är relaterade till sjukdomen avlägsnas genom en process som kallas utarmning, medan känslighet på 1 nanogramnivå uppstår när vanliga proteiner inte är utarmade.
Men de ville ändå upptäcka proteiner som finns i ännu mindre mängder. För att köra ett prov med ännu fler av de önskade proteinerna genom en masspektrometer, ett team av PNNL-forskare ledda av Tao Liu skapade en ny process som börjar med större provkvantiteter i förväg. Efter att ha skurit det första provet i 96 fraktioner, forskarna isolerade fraktionen med målproteinet på samma sätt som de hade gjort med PRISM. Nästa, de separerade just den fraktionen i en annan 96 - och isolerade återigen fraktionen som innehöll proteinet av intresse.
Kallas deep-dive SRM, detta nya tillvägagångssätt gör det möjligt att rikta in sig på proteiner som finns i det ursprungliga blodprovet i koncentrationer så lite som 10 pikogram per milliliter - och utan att behöva antikroppar eller utarmning i förväg. Det ökar denna metods känslighet med cirka 100, 000 gånger jämfört med konventionell SRM-analys och cirka 100 gånger jämfört med PRISM-SRM.
Deep-dive SRM skulle kunna användas av forskare för att identifiera sjukdomsindikerande proteiner i alla biologiska eller kliniska prov innan de blir mer rikliga - inklusive potentiellt diagnostisera cancer innan en tumör kan upptäckas på andra sätt. Sådan tidig upptäckt kan öka chanserna att rädda en patients liv.