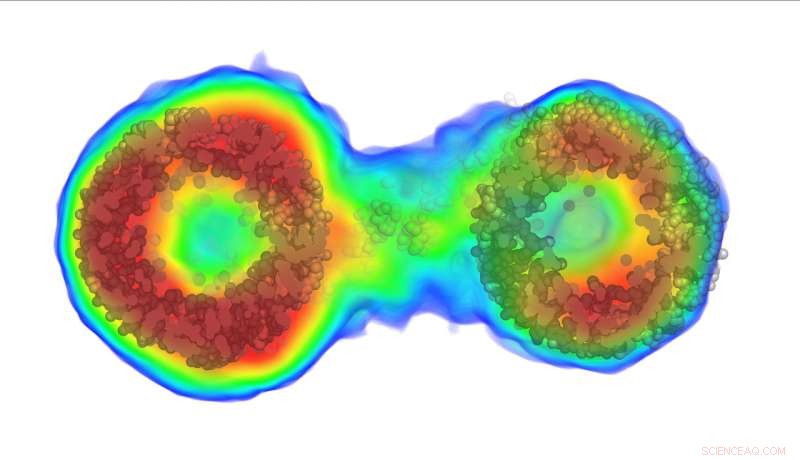
Tack vare den nya metoden, denna bild av en biomolekyl avslöjar dess invecklade inre struktur i orange, rött och gult. Tills nu, forskare hade bara kunnat se den blå konturen. Kreditera: Naturmetoder
Ny forskning publicerad i Naturmetoder kommer dramatiskt att förbättra hur forskare "ser inuti" molekylstrukturer i lösning, möjliggör mycket mer exakta sätt att bilddata i olika fält, från astronomi till upptäckt av droger.
Den nya metoden kommer att möjliggöra visualisering av många fler biologiska molekyler, ge kritisk information om vad som finns inuti molekyler till forskare som för närvarande bara kan komma åt sin yttre form eller hölje. Sådan information kan vara ett stort lyft för studier av virus, till exempel.
"Med befintlig teknik, du kan bara se konturen av viruset, "sa författaren Thomas D. Grant, Doktorsexamen, forskningsassistent professor vid institutionen för strukturbiologi vid Jacobs School of Medicine and Biomedical Sciences vid University of Buffalo och Institutionen för material, Design och innovation vid UB School of Engineering and Applied Sciences och Hauptman-Woodward Medical Research Institute. "Denna nya metod gör att vi kan se inuti virusmolekylen för att förstå hur den genetiska informationen är ordnad, potentiellt ge ny inblick i hur viruset injicerar denna genetiska information till sin värd. "
Grant är den enda författaren till tidningen, en sällsynthet bland tidningar publicerade i denna tidskrift. Han är forskare med BioXFEL (biologi med röntgenfria elektronlasrar), ett National Science Foundation Science and Technology Center bestående av åtta amerikanska forskningsuniversitet med huvudkontor vid UB. Dess uppdrag är att ta itu med grundläggande frågor inom biologi på molekylär nivå med hjälp av spetstekniker, inklusive röntgenlaservetenskap.
Löser fasproblemet
Grants metod har löst fasproblemet för en särskild molekylär bestämningsteknik som kallas lösningsspridning. Fasproblemet är där kritisk information om fasen i en molekyl går förlorad under den experimentella processen att göra en fysisk mätning.
Han förklarade att de flesta molekylära strukturer idag är lösta med röntgenkristallografi, där strukturerna sprider intensiva röntgenstrålar i mönster som består av hundratusentals unika bitar av information, som används för att slutligen avslöja strukturen vid högupplöst.
"Problemet är att mer än 75 procent av molekylstrukturerna inte lätt bildar de ordnade kristallerna som diffrakterar väl, "förklarade Grant." Det betyder att många molekyler är svåra att visualisera i tre dimensioner. "
Dessutom, han sa, biologiska molekyler kan uppvisa dynamiska rörelser som påverkar hur de fungerar men dessa rörelser saknas när strukturer kristalliseras, vilket förlorar viktig biologisk information.
En väg runt detta hinder är att använda en teknik som kallas lösningsspridning där röntgenstrålar sprids av molekyler som flyter i lösning istället för att placeras i en kristall.
"Spridning av lösningar gör att molekylerna kan röra sig dynamiskt i sina naturliga tillstånd, möjliggör visualisering av storskalig konformationsdynamik som är viktig för biologisk funktion, "sa Grant." Men när molekylerna tumlar i lösning, de sprider röntgenstrålarna i många olika riktningar, förlorar det mesta av informationen, ger vanligtvis bara 10 till 20 unika data. "Tills nu har sådan liten information gav endast konturer med låg upplösning av partikelformen.
Grant utvecklade en ny algoritm som möjliggör rekonstruktion av en tredimensionell elektrontäthet av en molekyl, liknande en 3D-rekonstruktion av hjärnan som produceras av en CT-skanning. Dock, hans algoritm gör detta med endast endimensionella data från lösningsspridningsexperiment.
Som att se ansiktsdrag istället för bara en silhuett
"För första gången, detta gör att vi kan "se inuti" dessa molekyler som flyter i lösning för att förstå de interna densitetsvariationerna istället för att bara se ytterkanterna eller "höljet" av partikelformen, "Grant sa." Som att kunna se alla en persons ansiktsdrag istället för bara silhuetten av deras ansikte, Denna extra information gör det möjligt för forskare att bättre förstå molekylära strukturer i lösningen. "
Han utvecklade den nya metoden genom att expandera på en välkänd matematisk teknik som kallas "iterativ fashämtning". Detta är en beräkningsteknik som ger ett sätt att lösa fasproblemet.
Grant förklarade:"Fasproblemet liknar att ha en kamera som exakt registrerar alla intensiteter för varje pixel, men krypterar där pixlarna är, baserat på en komplex matematisk ekvation. Så du sitter kvar med en värdelös bild av förvrängda pixlar. "
Forskare, han sa, har vanligtvis arbetat med att avkoda den matematiska ekvationen genom att ändra bilden lite för att se till att den ser ut ungefär som de förväntar sig. Till exempel, i ett liggande foto, de blå pixlarna som visar himlen ska naturligtvis vara överst.
Att lösa fasproblemet är som att avkoda den ekvationen, Grant fortsatte, och kunna placera alla pixlar där de ska vara, rekonstruera originalbilden.
"Dock, denna process förändrar vissa intensiteter, så du korrigerar dem utifrån den ursprungliga krypterade bilden du har, "sa han." Den här metoden kretsar iterativt igenom denna process, gradvis förbättra faserna för varje cykel, slutligen hämta de sista faserna, lösa fasproblemet och rekonstruera önskad bild. "
Grants metod, kallad "iterativ strukturfaktorhämtning, "gör det möjligt för forskare att rekonstruera inte bara de tredimensionella faserna utan också de tredimensionella intensiteterna som går förlorade i lösningsspridningsexperiment när molekylerna tumlar slumpmässigt i lösning.
"Detta är den första demonstrationen av förmågan att rekonstruera tredimensionella objekt från endimensionella experimentella data och det kommer sannolikt att ha stor inverkan på relaterade bildfält, " han sa.