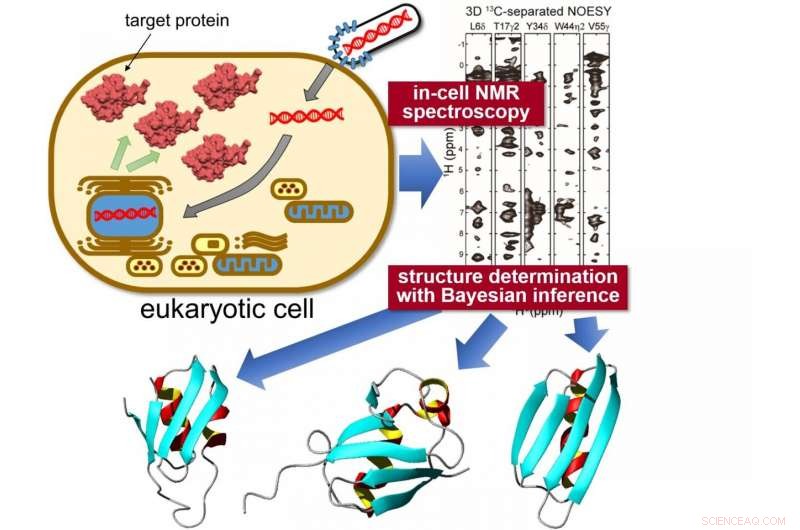
Målproteiner uttrycks inuti sf9 celler, mättes sedan med användning av "in-cell" NMR-spektroskopi. Unik statistisk analys med hjälp av Bayesiansk slutledning används för att beräkna den exakta 3D-strukturen av proteinerna med oöverträffad precision. Kredit:Tokyo Metropolitan University
Forskare från Tokyo Metropolitan University har framgångsrikt bestämt den högupplösta, tredimensionell struktur av proteiner inuti levande eukaryota celler. De kombinerade "in-cell" kärnmagnetisk resonans (NMR) spektroskopi, ett bioreaktorsystem och banbrytande beräkningsalgoritmer för att bestämma proteinstrukturer i trånga intracellulära miljöer för första gången. Tekniken lovar insikt i det intracellulära beteendet hos sjukdomsframkallande proteiner och nya läkemedelsscreeningapplikationer, möjliggör in-situ visualisering av hur proteiner svarar på biokemiska stimuli.
Eukaryota celler är byggstenarna i ett stort antal organismer, inklusive alla svampar, växter och djur. Deras inre struktur är extremt komplex och varierad, med en intrikat strukturell hierarki och ett stort utbud av biomakromolekyler fördelade runt ett cytoskelettnätverk. Detta har gjort det svårt att se vad varje protein inuti cellerna gör i sin naturliga miljö, trots de uppenbara biomedicinska fördelarna med att veta, t.ex. hur ett visst protein reagerar när celler utsätts för kemiska stimuli, som läkemedel.
För att tackla denna utmaning, ett team från Tokyo Metropolitan University ledd av biträdande professor Teppei Ikeya och professor Yutaka Ito tillämpade kärnmagnetisk resonans (NMR) spektroskopimätningar på specifika proteiner som uttrycks inuti sf9 odlade insektsceller, en stam av celler som ursprungligen härrörde från en typ av mallarver som ofta används för proteinproduktion. Teamets banbrytande NMR-arbete hade redan lyckats belysa högupplösta proteinstrukturer inuti bakterier (icke-eukaryoter). Problemet med att helt enkelt tillämpa samma tekniker på proteiner i sf9 celler var den signifikant lägre koncentrationen av målproteiner och kort livslängd för celler, vilket gör det svårt att samla in flerdimensionella NMR-spektra av hög kvalitet för nukleär Overhauser-effektspektroskopi (NOESY) som skulle ge exakt information om hur olika atomer är fördelade inuti enskilda molekyler. Således, de kombinerade ett gles provtagningsbaserat snabbt NMR-mätschema med state-of-the-art beräkningsmetoder som använder statistiska tekniker som Bayesiansk slutledning, metoder skräddarsydda för att belysa proteinstrukturer effektivt baserat på en begränsad mängd strukturell information från in-cell NMR-spektra med inneboende låg känslighet. Ett bioreaktorsystem var också utrustat inuti NMR-apparaten som höll cellerna i friskt tillstånd under mätningarna.
Med denna nya data, teamet kunde belysa 3D-strukturen av tre modellproteiner med oöverträffad hög upplösning, med en precision på 0,5 Ångström (0,05 nanometer) för positionen för proteinets huvudkedjeatomer. Särskilt, de identifierade en signifikant annorlunda konformation i en lokaliserad region av ett av proteinerna jämfört med dess referensstruktur i utspädd lösning. Konformationsskillnaden mellan proteiner "i celler" och "i provrör" orsakades förmodligen av icke-specifika interaktioner med andra molekyler inuti cellerna. Det blir tydligt att dessa interaktioner bidrar till proteinernas biologiska funktioner:förmågan att lokalisera och kvantifiera strukturella förändringar av proteiner i en intracellulär miljö förväntas ha en betydande inverkan på biomedicinsk forskning, gör det möjligt att se hur olika förhållanden t.ex. neurodegenerativa sjukdomar påverkar proteinkonformationer in situ, och kvantitativt mäta hur behandlingar påverkar strukturella anomalier.