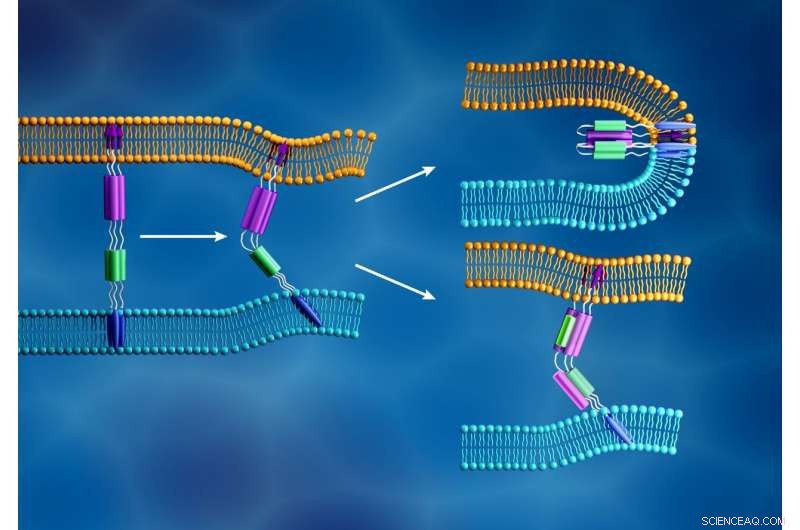
Det nya coronavirusmembranet (ljusblått) och det mänskliga cellmembranet (orange) smälter samman när den virala S2 -underenhetens fusionspeptid (lila pilar) infogas i cellmembranet och en annan komponent i S2 -subenheten (lila och gröna) veck för att bildas en tät struktur, som visas längst upp till höger. I kontrast, som visas längst ner till höger, fusionshämmare är utformade för att förhindra virusinfektion genom att störa denna process. Upphovsman:ORNL/Jill Hemman
SARS-CoV-2, coronaviruset som är ansvarigt för sjukdomen COVID-19, smittar världen i snabb takt. Att förstå hur denna infektion fungerar på molekylär nivå kan hjälpa experter att upptäcka sätt att mildra eller stoppa spridningen.
Ett team av forskare vid Department of Energy's (DOE:s) Oak Ridge National Laboratory (ORNL) använder neutronreflektometri för att göra just det.
Neutroner kan undersöka biologiska material under fysiologiska förhållanden utan att skada dem. Genom att utnyttja dessa egenskaper, forskarna kan mäta virusets infektionsdynamik när det händer.
Deras uppdrag är att få en detaljerad titt på några av de första stadierna av infektion som inträffar vid cellmembranet. Dessa fynd hjälper teamet att testa antivirala läkemedelskandidater som kan störa denna process. De data som erhållits från dessa experiment kan också informera andra studier fokuserade på att utveckla terapier och vacciner.
Forskarna fokuserar sin analys på SARS-CoV-2 spikproteiner, hullingliknande strukturproteiner som täcker virusets yta och utlöser infektionsprocessen. Spikproteinet binder till en receptor på värdcellens yttre skikt och underlättar sammansmältning mellan virala och cellulära membran, låta viruset komma in i cellen och släppa dess genetiska material. Cellens proteinframställningsmaskineri använder sedan denna genetiska information för att göra nya kopior av viruset.
När SARS-CoV-2 kapar en värdcell, dess spikprotein delas upp i två underenheter, kallas S1 och S2. De två delarna är båda viktiga för infektion. S1 -subenheten innehåller en receptorbindande domän som känner igen och låser fast en cellreceptor. Cellreceptorer är proteiner inbäddade i cellmembranet som kan binda till specifika molekyler utanför cellen. Denna anslutning kan få komponenterna att ändra form, vilket i sin tur kan inducera kaskadförändringar i cellen. För SARS-CoV-2 spikprotein, denna anslutning aktiverar S2 -subenheten, vilket hjälper viruset att slå ihop sitt membran med cellens. Därför, spikproteinets funktion liknar att öppna en låst dörr, med S1 som nyckel som låser upp dörren och S2 som kraften som skjuter upp dörren.
Att lära av tidigare epidemier
Den övergripande strukturen för SARS-CoV-2 spikproteinet är mycket lik den för SARS-CoV, ett tidigare coronavirus som orsakade allvarligt akut respiratoriskt syndrom (SARS), och denna likhet hjälpte laget att utveckla sin forskningsstrategi.
S1 -underenheten är i fokus för många läkemedelsutvecklingsstudier, eftersom denna del av spikproteinet har visat sig framkalla ett immunsvar i människokroppen. Dock, tidigare SARS-CoV-studier fann att S1-subenheten upplever höga mutationshastigheter, låta viruset undvika antikroppsbaserade behandlingar samtidigt som det behåller dess förmåga att infektera celler. "Detta är den läxa vi fick från den ursprungliga SARS -epidemin, "sa Minh Phan, en postdoktor vid ORNL och huvudutredare för detta projekt.
Phan och hans kollegor studerar S2 -subenheten eftersom denna komponent i spikproteinet inte muterar lika snabbt. Behandlingar som visar sig framgångsrika för att hämma S2 -funktionen kan förbli effektiva under en längre tid.
En nanoskala av coronaviruset
För att bättre förstå dynamiken hos virala S2 -subenheter och värdcellsmembran, forskarna använder vätskereflektometern (LIQREF) vid ORNL:s Spallation Neutron Source (SNS). Genom att mäta hur neutroner reflekterar i olika vinklar när de passerar genom olika typer av materia, instrumentet kan hjälpa till att belysa strukturen av biologiska material i molekylskala.
Teamet syntetiserade först ett lipidmembran som efterliknar det yttre membranet av celler som kantar ytorna inuti mänskliga lungor, där denna virusinfektion kan äga rum. De identifierade hur lipiderna var organiserade i membranet och hur detta arrangemang förändras när membranen utsätts för olika förhållanden, som temperatur, tryck, och surhet.
Vid LIQREF -instrumentet, forskarna sprider lipidmembranet ovanpå ett tunt lager vatten i en apparat som kallas ett Langmuir -tråg. De introducerar sedan S2 -subenheten till dessa membran för att i detalj observera hur S2- och lipidmembranen ändrar form när de interagerar.
Neutroner är också idealiska för denna studie eftersom de är känsliga för elementet väte, gemensam för alla biologiska molekyler, och dess isotoper. Genom att ersätta några väteatomer med deuteriumatomer, forskare kan skapa kontrast i sina prover och selektivt nolla på olika strukturella egenskaper. Denna teknik är användbar för att studera prover som involverar flera komponenter med liknande densiteter, som lipidmembran.
"Rent generellt, dessa membran är inte enkellipidmembran, "sade John Ankner, an instrument scientist involved with this study. "They consist of lipids of a certain structure, lipids of another structure, cholesterol, proteiner, and things that come in contact with them."
To capture this complexity, the research team is investigating multiple versions of the membrane, changing the contrast of the sample with deuterium each time.

Researchers at ORNL are using neutron scattering at the Spallation Neutron Source to better understand how spike proteins help the COVID-19 virus infect human cells and what drugs could be effective in stopping them. This research team includes John Ankner (left) and Minh Phan (right). Upphovsman:ORNL/Genevieve Martin
"By taking multiple measurements and assembling all of this information together, you can create a single picture of how these different components go together, " said Ankner.
The information derived from these experiments will then help steer the team's efforts in selecting and testing drug candidates that could block this interaction, such as fusion inhibitors that successfully blocked original SARS-CoV infections. If these inhibitors can stop the new coronavirus from invading healthy cells, existing drugs could potentially be repurposed to treat COVID-19 patients. The results may also help guide the design of new fusion inhibitors.
Capturing infection
While other studies have used protein crystallography to better understand the atomic structure of the coronavirus S2 subunit alone, this project is analyzing how S2 changes shape when interacting with a lipid membrane. A shape change could be important for inducing actions within a cell after the spike S1 subunit binds to the cell receptor. Phan also notes that the LIQREF instrument allows the team to measure these dynamics under physiological conditions, whereas protein crystallography only allows researchers to capture what the S2 subunit looks like in a crystallized form.
"At ORNL, we have the right tools to study the dynamics of the interaction under physiological conditions. This allows us to better understand how the S2 subunit moves and changes shape naturally in a wet environment, " said Phan. "Such information could complement what experts already know about the protein from crystallography. If we can help verify what this mechanism looks like, then we may have a clearer understanding to guide the development of drugs that block the fusion process.
Collaboration is key
Självklart, learning more about the S2 subunit and its certain behaviors depends on the ability to grow quality samples, which involves synthesizing S2 subunit proteins, purifying them, and preparing them for experimentation.
Phan and Ankner note that this part of their research has been made possible only through collaboration with labs across ORNL and at outside institutions.
The S2 subunit protein was synthesized in mammalian cell cultures by Steve Foster, a biomedical researcher at the University of Tennessee Medical Center in Knoxville, Tennessee. Through this method, he can develop S2 proteins for research that retain several aspects of its natural structure and function.
"In our lab we routinely use mammalian cell cultures for protein production, so we hope we've produced an S2 protein best suited for this research analysis. Our proximity to ORNL also works well in that the sample doesn't have to travel far, meaning less risk of damaging the protein or distorting its original structure, which is critical for this work, " said Foster.
Following its synthesis, the sample was purified by Jessy Labbé and Michael Melesse Vergara from ORNL's Biosciences Division. Scientists from the ORNL Neutron Sciences Directorate then performed a series of tests to confirm the structure of the sample protein and check its purity. This effort was implemented by Yichong Fan and Wellington Leite from the Bio-Labs team, and Jacob Kinnun and Mary Odom from the SNS team.
"We put an enormous effort into making sure the protein has the right properties going into the experiment. If it does not, we could get spurious results and misinterpret what we're doing, " said Hugh O'Neill, director of ORNL's Center for Structural and Molecular Biology and lead researcher for the Bio-Labs team.
"This virus is extremely delicate in its components, and it's a big challenge to get these materials to the neutron instrument, " said Ankner. "That's why involving various ORNL labs and the University of Tennessee is so crucial. Each step that eventually gets the sample onto our instrument requires the expertise of lots of people."
This project also relied on efforts from the LIQREF instrument staff, who were instrumental in developing the systems, protocols, and modeling frameworks necessary to run the experiments and interpret the data.
"Experts across the division, across ORNL, and from partner institutes have come together for this project, " said Phan. "We couldn't have done this without their support, and it's greater motivation to fulfill our mission."