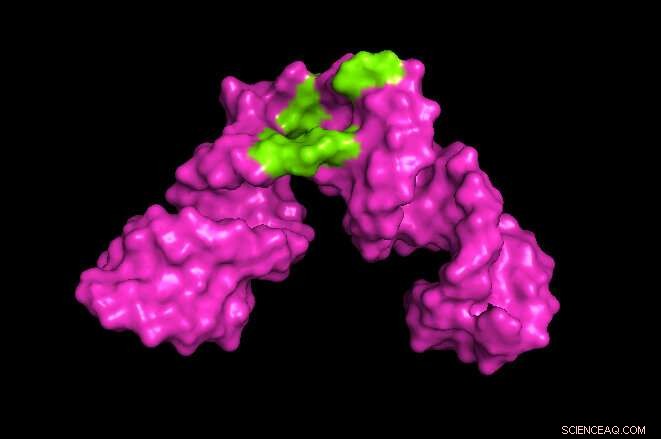
University of Maryland forskare utvecklade en metod för att utöka omfattningen av kärnmagnetisk resonans (NMR) spektroskopi. I exemplet ovan, forskarna kunde skapa en 3D-bild som avslöjar platsen på en bit av hepatit-RNA där små molekyler som ett läkemedel kunde binda (visas i grönt). Kredit:Kwaku Dayie/University of Maryland
Forskare från University of Maryland har utvecklat en metod för att bestämma strukturerna för stora RNA-molekyler i hög upplösning. Metoden övervinner en utmaning som har begränsat 3D-analys och avbildning av RNA till endast små molekyler och bitar av RNA under de senaste 50 åren.
Den nya metoden, som utökar omfattningen av kärnmagnetisk resonans (NMR) spektroskopi, kommer att göra det möjligt för forskare att förstå formen och strukturen hos RNA-molekyler och lära sig hur de interagerar med andra molekyler. De insikter som denna teknologi ger kan leda till riktade RNA-terapeutiska behandlingar för sjukdomar. Forskningsdokumentet om detta arbete publicerades i tidskriften Vetenskapens framsteg den 7 oktober, 2020.
"Fältet för kärnmagnetisk resonansspektroskopi har fastnat och tittat på saker som är små, säg 35 RNA-byggstenar eller nukleotider. Men de flesta intressanta saker som är biologiskt och medicinskt relevanta är mycket större, 100 nukleotider eller mer, " sa Kwaku Dayie, en professor i kemi och biokemi vid UMD och senior författare till uppsatsen. "Så, att kunna bryta ner stocksylten och titta på saker som är stora är väldigt spännande. Det kommer att tillåta oss att kika in i dessa molekyler och se vad som händer på ett sätt som vi inte har kunnat göra tidigare."
I NMR-spektroskopi, forskare riktar radiovågor mot en molekyl, excitera atomerna och "lyser upp" molekylen. Genom att mäta förändringar i magnetfältet runt de exciterade atomerna – kärnmagnetisk resonans – kan forskare rekonstruera egenskaper som formen, molekylens struktur och rörelse. Data som detta producerar kan sedan användas för att generera bilder, ungefär som MRI-bilder som ses inom medicin.
Vanligtvis, NMR-signaler från de många atomerna i en biologisk molekyl som RNA överlappar varandra, gör analysen mycket svår. Dock, på 1970-talet, forskare lärde sig att biokemiskt konstruera RNA-molekyler för att fungera bättre med NMR genom att ersätta väteatomerna med magnetiskt aktiva fluoratomer. I relativt små RNA-molekyler som består av 35 eller färre nukleotider, fluoratomerna lyser lätt upp när de träffas av radiovågor och förblir exciterade tillräckligt länge för högupplöst analys. Men när RNA-molekylerna blir större, fluoratomerna lyser bara kort, tappar sedan snabbt sin signal. Detta har förhindrat högupplöst 3D-analys av större RNA-molekyler.
Tidigare arbete av andra hade visat att fluor fortsatte att producera en stark signal när det var bredvid en kolatom som innehåller sex protoner och sju neutroner (C-13). Så, Dayie och hans team utvecklade en relativt enkel metod för att ändra den naturligt förekommande C-12 i RNA (som har 6 protoner och 6 neutroner) till C-13 och installera en fluoratom (F-19) direkt bredvid den.
Dayie och hans team visade först att deras metod kunde producera data och bilder som är lika med nuvarande metoder genom att applicera den på bitar av RNA från HIV som innehåller 30 nukleotider, som tidigare hade avbildats. De tillämpade sedan sin metod på bitar av hepatit B-RNA innehållande 61 nukleotider - nästan dubbelt så stor som tidigare NMR-spektroskopi som är möjlig för RNA.
Deras metod gjorde det möjligt för forskarna att identifiera platser på hepatit B-RNA:t där små molekyler binder och interagerar med RNA:t. Det kan vara användbart för att förstå effekten av potentiella terapeutiska läkemedel. Nästa steg för forskarna är att analysera ännu större RNA-molekyler.
"Detta arbete tillåter oss att utöka det som kan sättas i fokus, " sa Dayie. "Våra beräkningar säger oss att, i teorin, vi kan titta på riktigt stora saker, som en del av ribosomen, som är den molekylära maskinen som syntetiserar proteiner inuti celler."
Genom att förstå formen och strukturen hos en molekyl, forskare kan bättre förstå dess funktion och hur den interagerar med sin omgivning. Vad mer, denna teknik kommer att göra det möjligt för forskare att se 3D-strukturen när den förändras, eftersom särskilt RNA-molekyler ändrar form ofta. Denna kunskap är nyckeln till att utveckla läkemedel som snävt riktar sig mot sjukdomsspecifika molekyler utan att påverka friska cellfunktioner.
"Förhoppningen är att om forskarna känner till skrymslen och vråren i en molekyl som är dysfunktionell, sedan kan de designa droger som fyller skrymslen och vråren för att ta det ur drift, ", sa Dayie. "Och om vi kan följa dessa molekyler när de ändrar form och struktur, då kommer deras svar på potentiella droger att vara lite mer förutsägbart, och att designa läkemedel som är effektiva kan vara mer effektivt."