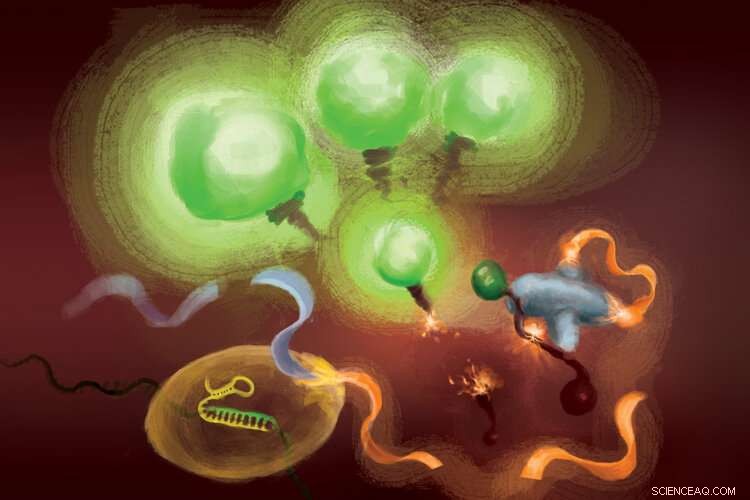
Reaktionerna involverade i FIND-IT-analysen för att upptäcka infektion med SARS-CoV-2-viruset. När Cas13-enzymet (nederst till vänster) binder till sitt mål-RNA, den klipper av en molekyl (orange och ljusblått band) för att frigöra en aktivator (orange) som överladdar Csm6-nukleaset (höger) för att klyva och frigöra fluorescerande molekyler som lyser upp (grönt) och signalerar närvaron av viralt RNA. Kredit:Margaret L. Liu, University of Chicago Pritzker School of Medicine.
Frekvent, snabba tester för covid-19 är avgörande för att kontrollera spridningen av utbrott, speciellt som ny, fler överföringsbara varianter dyker upp.
Medan dagens gyllene standard COVID-19 diagnostiska test, som använder qRT-PCR - kvantitativ omvänd transkriptas-polymeraskedjereaktion (PCR) - är extremt känslig, detektera ner till en kopia av RNA per mikroliter, det kräver specialutrustning, en körtid på flera timmar och en centraliserad laboratorieanläggning. Som ett resultat, testning tar vanligtvis minst en till två dagar.
En forskargrupp ledd av forskare i Jennifer Doudnas labb, David Savage och Patrick Hsu vid University of California, Berkeley, syftar till att utveckla ett diagnostiskt test som är mycket snabbare och lättare att distribuera än qRT-PCR. Den har nu kombinerat två olika typer av CRISPR-enzymer för att skapa en analys som kan detektera små mängder viralt RNA på mindre än en timme. Doudna delade 2020 Nobelpriset i kemi för uppfinningen av CRISPR-Cas9 genomredigering.
Även om den nya tekniken ännu inte är i det skede där den konkurrerar med känsligheten hos qRT-PCR, som bara kan upptäcka några få kopior av viruset per mikroliter vätska, den kan redan ta upp nivåer av viralt RNA – cirka 30 kopior per mikroliter – tillräckligt för att användas för att övervaka befolkningen och begränsa spridningen av infektioner.
"Du behöver inte PCR-känsligheten för att i princip fånga och diagnostisera covid-19 i samhället, om testet är tillräckligt bekvämt och tillräckligt snabbt, " sa medförfattaren David Savage, professor i molekylär- och cellbiologi. "Vår förhoppning var att driva biokemin så långt som möjligt till den punkt där du kunde föreställa dig ett mycket bekvämt format i en miljö där du kan testas varje dag, säga, vid ingången till jobbet."
Forskarna kommer att rapportera sina resultat online den 5 augusti i tidskriften Naturens kemiska biologi .
Flera CRISPR-baserade analyser har godkänts för akut användning av Food and Drug Administration, men alla kräver ett första steg där det virala RNA:t amplifieras så att detektionssignalen – som involverar frisättning av en fluorescerande molekyl som lyser under blått ljus – är tillräckligt ljus för att se. Även om denna initiala amplifiering ökar testets känslighet till en liknande nivå som qRT-PCR, den introducerar också steg som gör testet svårare att utföra utanför ett laboratorium.
Det UC Berkeley-ledda teamet försökte nå en användbar känslighet och hastighet utan att offra analysens enkelhet.
"För vårdtillämpningar, du vill ha ett snabbt svar så att folk snabbt kan veta om de är smittade eller inte, innan du sätter dig på ett flyg, till exempel, eller besök släktingar, " sa teamledaren Tina Liu, en forskare i Doudnas labb vid Innovative Genomics Institute (IGI), ett CRISPR-fokuserat center som involverar UC Berkeley och UC San Francisco forskare.
Förutom att ha ett extra steg, en annan nackdel med initial förstärkning är att, eftersom det gör miljarder kopior av viralt RNA, det finns en större chans för korskontaminering över patientprover. Den nya tekniken som utvecklats av teamet vänder på detta och ökar istället den fluorescerande signalen, eliminera en viktig källa till korskontaminering.
Den förstärkningsfria tekniken, som de kallar Fast Integrated Nuclease Detection In Tandem (FIND-IT), skulle kunna möjliggöra snabba och billiga diagnostiska tester för många andra infektionssjukdomar.
"Medan vi startade det här projektet i det uttryckliga syftet att påverka COVID-19, Jag tror att denna speciella teknik kan vara tillämpbar på mer än bara denna pandemi eftersom, i sista hand, CRISPR är programmerbar, " sa Liu. "Så, du kan ladda CRISPR-enzymet med en sekvens riktad mot influensavirus eller HIV-virus eller någon typ av RNA-virus, och systemet har potential att fungera på samma sätt. Den här uppsatsen slår verkligen fast att denna biokemi är ett enklare sätt att detektera RNA och har förmågan att detektera det RNA i en känslig och snabb tidsram som kan vara tillgänglig för framtida tillämpningar inom vårddiagnostik."
Forskarna håller för närvarande på att bygga en sådan diagnostik med hjälp av FIND-IT, vilket skulle innefatta steg för att samla in och bearbeta prover och att köra analysen på en kompakt mikrofluidisk enhet.
Använder tandem Cas-proteiner
För att ta bort målförstärkning från ekvationen, teamet använde ett CRISPR-enzym – Cas13 – för att först detektera virus-RNA, och en annan typ av Cas-protein, kallas Csm6, för att förstärka fluorescenssignalen.
Cas13 är en sax för allmänt bruk för att klippa RNA; när den väl binder till sin målsekvens, specificeras av ett guide-RNA, den är förberedd för att skära ett brett spektrum av andra RNA-molekyler. Denna målutlösta skärningsaktivitet kan utnyttjas för att koppla detektion av en specifik RNA-sekvens till frisättning av en fluorescerande reportermolekyl. Dock, på egen hand, Cas13 kan kräva timmar för att generera en detekterbar signal när mycket låga mängder mål-RNA är närvarande.
Lius insikt var att använda Csm6 för att förstärka effekten av Cas13. Csm6 är ett CRISPR-enzym som känner av närvaron av små ringar av RNA och aktiveras för att skära ett brett spektrum av RNA-molekyler i celler.
För att öka Cas13-detektion, hon och hennes kollegor designade en speciellt konstruerad aktivatormolekyl som skärs när Cas13 upptäcker viralt RNA. Ett fragment av denna molekyl kan binda till och utlösa Csm6 att skära och frigöra en ljus fluorescerande molekyl från en bit RNA. I vanliga fall, aktivatormolekylen bryts snabbt ned av Csm6, vilket begränsar mängden fluorescerande signal den kan generera. Liu och hennes kollegor utarbetade ett sätt att kemiskt modifiera aktivatorn så att den skyddas från nedbrytning och kan överladda Csm6 för att upprepade gånger skära och frigöra fluorescerande molekyler kopplade till RNA. Detta resulterar i en känslighet som är 100 gånger bättre än den ursprungliga aktivatorn.
"När Cas13 aktiveras, det klyver denna lilla aktivator, ta bort ett segment som skyddar det, " sa Liu. "Nu när det är befriat, det kan aktivera många olika molekyler av det andra enzymet, Csm6. Och så, ett mål som känns igen av Cas13 leder inte bara till aktivering av sin egen RNA-skärningsförmåga; det leder till genereringen av många fler aktiva enzymer som var och en sedan kan klyva ännu fler fluorescerande reportrar."
Teamet av forskare inkorporerade också en optimerad kombination av guide-RNA som möjliggör mer känslig igenkänning av det virala RNA:t av Cas13. När detta kombinerades med Csm6 och dess aktivator, teamet kunde detektera ner till 31 kopior per mikroliter av SARS-CoV-2 RNA på så lite som 20 minuter.
Forskarna lade också extraherat RNA från patientprover till FIND-IT-analysen i en mikrofluidisk patron, för att se om denna analys kan anpassas för att köras på en bärbar enhet. Använda en liten enhet med en kamera, de kunde upptäcka SARS-CoV-2-RNA extraherat från patientprover med en känslighet som skulle fånga COVID-19-infektioner på sin topp.
"Denna tandemnukleasmetod – Cas13 plus Csm6 – kombinerar allt till en enda reaktion vid en enda temperatur, 37 grader Celsius, så det kräver inte högtemperaturuppvärmning eller flera steg, som är nödvändigt för andra diagnostiska tekniker, " sa Liu. "Jag tror att det här öppnar möjligheter för snabbare, enklare tester som kan nå en känslighet som är jämförbar med andra nuvarande tekniker och som potentiellt kan nå ännu högre känsligheter i framtiden."
Utvecklingen av denna amplifieringsfria metod för RNA-detektion resulterade från en omorientering av forskningen inom IGI när pandemin började mot problem med diagnos och behandling av covid-19. I sista hand, fem labb vid UC Berkeley och två labb vid UCSF blev involverade i detta forskningsprojekt, en av många inom IGI.
"När vi började med det här, vi hade förhoppningar om att skapa något som nådde paritet med PCR, men krävde ingen förstärkning – det skulle vara drömmen, sa Savage, som var huvudutredare för projektet. "Och ur ett känslighetsperspektiv, vi hade ungefär ett tiotusenfaldigt gap att hoppa. Vi har gjort det ungefär tusen gånger; vi har kört ner det ungefär tre storleksordningar. Så, Vi är nästan där. Sista april, när vi verkligen började kartlägga det, det verkade nästan omöjligt."