Kredit:University of Oregon
Forskare har vetat i decennier att en viss klass av enzymer är en viktig aktör inom cellbiologi eftersom de ofta muterar och blir stora drivkrafter för cancer.
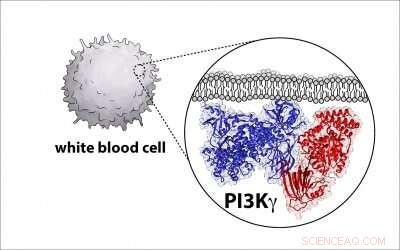
Biopharmaföretag försöker utveckla läkemedel som riktar sig mot och inaktiverar dessa enzymer, känd som fosfoinositid 3-kinas, eller PI3K för kort, på grund av deras roll i att orsaka cancer hos människor. Men för att göra det, forskare behöver en detaljerad ritning av enzymarkitekturen, och UO biokemiprofessor Scott Hansen är en del av en grupp som avslöjar det diagrammet.
Hansen, en biträdande professor vid UO:s institution för kemi och biokemi, nämnda del av utmaningen är att molekylerna är komplexa, med två stora proteiner som går samman och bildar ett komplex.
"Så det är riktigt utmanande att ta reda på vad som är strukturen och organisationen av detta protein, ”, sade han. ”En förutsättning för att utveckla ett läkemedel är att lära sig hur läkemedlet binder till molekylen. Du behöver en ritning av proteinet att docka läkemedlet på."
Nu har Hansen och kollegor från flera andra universitet utvecklat en sådan ritning för PI3K och identifierat var ett kritiskt regulatoriskt protein binder till enzymet, ett fynd med potential att hjälpa till att öka ansträngningen att bygga mer specifika cancerläkemedel.
"Detta kommer definitivt att forma hur människor tänker om att utveckla cancerterapier, " han sa.
Hansen är en av 13 medförfattare på den tidning som publicerats i tidskriften Vetenskapens framsteg , tillsammans med kollegor vid universiteten i Victoria, British Columbia, Washington och Genève, och Vrije Universiteit Brussel i Belgien.
Det som gör uppsatsen unik är "strukturell information med högre upplösning och att kunna mäta biologiskt relevanta interaktioner med andra molekyler för att definiera sekvensen och prioriteringen av interaktioner, sa Hansen.
Men han sa att mycket arbete återstår för att bättre förstå hur andra molekyler binder till proteinet och modulerar dess funktion.
För att lära sig enzymets struktur, forskarna använde en teknik som kallas kryo-elektronmikroskopi, som samlar tiotusentals bilder av enstaka P13K-enzymer i ett ultratunt lager is med hjälp av ett elektronmikroskop. Bilderna kompileras och medelvärdesbildas för att skapa en enda högupplöst bild som avslöjar enzymets strukturella egenskaper.
I detta fall, enzymets struktur var annorlunda än man tidigare trott. Den nya ritningen kommer att tillåta forskare att definiera hur andra molekyler aktiverar proteinet.
"Många gånger forskar vi och vi får ledtrådar om hur proteiner är organiserade eller så tittar vi på strukturer av relaterade molekyler och extrapolerar, men ibland är dessa antaganden felaktiga och kan leda ett fält på fel väg, "Sade Hansen. "Att ha en högre upplösningsritning av organisationen av molekyler tillåter människor att utveckla läkemedel som är specifika för endast den molekylen."
Hansen sa att forskare som är involverade i tidningen har ägnat 10 år åt att arbeta med problemet.
"Det är inte ovanligt för riktigt komplicerade proteiner, " han sa.
Hansen sa att hans labb vid UO och John Burkes labb vid University of British Columbia bedriver kompletterande forskning. Burkes labb använder kryo-elektronmikroskopi för att få högupplösta bilder av molekyler, och Hansens labb kan "göra mer dynamiska åtgärder som gör att vi kan mäta interaktionerna mellan detta enzym och andra faktorer som är avgörande för aktivering."
Han sa att det är spännande att samarbeta med andra forskare som har expertis inom områden som han inte har.
"Mer och mer, för att vi ska kunna uppnå våra forskningsambitioner kan vi inte bara lita på kompetensen i vårt eget labb, ", sa han. "Problemen blir för komplicerade och man måste närma sig dem från olika vinklar och integrera en massa olika expertis."