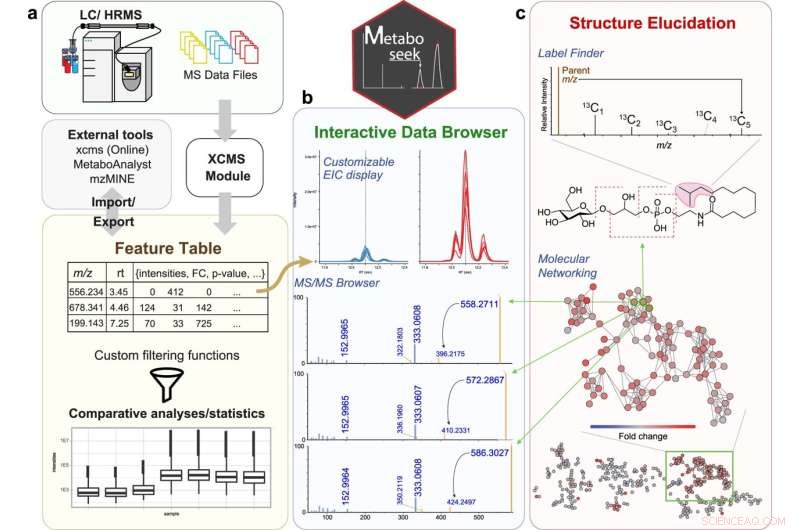
Jämförande metabolomik med Metaboseek. en Metaboseek inkluderar en integrerad XCMS-modul för funktionsdetektering och funktionsgruppering (med CAMERA-kommentarer) och accepterar funktionstabeller genererade av annan programvara. b Funktioner kan kommenteras och prioriteras med hjälp av omfattande filtreringsalternativ och integrerade statistikverktyg. Rådata för varje molekylär funktion kan bläddras snabbt, inklusive tillhörande EIC, MS1 och MS/MS-spektra. c Datawebbläsaren interagerar med en uppsättning verktyg för att klargöra strukturer, t.ex. SIRIUS-baserad molekylformel och strukturförutsägelse, Label Finder för att identifiera isotopmärkta föreningar och MS/MS-mönstersökaren för att identifiera MS-funktioner med karakteristiska fragmenteringsmönster. Kredit:Nature Communications (2022). DOI:10.1038/s41467-022-28391-9
Som postdoktoral forskarassistent i labbet hos BTI-fakultetsmedlemmen Frank Schroeder, såg Max Helf sina labbkamrater ständigt kämpa när de analyserade data. Så han bestämde sig för att göra något åt det och utvecklade en gratis app med öppen källkod som heter Metaboseek, som nu är avgörande för labbets arbete.
Schroeder-labbet studerar rundmasken Caenorhabditis elegans, ett av de mest framgångsrika modellsystemen för mänsklig biologi, för att upptäcka nya metaboliter som styr evolutionärt bevarade signalvägar och som kan vara användbara som ledar för utvecklingen av nya läkemedel eller jordbrukskemikalier. Forskarna utför denna uppgift genom att jämföra metaboliterna mellan två olika maskpopulationer – en process som kallas jämförande metabolomik.
Med tanke på att prover rutinmässigt har mer än 100 000 föreningar i sig, är beräkningsmetoder viktiga för att utföra analysen.
Teamet hade förlitat sig på mjukvarupaket som inte erbjöd den flexibilitet som krävs för att enkelt kunna anpassa analysparametrar. Den begränsningen, och avsaknaden av ett lämpligt grafiskt användargränssnitt, innebar att Helfs kollegor stod inför den besvärliga uppgiften att visuellt inspektera högar av data - till exempel för att upptäcka möjliga falska positiva - och hoppa mellan flera andra programvaruverktyg för att bekräfta och filtrera bort de meningslösa resultat.
"Det verkade bara väldigt ineffektivt för mig, och jag kunde inte komma över bristerna med andra mjukvarulösningar för det här problemet," sa Helf. "Jag trodde att det måste finnas ett enklare sätt, så jag började skriva kod för min egen programvara."
Helf utvecklade den första versionen av sin mjukvara 2017 och fortsatte att förbättra den under de kommande två åren. "Förutom att ta itu med problemen som mina labbkamrater redan stod inför, pratade jag med dem om vad som höll dem tillbaka - vad de ville göra men inte ens försökte - och byggde de funktionerna i appen", säger Helf, som nu är en bioinformatik produktchef på proteomikföretaget Biognosys AG. "Jag ville att det här nya verktyget skulle vara användarvänligt och tillgängligt för alla som arbetar med kemisk biologi."
Resultatet blev Metaboseek, en app med ett grafiskt gränssnitt som innehåller flera dataanalysverktyg som icke-kodande forskare annars inte skulle ha. Appen effektiviserar analysen av jämförande metabolomikdata genom att hjälpa forskaren att avgöra vilka datafunktioner som är verkliga och låta dem gräva djupare i dessa funktioner – allt inom samma verktyg.
"Max gjorde det här utan att jag ens bad om det," sa Schroeder. "Innan jag visste att det här hände fanns Metaboseek. Vi började använda det, och nu kunde vårt labb och många medarbetare inte existera utan det."
I en studie publicerad i Nature Communications den 10 februari gav Schroeders team proof-of-concept för Metaboseek genom att tillämpa det på en viktig fettmetabolismväg som ännu inte hade studerats:α-oxidationsvägen i C. elegans som hjälper till att bryta ner en klass av fettsyror.
Med hjälp av Metaboseek fann teamet att rundmaskar som saknar en nyckelgen i α-oxidationsvägen ackumulerade hundratals tidigare orapporterade metaboliter. Fynden är viktiga eftersom α-oxidation är en grundläggande biokemisk väg i maskar som är bevarad hos människor, sa Schroeder.
"Bennett Fox gjorde kemiarbetet, så den här studien var ett trevligt samarbete mellan de två postdoktorerna," tillade Schroeder, som också är professor vid Cornell Universitys institution för kemi och kemisk biologi.
Enligt Schroeder och Helf finns det några anledningar till varför det inte finns många bra analysverktyg för att jämföra metabolomikdata. För det första är jämförande metabolomik ett relativt ungt område jämfört med andra datatunga områden inom biologi som genomik (som fokuserar på DNA) och proteomik (som fokuserar på proteiner), så det har inte funnits tillräckligt med tid för att utveckla mjukvaruverktyg och databasinfrastruktur .
Dessutom, under det senaste decenniet, har tillkomsten av prisvärda, ultrahögupplösta masspektrometrar för insamling av metabolomikdata ökat med kanske mer än tio gånger mängden data som ett prov kan generera – vilket skapar ett ännu större behov av sofistikerade verktyg som kan behålla upp med floden av data.
Metaboseek möter dessa behov med en rad funktioner för att analysera olika typer av data för att underlätta identifiering av föreningar, strukturbestämning, tilldelning av metaboliter till familjer baserat på strukturella likheter, spårning av radiomärkta föreningar och mer. + Utforska vidare