Ett team av forskare har nyligen visat nyttan av att använda ett tidigare etablerat screeningsystem för att undersöka interaktionerna mellan små molekyler och RNA. Deras studie är publicerad i Communications Chemistry .
Teamet leddes av professor Hirohide Saito (Department of Life Science Frontiers, CiRA), Dr. Kaoru R. Komatsu (fd doktorand i CiRA), docent Kazumitsu Onizuka och professor Fumi Nagatsugi (Institutet för multidisciplinär forskning). för avancerade material, Tohoku University).
Från de senaste SARS-CoV-2 mRNA-vaccinerna för att bekämpa covid-19-pandemin till risdiplam, en RNA-skarvningsmodifierare som godkänts av US Food and Drug Administration för spinal muskelatrofi, har ordet "RNA" kommit in i ett vanligt språk eftersom det representerar både nya klasser av terapeutiska medel och läkemedelsmål.
Men vår förståelse av hur olika RNA-sekvenser och i sin tur strukturer dikterar interaktioner med små molekyler som läkemedelsföreningar eller stora biomolekyler som proteiner är fortfarande ofullständig.
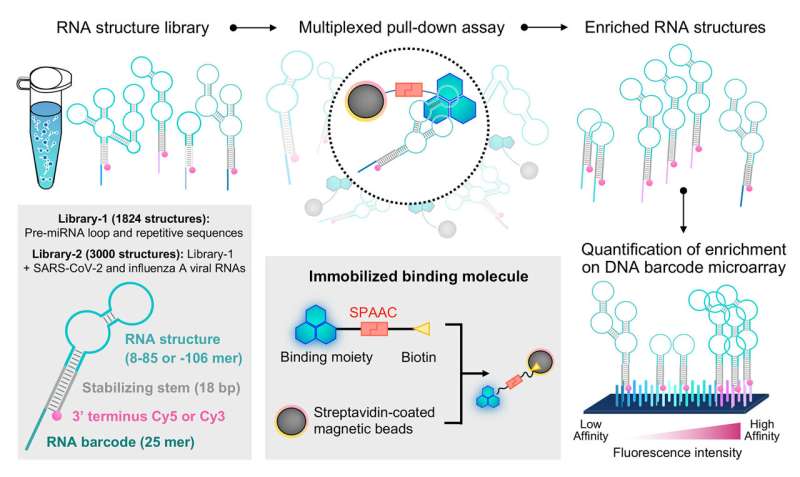
I en tidigare studie designade professor Saito och hans forskargrupp ett system som kallas folded RNA element profilering with structure library, eller FOREST, för att undersöka de molekylära detaljerna om hur RNA interagerar med kända RNA-bindande proteiner.
För denna nya studie, genom ett samarbete med forskare från Tohoku University, illustrerade det gemensamma forskarteamet hur FOREST kan användas för att analysera små molekylära interaktioner med RNA.
Forskargruppen validerade först tillämpbarheten av FOREST-metoden på små molekyler genom att undersöka hur ett RNA-strukturbibliotek interagerar med kända småmolekylära RNA-interaktorer:G-clamp och tiazinorange (TO) derivat.
Forskarna isolerade RNA-strukturer bundna till G-clamp från ett bibliotek som omfattar mer än 1 800 RNA-sekvenser härrörande från mänskliga pre-miRNA och andra repetitiva och kontrollsekvenser. Eftersom dessa RNA-strukturer är kopplade till ett fluorescerande färgämne och RNA-streckkod, kan de lätt avkodas och kvantifieras av en DNA-mikroarray med sekvenser som är komplementära till RNA-streckkoderna för att ge en kvantitativ analys av hur RNA-strukturer interagerar med den specifika lilla molekylen av intresse.
Som förväntat identifierade de preferentiell bindning till guanosin (G)-innehållande enkel- och dubbelsträngade RNA-sekvenser (ssRNA respektive dsRNA) genom G-clamp. Från RNA-strukturbiblioteket valde forskargruppen sekvenser som visar hög-, mellan- eller lågaffinitetsbindning till G-clamp för validering av ett oberoende fluorescensbaserat experiment som direkt mäter skenbara dissociationskonstanter för individuella interaktioner.
Anmärkningsvärt nog observerade de en bra korrelation mellan den relativa bindningsaffiniteten uppskattad av FOREST-metoden och de skenbara dissociationskonstanter som bestäms av den fluorescensbaserade bindningsanalysen, vilket indikerar den höga robustheten hos denna metod för att kvantifiera interaktioner mellan små molekyler och RNA.
Dessutom, genom att mutera en specifik RNA-loopstruktur med flera guanosiner på olika platser, upptäckte de att G-clamp inte interagerar lika med alla guanosiner på slingan, men att ytterligare strukturell kontext kan påverka interaktionen.
Omvänt är TO-derivat ofta använda prober för fluorescerande indikatorförskjutningsanalyser (FID). Forskarna blandade sedan TO och TO-3 separat med ett utökat RNA-strukturbibliotek innehållande ytterligare sekvenser härledda från SARS-CoV-2 och influensa A-virus-RNA för att bättre karakterisera TO-derivat för RNA-mätningar.
Som väntat, medan det inte fanns någon korrelation mellan bindningsprofiler för RNA-strukturer som interagerar med G-clamp och TO-derivat, delade TO och TO-3 liknande bindningsprofiler med några mindre skillnader.
Ytterligare jämförelser mellan TO-N3 , TO-N3 -2 och TO-3-N3 avslöjade att länkpositionen har ett blygsamt inflytande över RNA-bindningsprofiler. Dessutom, baserat på dessa bindningsprofiler, urskiljde forskarna vissa bas- och looppositionspreferenser som TO-derivat har när de interagerar med RNA-strukturer.
Forskargruppen utökade dessutom sin analys av TO-derivat genom att jämföra relativa bindningsaffiniteter som bestämts av FOREST med de uppenbara dissociationskonstanterna uppmätta för kommersiellt tillgängliga fluorescerande nukleinindikatorer, TO-PRO-1 och TO-PRO-3, med den fluorescensbaserade bindningsanalysen .
Genom denna analys avslöjade de att medan TO-N3 -2 kan mer exakt återge bindningsprofilen för TO-PRO1 jämfört med TO-3-N3 , båda TO-N3 -2 och TO-3-N3 simulera TO-PRO-3 ungefär lika bra, vilket ger avgörande strukturella insikter för att förbättra parningar av mål-RNA och fluorescerande indikatorer för FID-analyser.
Med hjälp av de bindningsprofiler som bestämts för TO-derivat, valde forskargruppen kombinationer av fluorescerande indikatorer (TO-PRO-1 eller TO-PRO-3) och pre-miRNA-sekvenser som tidigare visat sig vara dysreglerade i tumörer med mellanliggande bindningsaffiniteter för FID-analyser.
Som ett proof-of-concept screenade forskarna ett kommersiellt tillgängligt kemiskt bibliotek med 118 föreningar för att identifiera små molekyler som kan interagera med sjukdomsassocierade pre-miRNA. Genom detta försök identifierade de baicalein (Bai), myricetin (Myr), chelerytrinklorid (Che) och AS 602801 (AS) som kandidatföreningar. Medan Myr och Che är kända för att binda DNA och RNA, var detta den första demonstrationen av AS som en nukleinsyrainteraktör.
Noterbart observerade forskarna olika resultat när TO-PRO-1 eller TO-PRO-3 användes som fluorescerande indikator, vilket tyder på att distinkta indikatorer bör användas för att undvika falska positiva och negativa identifieringar. Ytterligare undersökning av AS bekräftade bindning till flera mänskliga pre-miRNA av intresse, men forskarna märkte också att föreningen uppvisade starka ljusegenskaper när de interagerar med RNA.
Strukturell undersökning av föreningen tyder på att den innehåller en kemisk del som troligen är ansvarig för ljusegenskaperna, vilket gör den till en förening av intresse för vidare utveckling till en ny RNA-interaktor och fluorescerande sond.
I denna studie illustrerar den gemensamma forskningssatsningen återigen användbarheten av FOREST-metoden, inte bara för att inspektera RNA-proteininteraktioner utan också för att undersöka de fina detaljerna i interaktioner mellan RNA och små molekyler.
Med tanke på den enorma potentialen för RNA som ett nytt terapeutiskt tillvägagångssätt inom nästa generations medicin, kommer förmågan att systemiskt karakterisera små molekyl-RNA-interaktioner i stor skala ha enorma effekter på grundläggande RNA-forskning och översättningen av den kunskapen till terapier.
Mer information: Ryosuke Nagasawa et al, storskalig analys av småmolekyl-RNA-interaktioner med hjälp av multiplexerade RNA-strukturbibliotek, Communications Chemistry (2024). DOI:10.1038/s42004-024-01181-8
Journalinformation: Kommunikationskemi
Tillhandahålls av Kyoto University