Medan E. coli är en av de mest studerade organismerna, har funktionen hos 30 % av proteinerna som utgör E. coli ännu inte avslöjats tydligt. För detta användes en artificiell intelligens för att upptäcka 464 typer av enzymer från proteinerna som var okända, och forskarna fortsatte med att verifiera förutsägelserna om tre typer av proteiner som framgångsrikt identifierades genom in vitro enzymanalys.
Ett gemensamt forskarlag, inklusive Gi Bae Kim, Ji Yeon Kim, Dr Jong An Lee och den framstående professorn Sang Yup Lee från Institutionen för kemi- och biomolekylär teknik vid KAIST, och Dr Charles J. Norsigian och professor Bernhard O. Palsson från Institutionen för bioteknik vid UCSD har utvecklat DeepECtransformer, en artificiell intelligens som kan förutsäga enzymfunktionerna utifrån proteinsekvensen. Dessutom har teamet etablerat ett prediktionssystem genom att använda AI för att snabbt och exakt identifiera enzymfunktionen.
Teamets arbete beskrivs i artikeln med titeln "Funktionell annotering av enzymkodande gener med djupinlärning med transformatorlager." Tidningen publicerades den 14 november i Nature Communications .
Enzymer är proteiner som katalyserar biologiska reaktioner, och identifiering av varje enzyms funktion är avgörande för att förstå de olika kemiska reaktionerna som finns i levande organismer och de metaboliska egenskaperna hos dessa organismer.
Enzyme Commission (EC) nummer är ett enzymfunktionsklassificeringssystem designat av International Union of Biochemistry and Molecular Biology, och för att förstå de metaboliska egenskaperna hos olika organismer är det nödvändigt att utveckla en teknologi som snabbt kan analysera enzymer och EC-nummer av enzymerna som finns i genomet.
Olika metoder baserade på djupinlärning har utvecklats för att analysera egenskaperna hos biologiska sekvenser, inklusive förutsägelse av proteinfunktion, men de flesta av dem har ett problem med en svart låda, där slutledningsprocessen för AI inte kan tolkas.
Olika prediktionssystem som använder AI för förutsägelse av enzymfunktioner har också rapporterats, men de löser inte detta problem med svarta lådan, eller kan inte tolka resonemangsprocessen på en finkornig nivå (t.ex. nivån av aminosyrarester i enzymsekvensen ).
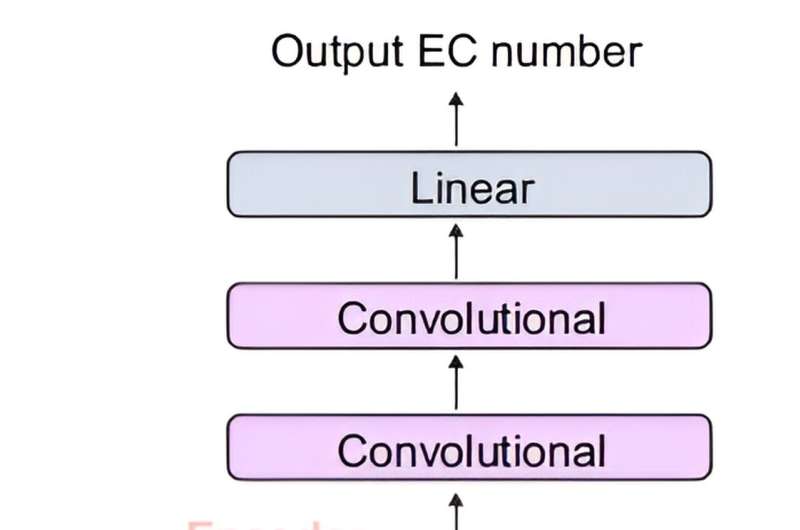
Det gemensamma teamet utvecklade DeepECtransformer, en AI som använder djupinlärning och en analysmodul för proteinhomologi för att förutsäga enzymfunktionen hos en given proteinsekvens.
För att bättre förstå egenskaperna hos proteinsekvenser användes transformatorarkitekturen, som vanligtvis används i naturlig språkbehandling, dessutom för att extrahera viktiga egenskaper om enzymfunktioner i sammanhanget av hela proteinsekvensen, vilket gjorde det möjligt för teamet att exakt förutsäga EC enzymets nummer. Den utvecklade DeepECtransformer kan förutsäga totalt 5360 EC-nummer.
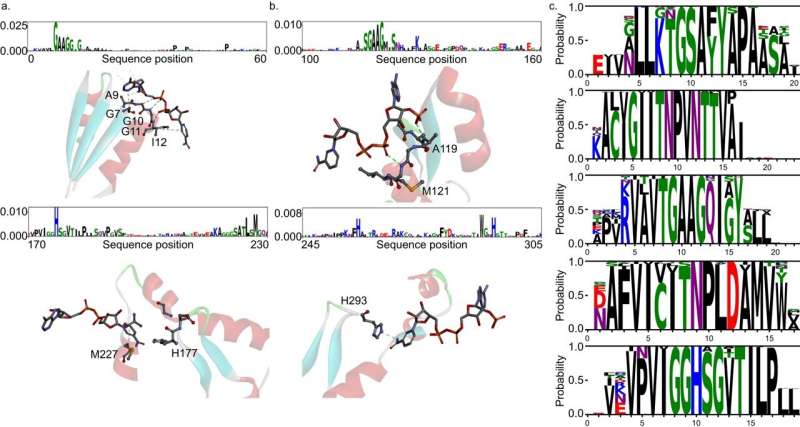
Det gemensamma teamet analyserade vidare transformatorarkitekturen för att förstå slutledningsprocessen för DeepECtransformer och fann att i slutledningsprocessen använder AI information om katalytiska aktiva platser och/eller kofaktorbindningsställen som är viktiga för enzymfunktionen. Genom att analysera den svarta lådan av DeepECtransformer, bekräftades det att AI kunde identifiera de funktioner som är viktiga för enzymfunktionen på egen hand under inlärningsprocessen.
"Genom att använda det förutsägelsesystem vi utvecklade kunde vi förutsäga funktionerna hos enzymer som ännu inte hade identifierats och verifiera dem experimentellt", säger Gi Bae Kim, uppsatsens första författare.
"Genom att använda DeepECtransformer för att identifiera tidigare okända enzymer i levande organismer, kommer vi att kunna analysera olika aspekter som är involverade i organismers metaboliska processer mer exakt, såsom enzymerna som behövs för att biosyntetisera olika användbara föreningar eller enzymerna som behövs för att biologiskt bryta ned plast." tillade han.
"DeepECtransformer, som snabbt och exakt förutsäger enzymfunktioner, är en nyckelteknologi inom funktionell genomik, som gör det möjligt för oss att analysera funktionen hos hela enzymer på systemnivå", säger professor Sang Yup Lee.
Han tillade, "Vi kommer att kunna använda det för att utveckla miljövänliga mikrobiella fabriker baserade på omfattande metaboliska modeller i genomskala, vilket potentiellt minimerar saknad information om metabolism."
Mer information: Gi Bae Kim et al, Funktionell anteckning av enzymkodande gener med djupinlärning med transformatorlager, Nature Communications (2023). DOI:10.1038/s41467-023-43216-z
Journalinformation: Nature Communications
Tillhandahålls av Korea Advanced Institute of Science and Technology (KAIST)