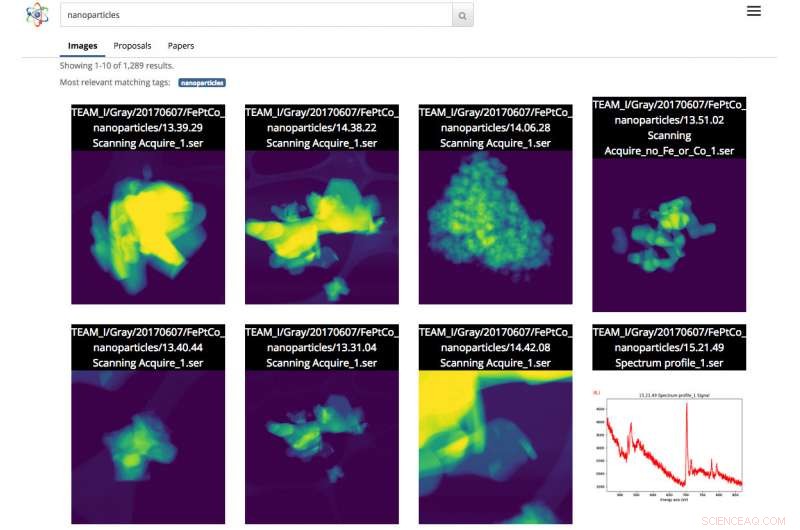
Skärmdump av Science Search -gränssnittet. I detta fall, användaren gjorde en bildsökning av nanopartiklar. Upphovsman:Gonzalo Rodrigo, Berkeley Lab
När vetenskapliga datamängder ökar i både storlek och komplexitet, förmågan att märka, filtrera och sök efter denna översvämning av information har blivit en mödosam, tidskrävande och ibland omöjlig uppgift, utan hjälp av automatiserade verktyg.
Med detta i åtanke, ett team av forskare från Lawrence Berkeley National Laboratory (Berkeley Lab) och UC Berkeley utvecklar innovativa maskininlärningsverktyg för att dra kontextuell information från vetenskapliga datamängder och automatiskt generera metadatataggar för varje fil. Forskare kan sedan söka i dessa filer via en webbaserad sökmotor efter vetenskapliga data, kallad Science Search, som Berkeley -teamet bygger.
Som ett bevis på konceptet, teamet arbetar med personal vid Department of Energy's (DOE) Molecular Foundry, ligger på Berkeley Lab, för att demonstrera begreppen Science Search på bilderna som tagits av anläggningens instrument. En betaversion av plattformen har gjorts tillgänglig för Foundry -forskare.
"Ett verktyg som Science Search har potential att revolutionera vår forskning, "säger Colin Ophus, forskare inom Molecular Foundry inom National Center for Electron Microscopy (NCEM) och Science Search Collaborator. "Vi är en skattebetalarfinansierad nationell användaranläggning, och vi skulle vilja göra all information allmänt tillgänglig, snarare än det lilla antalet bilder som valts för publicering. Dock, i dag, de flesta uppgifterna som samlas in här tittas bara på riktigt av en handfull människor - dataproducenterna, inklusive PI (huvudutredare), sina postdoktorer eller doktorander - för det finns för närvarande inget enkelt sätt att sålla igenom och dela data. Genom att göra denna rådata lätt sökbar och delbar, via Internet, Science Search kan öppna denna reservoar med "mörk data" för alla forskare och maximera vår anläggnings vetenskapliga inverkan. "
Utmaningarna med att söka efter vetenskapsdata
I dag, sökmotorer används allestädes närvarande för att hitta information på Internet, men sökning av vetenskapliga data innebär en annan uppsättning utmaningar. Till exempel, Googles algoritm förlitar sig på mer än 200 ledtrådar för att uppnå en effektiv sökning. Dessa ledtrådar kan komma i form av nyckelord på en webbsida, metadata i bilder eller publikåterkoppling från miljarder människor när de klickar på den information de letar efter. I kontrast, vetenskapliga data finns i många former som är radikalt annorlunda än en genomsnittlig webbsida, kräver sammanhang som är specifikt för vetenskapen och ofta också saknar metadata för att ge sammanhang som krävs för effektiva sökningar.
Vid nationella användarfaciliteter som Molecular Foundry, forskare från hela världen ansöker om tid och reser sedan till Berkeley för att använda extremt specialiserade instrument gratis. Ophus noterar att de nuvarande kamerorna på mikroskop vid gjuteriet kan samla upp till en terabyte data på under 10 minuter. Användare måste sedan manuellt bläddra igenom dessa data för att hitta kvalitetsbilder med "bra upplösning" och spara den informationen i ett säkert delat filsystem, som Dropbox, eller på en extern hårddisk som de så småningom tar med sig hem för att analysera.
Ofta, forskarna som kommer till Molecular Foundry har bara ett par dagar på sig att samla in sina data. Eftersom det är väldigt tråkigt och tidskrävande att manuellt lägga till anteckningar till terabyte med vetenskaplig data och det inte finns någon standard för att göra det, de flesta forskare skriver bara kortfattade beskrivningar i filnamnet. Detta kan vara meningsfullt för den som sparar filen, men gör ofta inte mycket mening för någon annan.
"Bristen på riktiga metadatatiketter orsakar så småningom problem när forskaren försöker hitta data senare eller försöker dela den med andra, "säger Lavanya Ramakrishnan, en personalvetare vid Berkeley Labs division för beräkningsforskning (CRD) och medhuvudutredare för projektet Science Search. "Men med maskininlärningstekniker, vi kan få datorer att hjälpa till med det som är jobbigt för användarna, inklusive att lägga till taggar i data. Sedan kan vi använda dessa taggar för att effektivt söka i data. "
För att åtgärda metadatafrågan, Berkeley Lab-teamet använder maskininlärningstekniker för att bryta "vetenskapens ekosystem"-inklusive instrumentstämplar, anläggningens användarloggar, vetenskapliga förslag, publikationer och filsystemstrukturer - för kontextuell information. Den kollektiva informationen från dessa källor inklusive experimentets tidsstämpel, anteckningar om upplösning och filter som används och användarens begäran om tid, alla ger kritisk kontextuell information. Berkeley-labteamet har sammanställt en innovativ mjukvarustack som använder maskininlärningstekniker, inklusive bearbetning av naturligt språk, drar sammanhangsord för det vetenskapliga experimentet och skapar automatiskt metadatataggar för data.
För proof-of-concept, Ophus delade data från Molecular Foundrys TEAM 1 -elektronmikroskop vid NCEM som nyligen samlades in av anläggningspersonalen, med Science Search Team. Han erbjöd sig också att märka några tusen bilder för att ge verktygen för maskininlärning några etiketter att börja lära sig. Även om detta är en bra början, Gunther Weber, forskare i Science Search, påpekar att de flesta framgångsrika maskininlärningsprogram vanligtvis kräver betydligt mer data och feedback för att ge bättre resultat. Till exempel, för sökmotorer som Google, Weber noterar att utbildningsdatauppsättningar skapas och maskininlärningstekniker valideras när miljarder människor världen över verifierar sin identitet genom att klicka på alla bilder med gatuskyltar eller skyltfönster efter att ha skrivit in sina lösenord, eller på Facebook när de taggar sina vänner i en bild.
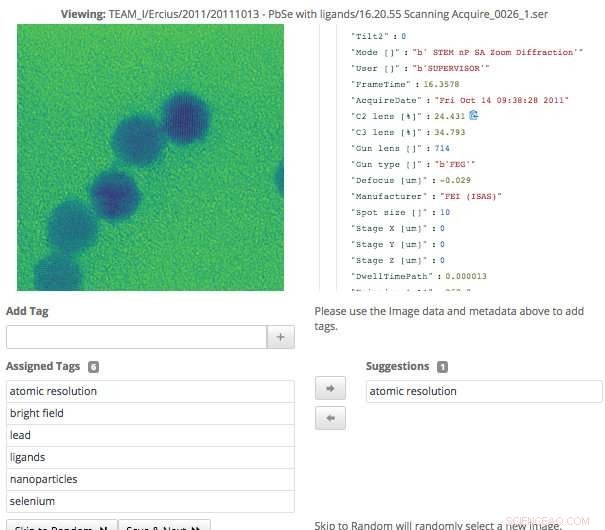
Denna skärmdump av Science Search -gränssnittet visar hur användare enkelt kan validera metadatataggar som har genererats via maskininlärning, eller lägg till information som inte redan har fångats. Upphovsman:Gonzalo Rodrigo, Berkeley Lab
"När det gäller vetenskapsdata kan bara en handfull domänexperter skapa utbildningsuppsättningar och validera maskininlärningstekniker, så ett av de stora pågående problemen vi står inför är ett extremt litet antal träningsuppsättningar, "säger Weber, som också är personalvetare i Berkeley Labs CRD.
För att övervinna denna utmaning, forskarna i Berkeley Lab använde transferlärning för att begränsa frihetsgraderna, eller parameterräkningar, på deras konvolutionella neurala nätverk (CNN). Transferlärning är en metod för maskininlärning där en modell som utvecklats för en uppgift återanvänds som utgångspunkt för en modell på en andra uppgift, vilket gör att användaren kan få mer exakta resultat från en mindre träningssats. När det gäller TEAM I -mikroskopet, data som produceras innehåller information om vilket driftläge instrumentet var i vid tidpunkten för insamlingen. Med den informationen, Weber kunde träna det neurala nätverket på den klassificeringen så att den automatiskt kunde generera den här driftsättetiketten. Han frös sedan det konvolutionella lagret av nätverket, vilket innebar att han bara skulle behöva träna om de tätt sammankopplade skikten. Detta tillvägagångssätt minskar effektivt antalet parametrar på CNN, så att laget kan få några meningsfulla resultat från sina begränsade träningsdata.
Maskininlärning för att bryta det vetenskapliga ekosystemet
Förutom att generera metadatataggar genom utbildningsdatauppsättningar, Berkeley Lab-teamet utvecklade också verktyg som använder maskininlärningstekniker för att utvinna vetenskapens ekosystem för datakontext. Till exempel, datainmatningsmodulen kan titta på en mängd informationskällor från det vetenskapliga ekosystemet - inklusive instrumentstämplar, användarloggar, förslag och publikationer - och identifiera gemensamma saker. Verktyg utvecklade på Berkeley Lab som använder naturliga språkbehandlingsmetoder kan sedan identifiera och rangordna ord som ger data sammanhang och underlättar meningsfulla resultat för användare senare. Användaren kommer att se något som liknar resultatsidan för en internetsökning, där innehåll med mest text som matchar användarens sökord kommer att visas högre på sidan. Systemet lär sig också av användarfrågor och sökresultaten de klickar på.
Eftersom vetenskapliga instrument genererar en ständigt växande mängd data, alla aspekter av Berkeley -teamets vetenskapliga sökmotor behövde vara skalbara för att hålla jämna steg med hastigheten och skalan på de datavolymer som produceras. Teamet uppnådde detta genom att installera sitt system i en spin -instans på Cori -superdatorn vid National Energy Research Scientific Computing Center (NERSC). Spin är en Docker-baserad edge-services-teknik utvecklad på NERSC som kan komma åt anläggningens högpresterande datorsystem och lagring på baksidan.
"En av anledningarna till att vi kan bygga ett verktyg som Science Search är vår tillgång till resurser på NERSC, "säger Gonzalo Rodrigo, en postdoktoral forskare i Berkeley Lab som arbetar med naturliga språkhantering och infrastrukturutmaningar i Science Search. "Vi måste lagra, analysera och hämta riktigt stora datamängder, och det är användbart att ha tillgång till en superdatoranläggning för att klara det tunga för dessa uppgifter. NERSC's Spin är en bra plattform för att köra vår sökmotor som är en användarvänlig applikation som kräver tillgång till stora datamängder och analytisk data som bara kan lagras på stora superdatorlagringssystem. "
Ett gränssnitt för validering och sökning av data
När Berkeley Lab -teamet utvecklade gränssnittet för användare att interagera med sitt system, de visste att det skulle behöva uppnå ett par mål, inklusive effektiv sökning och möjliggör mänsklig input till maskininlärningsmodellerna. Eftersom systemet förlitar sig på domänsexperter för att hjälpa till att generera utbildningsdata och validera maskininlärningsmodellutmatningen, gränssnittet som behövs för att underlätta det.
"Märkningsgränssnittet som vi utvecklat visar de tillgängliga originaldata och metadata, liksom alla maskingenererade taggar vi har hittills. Expertanvändare kan sedan bläddra i data och skapa nya taggar och granska alla maskingenererade taggar för noggrannhet, "säger Matt Henderson, som är datorsystemingenjör inom CRD och leder utvecklingsarbetet för användargränssnitt.
För att underlätta en effektiv sökning efter användare baserat på tillgänglig information, teamets sökgränssnitt ger en sökmekanism för tillgängliga filer, förslag och papper som de Berkeley-utvecklade maskininlärningsverktygen har analyserat och extraherat taggar från. Varje listat sökresultatobjekt representerar en sammanfattning av data, med en mer detaljerad sekundärvy tillgänglig, inklusive information om taggar som matchade det här objektet. Teamet undersöker för närvarande hur man bäst kan integrera användarfeedback för att förbättra modellerna och taggarna.
"Att ha förmågan att utforska datamängder är viktigt för vetenskapliga genombrott, och detta är första gången som något liknande Science Search har försökts, "säger Ramakrishnan." Vår ultimata vision är att bygga grunden som så småningom kommer att stödja ett "Google" för vetenskaplig data, där forskare till och med kan söka i distribuerade datamängder. Vårt nuvarande arbete ger den grund som behövs för att nå den ambitiösa visionen. "
"Berkeley Lab är verkligen en idealisk plats att bygga ett verktyg som Science Search eftersom vi har ett antal användarfaciliteter, som Molecular Foundry, som har decenniers värde av data som skulle ge ännu mer värde för det vetenskapliga samfundet om data kunde sökas och delas, "tillägger Katie Antypas, som är huvudutredare för Science Search och chef för NERSC:s dataavdelning. "Dessutom har vi stor tillgång till maskininlärningsexpertis inom Berkeley Lab Computing Sciences-området samt HPC-resurser på NERSC för att bygga upp dessa möjligheter."