
Kredit:DeepMind
Spelarna applåderar, säg ord som Whoo, slå plastknivar i bordet och njut av de bästa helgerna med artificiell intelligens som huvudakt, tack vare AI som släpps lös i spel.
WIRED UK vetenskapsredaktör, Matt Reynolds, tittade på DeepMinds inverkan på AI-milstolpar:"Det har spelat ut Go-mästare, överträffade professionella StarCraft-spelare och riktade sin uppmärksamhet mot schack och shogi."
Låt spelen fortsätta men det seriösa måste på allvar lysa. I korthet, vi kan beundra att släppandet av AI i syfte att vetenskaplig upptäckt har blivit särskilt levande och friskt tack vare forskningen vid DeepMind.
Teknikbevakare kommenterade denna vecka forskningsartiklar som visar styrkorna hos AI. "När AI mognar som ett område (och får slut på videospel att erövra) kommer förmodligen fler av dess prestationer att se ut så här:solida förbättringar inom viktiga forskningsdomäner."
En forskningsartikel publicerad i Natur gjorde omedelbara nyheter som en tidning om hur AI kan användas för att förutsäga proteinveckning. VentureBeat hänvisade till formidabla utmaningar som närmar sig målen.
Uppsatsen är "Förbättrad proteinstrukturförutsägelse med hjälp av potentialer från djupinlärning, "Artikeln publicerades online den 15 december i Natur .
DeepMind-systemet kallas AlphaFold. Reynolds hade en intressant anteckning om AlphaFold och datamängden:"Proteinvikningsfältet är också väl inrättat för att träna artificiellt intelligenta agenter. Det har ett stort dataset - Protein Data Bank, ett förråd av 3D-strukturen och den genetiska sammansättningen av 150, 000 proteiner, som användes för att träna DeepMinds proteinstrukturförutsägande system, kallas AlphaFold."
Uppsatsen utforskade DeepMinds framgångar hittills med att använda AI för att förutsäga proteinveckning. Det stora med proteinveckning är att, som Kelsey Piper i Vox anmärkte, det är en "avgörande fråga för att utveckla nya läkemedel."
Tre i DeepMind-teamet, Andrew Senior, John Jumper och Demis Hassabis, postade ett blogginlägg den 15 januari som förklarar proteinstrukturer, proteinveckningsutmaningen och vad de ville uppnå.
"Som demonstreras av Levinthals paradox, det skulle ta längre tid än det kända universums ålder att slumpmässigt räkna upp alla möjliga konfigurationer av ett typiskt protein innan de når den sanna 3D-strukturen – men proteinerna själva viker sig spontant, inom millisekunder. Att förutsäga hur dessa kedjor kommer att vikas in i den intrikata 3D-strukturen av ett protein är vad som kallas "proteinveckningsproblemet" – en utmaning som forskare har arbetat med i decennier."
Teamet visade att de kunde träna ett neuralt nätverk för att göra exakta förutsägelser "av avstånden mellan par av rester, som förmedlar mer information om strukturen än kontaktförutsägelser."
De pratade om optimering via "en enkel gradientnedstigningsalgoritm för att generera strukturer utan komplexa samplingsprocedurer."
Det stora med proteinfet är att, sa Piper, det är en "avgörande fråga för att utveckla nya läkemedel." Hur så? Och, varför är "former" så viktiga? "Formen som proteiner tar förutsäger vilka andra ämnen de kommer att interagera med, så att förstå proteinveckning är avgörande för läkemedelsupptäckten och kan till och med användas för att utveckla nya tillverkningsprocesser, sa Piper.
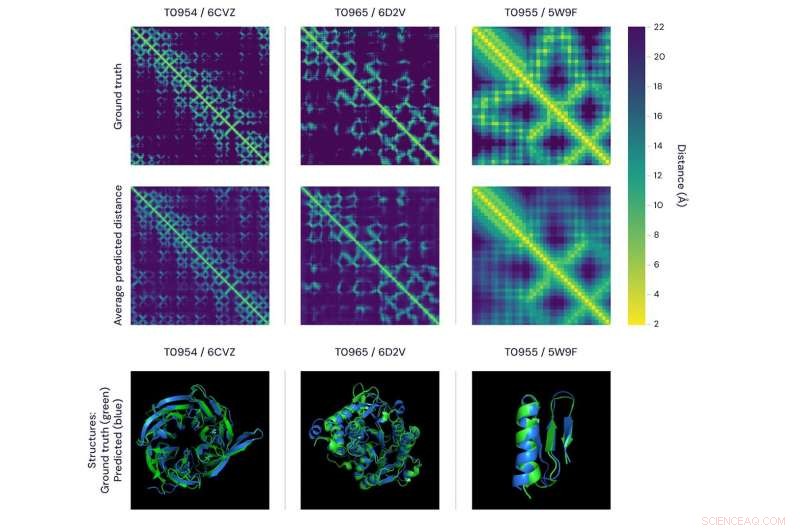
Två sätt att visualisera noggrannheten i alphafolds förutsägelser. den översta siffran visar avståndsmatriserna för tre proteiner. Ljusstyrkan för varje pixel representerar avståndet mellan aminosyrorna i sekvensen som består av proteinet – ju ljusare pixel, ju närmare paret. På den översta raden visas de verkliga, experimentellt bestämda avstånd och, på nedre raden, medelvärdet av AlphaFolds förutsagda avståndsfördelningar. Viktigt, dessa matchar väl på både global och lokal skala. de nedre panelerna representerar samma jämförelse med 3D-modeller, med AlphaFolds förutsägelser (blå) kontra markens sanningsdata (grön) för samma tre proteiner. Kredit:DeepMind
DeepMind-bloggen sa att "ett proteins form tros styra dess funktion. När väl ett proteins form är förstått, dess roll i cellen kan gissas på, och forskare kan utveckla läkemedel som fungerar med proteinets unika form."
AlphaFold kan hjälpa forskare att få ett bättre grepp om proteiners funktion och felfunktion.
Ett anmärkningsvärt citat i VentureBeat från chefen för UCLs bioinformatikgrupp David Jones, som gav DeepMind-teamet råd om delar av projektet:
"Experimentella tekniker för att bestämma proteinstrukturer är tidskrävande och dyra, så det finns en stor efterfrågan på bättre datoralgoritmer för att beräkna strukturerna av proteiner direkt från gensekvenserna som kodar för dem, och DeepMinds arbete med att tillämpa AI på detta långvariga problem inom molekylärbiologi är ett definitivt framsteg."
Vilka typer av experimentella tekniker har varit tidskrävande och dyra? Bloggen nämnde kryo-elektronmikroskopi, kärnmagnetisk resonans och röntgenkristallografi, var och en beroende på trial and error, "som kan ta år av arbete, och kostar tiotals eller hundratusentals dollar per proteinstruktur."
Kyle Wiggers in VentureBeat :"Med AlphaFold, DeepMinds forskargrupp fokuserade på problemet med att modellera målformer från grunden utan att använda lösta proteiner som mallar."
Hur naturen definierar proteinveckning:"Proteinveckning är den process genom vilken proteiner uppnår sin mogna funktionella (native) tertiära struktur, och börjar ofta med översättning. Proteinveckning kräver chaperoner och involverar ofta stegvis etablering av vanliga sekundära och supersekundära strukturer, nämligen α-helixar och β-sheets, som viker sig snabbt, stabiliserad av vätebindning och disulfidbryggor, och sedan tertiär struktur."
Tillbaka i juli, före publiceringen av DeepMind-tidningen, Natur hade redan noterat att "loppet att knäcka en av biologins största utmaningar - att förutsäga 3D-strukturerna hos proteiner från deras aminosyrasekvenser - intensifieras, tack vare nya metoder för artificiell intelligens (AI).
DeepMind-blogginlägget sa att de gjorde kod tillgänglig (GitHub), och erbjöd länken, för alla som vill lära sig mer eller replikera sina resultat.
I sitt blogginlägg sa de varför deras system är viktigt:"Vårt system, AlphaFold... bygger på årtionden av tidigare forskning med hjälp av stora genomiska datauppsättningar för att förutsäga proteinstruktur. 3D-modellerna av proteiner som AlphaFold genererar är mycket mer exakta än någon som har kommit tidigare."
Går vidare, bloggen angav att ett verktyg som AlphaFold kan hjälpa forskare med sällsynta sjukdomar att snabbt och ekonomiskt förutsäga formen på ett protein av intresse och så småningom kan hjälpa dem att bidra till effektiv läkemedelsupptäckt, samtidigt som kostnaderna för experiment minskas.
Forskning om sällsynta sjukdomar är inte det enda område som kan dra nytta av vad de har åstadkommit. Bloggen pratade om föroreningar som plast och olja; framsteg inom biologiskt nedbrytbara enzymer som möjliggörs av proteindesign kan hjälpa till att bryta ner avfall på ett sätt som är mer miljövänligt.
"Faktiskt, Forskare har redan börjat konstruera bakterier för att utsöndra proteiner som gör avfall biologiskt nedbrytbart, och lättare att bearbeta, " enligt bloggen.
© 2020 Science X Network