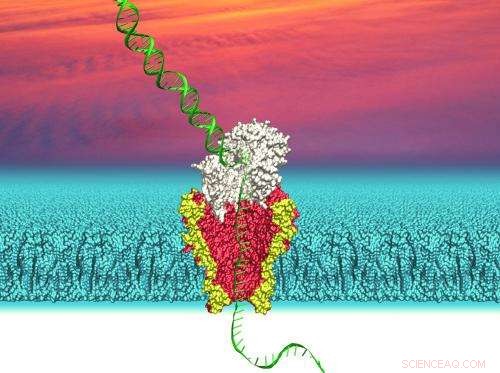
Illustration av en nanopor härledd från en genetiskt modifierad bakteriell membrankanal som används för att sekvensera DNA. Kredit:Ian Derrington
(Phys.org) —En lågkostnadsteknologi kan göra det möjligt att läsa långa DNA-sekvenser mycket snabbare än nuvarande tekniker.
Forskningen utvecklar en teknik, kallas nanopore DNA-sekvensering. Om den skulle fulländas skulle den en dag kunna användas för att skapa handhållna enheter som snabbt kan identifiera DNA-sekvenser från vävnadsprover och miljön, sade forskare från University of Washington som utvecklade och testade metoden.
"En anledning till att människor är så exalterade över nanopore DNA-sekvensering är att tekniken möjligen skulle kunna användas för att skapa "tricorder"-liknande enheter för att upptäcka patogener eller diagnostisera genetiska störningar snabbt och på plats, sa Andrew Laszlo, huvudförfattare och doktorand i Jen Gundlachs laboratorium, en UW-professor i fysik som ledde projektet.
Uppsatsen "Decoding long nanopore sequencing reads of natural DNA" beskriver den nya tekniken. Den visas den 25 juni i den avancerade onlineupplagan av tidskriften Naturens bioteknik .
De flesta av de nuvarande gensekvenseringsteknologierna kräver att man arbetar med korta DNA-bitar, typiskt 50 till 100 nukleotider långa. Dessa måste bearbetas av stora sekvenserare i ett laboratorium. Den besvärliga processen kan ta dagar till veckor att slutföra.
Nanopore-teknologin drar fördel av det lilla, tunnelliknande strukturer som finns i bakteriemembran. I naturen, sådana porer tillåter bakterier att kontrollera flödet av näringsämnen över sina membran.
UW-forskare använde nanopore Mycobacterium smegmatis porin A (MspA). Denna bakteriella por har förändrats genetiskt så att den smalaste delen av kanalen har en diameter på ungefär en nanometer, eller 1 miljarddels meter. Detta är tillräckligt stort för att en enda DNA-sträng ska kunna passera igenom. Den modifierade nanoporen sätts sedan in i ett membran som separerar två saltlösningar för att skapa en kanal som förbinder de två lösningarna.
För att läsa en sekvens av DNA med detta system, en liten spänning appliceras över membranet för att få jonerna i saltlösningen att strömma genom nanoporen. Jonflödet skapar en mätbar ström. Om en DNA-sträng läggs till lösningen på ena sidan av membranet och sedan går in i en por, de skrymmande DNA-molekylerna kommer att hindra flödet av den mycket mindre jonen och därigenom förändra strömmen. Hur mycket strömmen förändras beror på vilka nukleotider – de enskilda molekylerna adenin, guanin, cytosin och tymin som utgör DNA-kedjan – finns inne i poren. Att upptäcka förändringar i ström kan avslöja vilka nukleotider som passerar genom nanoporens kanal vid ett givet ögonblick.
Sedan tekniken först föreslogs på 1990-talet, forskare hoppades att nanopore DNA-sekvensering skulle erbjuda en billig, snabbt alternativ till nuvarande gensekvensering. Men deras försök har frustrerats av flera utmaningar. Det är svårt att identifiera varje nukleotid en efter en när de passerar genom nanoporen. Istället, forskare måste arbeta med förändringar i strömmen i samband med fyra nukleotider åt gången. Dessutom, vissa nukleotider kan missas eller läsas mer än en gång. Följaktligen, nuvarande nanopore-sekvenseringsteknologi ger en oprecis avläsning av en DNA-sekvens.
UW-forskarna beskriver hur de gick förbi dessa problem. Forskarna identifierade först de elektroniska signaturerna för alla möjliga nukleotidkombinationer med de fyra nukleotiderna som utgör DNA - totalt 256 kombinationer (4 x 4 x 4 x 4).
De skapade sedan datoralgoritmer för att matcha de aktuella förändringarna som genereras när ett segment av DNA passerar genom poren med aktuella förändringar som förväntas från DNA-sekvenser av kända gener och genom lagrade i en databas. En matchning skulle visa att sekvensen av DNA:t som passerade genom poren var identisk eller nära den DNA-sekvens som lagras i databasen. Hela processen skulle ta minuter till några timmar, istället för veckor.
För att testa detta tillvägagångssätt, forskarna använde sitt nanoporsystem för att läsa sekvensen av bakteriofag Phi X 174, ett virus som infekterar bakterier och som vanligtvis används för att utvärdera nya genomsekvenseringstekniker. De fann att tillvägagångssättet på ett tillförlitligt sätt läste bakteriofagens DNA-sekvenser och kunde läsa sekvenser så länge som 4, 500 nukleotider.
"Det här är första gången någon har visat att nanoporer kan användas för att generera tolkbara signaturer som motsvarar mycket långa DNA-sekvenser från verkliga genom, " sa medförfattaren Jay Shendure, en UW docent i genomvetenskap vars labb utvecklar tillämpningar av genomsekvenseringsteknologier. "Det är ett stort steg framåt."
Eftersom tekniken bygger på att matcha avläsningar till databaser med tidigare sekvenserade gener och genom, det kan ännu inte användas för att sekvensera en nyupptäckt gen eller genom, forskarna sa, men med vissa finesser, de tillade, det borde vara möjligt att förbättra prestandan på detta område. För att påskynda forskningen om denna nya teknik, forskarna gör sina metoder, data och datoralgoritmer fullt tillgängliga för alla.
"Trots de återstående hindren, vår demonstration att en billig enhet på ett tillförlitligt sätt kan läsa sekvenserna av naturligt förekommande DNA och kan tolka DNA-segment så länge som 4, 500 nukleotider i längd representerar ett stort framsteg inom nanopore DNA-sekvensering, sa Gundlach.