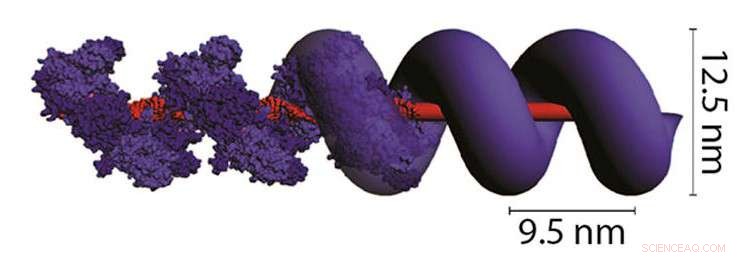
Proteinet RecA (lila enheter), sveper runt och förstärker dubbelsträngat DNA, gör det möjligt för forskare att bygga stora strukturer med det genetiska materialet. Kredit:NIST
DNA är livets grejer, men det är också nanoteknikens grejer. Eftersom DNA-molekyler med komplementära kemiska strukturer känner igen och binder till varandra, DNA-strängar kan passa ihop som legoklossar för att göra föremål i nanoskala med komplex form och struktur.
Men forskare måste arbeta med mycket större samlingar av DNA för att förverkliga ett nyckelmål:att bygga hållbara miniatyrenheter som biosensorer och läkemedelsleveransbehållare. Det har varit svårt eftersom långa DNA-kedjor är diskettiga och standardmetoden för att sätta ihop långa kedjor är benägen att göra fel.
Genom att använda ett DNA-bindande protein som kallas RecA som ett slags armeringsjärn i nanoskala, eller armeringsjärn, för att stödja den diskettiga DNA-ställningen, forskare vid National Institute of Standards and Technology (NIST) har konstruerat flera av de största rektangulära, linjära och andra former som någonsin satts ihop från DNA. Strukturerna kan vara två till tre gånger större än de som byggs med standardtekniker för DNA-självmontering.
Dessutom, eftersom den nya metoden kräver färre kemiskt distinkta delar för att bygga organiserade strukturer än standardtekniken, känd som DNA-origami, det kommer sannolikt att minska antalet fel vid konstruktionen av formerna. Det är ett stort plus för ansträngningen att producera pålitliga DNA-baserade enheter i stora kvantiteter, sa NIST-forskaren Alex Liddle.
Även om RecAs förmåga att binda till dubbelsträngat DNA har varit känd i flera år, NIST-teamet är det första att integrera filament av detta protein i sammansättningen av DNA-strukturer. Tillägget av RecA ger en särskild fördel:När en enhet av proteinet binder till ett litet segment av dubbelsträngat DNA, den lockar automatiskt andra enheter att ställa upp bredvid den, på samma sätt som stångmagneter kommer att sammanfoga ände till ände. Som tegelstenar som fyller en grund, RecA kantar hela längden av DNA-strängen, stretching, vidga och stärka den. En diskett, 2 nanometer bred DNA-sträng kan förvandlas till en stel struktur som är mer än fyra gånger så bred.
"RecA-metoden utökar i hög grad förmågan hos DNA-självsammansättningsmetoder för att bygga större och mer sofistikerade strukturer, " sa NIST:s Daniel Schiffels.
Schiffels, Liddle och deras kollega Veronika Szalai beskriver sitt arbete i en ny artikel i ACS Nano .
Den nya metoden innehåller DNA-origamitekniken och går utöver den, enligt Liddle. I DNA-origami, korta DNA-strängar som har en specifik sekvens av fyra baspar används som häftklamrar för att binda samman långa sektioner av DNA. För att göra det magra DNA-skelettet starkare och tjockare, strängen kan loopa tillbaka på sig själv, snabbt använda upp det långa snöret.
Om DNA-origami handlar om vikning, Liddle liknade sitt teams nya metod för att bygga ett rum, börjar med en planlösning. Kortets läge, enkelsträngade bitar av DNA som fungerar som häftklamrar markerar hörnen av rummet. Mellan hörnen ligger en lång, smal bit av enkelsträngat DNA. Enzymet DNA-polymeras omvandlar en del av den långa biten av enkelsträngat DNA till den dubbelsträngade versionen av molekylen, ett nödvändigt steg eftersom RecA endast binder starkt till dubbelsträngat DNA. Sedan monteras RecA längs med dubbelsträngen, förstärker DNA-strukturen och begränsar behovet av extra häftklamrar för att behålla sin form.
Med färre häftklamrar krävs, RecA-metoden kan sannolikt bygga organiserade strukturer med färre fel än DNA-origami, sa Liddle.