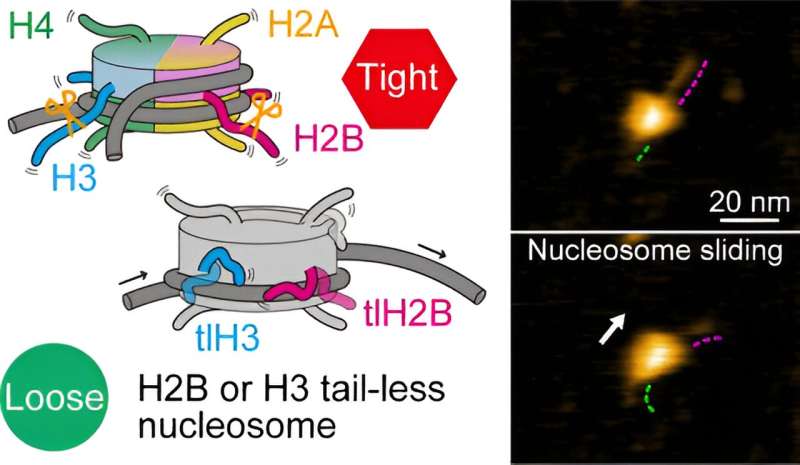
Hos växter och djur är de grundläggande förpackningsenheterna av DNA, som bär genetisk information, de så kallade nukleosomer. En nukleosom består av ett segment av DNA lindat runt åtta proteiner som kallas histoner.
Under genuttryck (processen som ligger till grund för proteinproduktion) är nukleosomer involverade i olika dynamiska strukturella förändringar, såsom nukleosomglidning, DNA-avveckling och andra DNA-histoninteraktioner. Av särskild betydelse i dessa processer är histonernas slutstrukturer, eller svansar.
Histonsvansar genomgår kemiska modifieringar, vilket ändrar histonens funktion efter behov. Detaljerade studier, och särskilt visualiseringar, av nukleosomens dynamik är avgörande för att bättre förstå rollen av histonsvansar.
Mikihiro Shibata från Kanazawa University och kollegor har nu lyckats göra videoinspelningar av svanslösa nukleosomer, vilket visar att frånvaron av histonsvansar avsevärt ökar en nukleosomes dynamiska aktivitet. Studien publiceras i Nano Letters .
Forskarna använde höghastighets atomkraftsmikroskopi (HS-AFM), ett kraftfullt nanobildverktyg för att visualisera molekylära strukturer och deras dynamik vid hög rumslig och tidsmässig upplösning.
För detta behövde nukleosomerna placeras på ett substrat. Shibata och kollegor använde en film av så kallade pelar[5]arener (molekyler med en pentagonal rörformig struktur) som substrat, vilket bildar en idealisk yta eftersom nukleosomerna lätt adsorberas till den utan att dynamiska processer undertrycks.
Forskarna tittade först på nukleosomer där alla åtta histoner saknade svansar. Baserat på deras HS-AFM-observationer drog de slutsatsen att nukleosomglidning och DNA-upplindning/omlindning inträffade oftare än för normala (kanoniska) nukleosomer. Detta tyder på att utan svansar försvagas histon-DNA-interaktionen, vilket leder till en situation där DNA lättare kan lossna från histonerna.
För att bättre förstå rollerna för specifika histonsvansar förberedde Shibata och kollegor nukleosomer där en typ av histon var svanslös. Det finns fyra olika typer av histoner, kallade H2A, H2B, H3 och H4. HS-AFM-experiment på nukleosomerna visade att H2B och H3 svanslösa nukleosomer visade en ökad dynamikfrekvens. Omvänt betyder detta att kanoniska H2B- och H3-histoner är avgörande för nukleosomstabilitet.
Forskarna påpekar att de inte kunde observera någon faktisk rörelse av histonsvansar - troligen var den tidsmässiga upplösningen av studien, 0,3 sekunder, mycket långsammare än hastigheten för svansarnas inlindnings-/avlindningsdynamik. Trots denna begränsning bevisar Shibatas och hans kollegors arbete tydligt att svansarna av H2B- och H3-histoner är de huvudsakliga bidragsgivarna till nukleosomdynamiken.
När det gäller framtida arbete, citerar forskarna, "en teknik för att tagga histonsvansspetsar kan göra det möjligt för HS-AFM att fånga rörelserna av histonsvansarna själva."
Höghastighets atomkraftsmikroskopi
Den allmänna principen för atomkraftsmikroskopi (AFM) är att få en mycket liten spets att skanna ytan på ett prov. Under denna horisontella (xy) skanning följer spetsen, som är fäst vid en liten konsol, provets vertikala (z) profil, vilket inducerar en kraft på konsolen som kan mätas.
Storleken på kraften vid xy-positionen kan relateras till z-värdet; xyz-data som genereras under en skanning resulterar sedan i en höjdkarta som ger strukturell information om det undersökta provet. I höghastighets-AFM (HS-AFM) är arbetsprincipen något mer involverad:konsolen är gjord för att oscillera nära sin resonansfrekvens.
När spetsen flyttas runt en yta registreras variationerna i amplituden (eller frekvensen) av fribärarens oscillation – som resulterar från spetsens interaktion med provets yta – eftersom dessa ger ett mått på det lokala z-värdet. AFM involverar inte linser, så dess upplösning begränsas inte av den så kallade diffraktionsgränsen som till exempel vid röntgendiffraktion.
HS-AFM resulterar i en video, där tidsintervallet mellan bildrutor beror på hastigheten med vilken en enskild bild kan genereras (genom att xy-scanna provet).
Forskare vid Nano Life Science Institute (WPI-NanoLSI), Kanazawa University har under de senaste åren utvecklat HS-AFM vidare, så att den kan användas för att studera biokemiska molekyler och biomolekylära processer i realtid. Mikihiro Shibata och hans kollegor har nu tillämpat metoden för att studera nukleosomdynamiken i detalj, och i synnerhet rollen av histonernas molekylära ändar – proteiner som spelar en avgörande roll för DNA-tillgänglighet.
Mer information: Shin Morioka et al, High-Speed Atomic Force Microscopy avslöjar nukleosomens glidning och DNA-upptagning/inpackningsdynamiken hos svanslösa nukleosomer, nanobokstäver (2024). DOI:10.1021/acs.nanolett.4c00801
Journalinformation: Nanobokstäver
Tillhandahålls av Kanazawa University