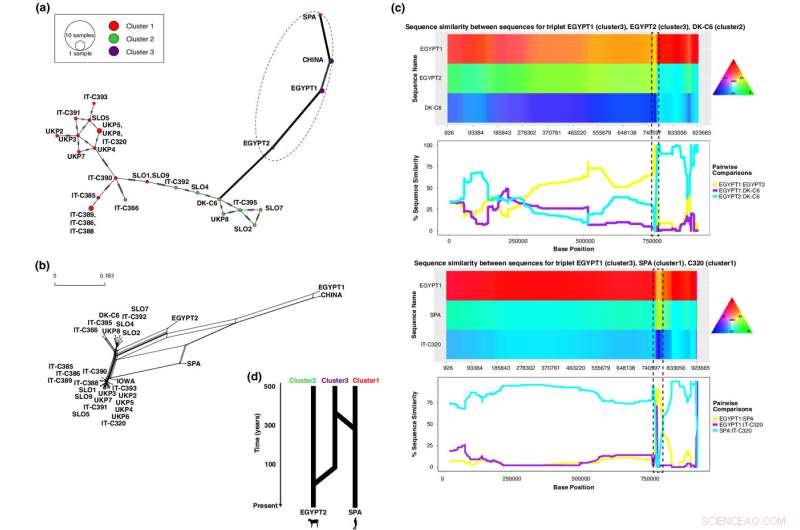
Rekombinationsanalys av kromosom 1 identifierar två hybridsekvenser, Spa1 (ett mänskligt isolat från kluster 1) och Egypten 2 (ett kalvisolat från kluster 3). (a) Nätverk som visar att haplotyper av hybridisolaten Spa1 och Egypt2 skiljer sig från resten av isolaten och är nära associerade med deras mindre föräldrasekvenser (kluster 3-isolat). (b) Fylogenetiskt nätverk som visar loopar mellan hybridisolat, som representerar potentiell rekombination. (c) Sekvenslikhetsdiagram erhållna med HybridCheck för olika tripletter av isolat involverade i en rekombinationshändelse (hybrid, huvudförälder, mindre förälder). Sekvenslikheten visas längs kromosomala positioner (x-axeln) av en färgkarta där regioner med samma polymorfism delar färger (överst) och av linjediagram som rapporterar den procentuella likheten på y-axeln (nederst). Grafer över rekombinanta regioner som visar hög likhet mellan hybridisolat och deras mindre parentala sekvenser vid de rekombinanta regionerna är inneslutna i streckade rutor. (d) Schematisk representation av rekombinationshändelser i hybridisolat, som visar att Egypt2 och Spa1 båda fick genetisk variation från ett kluster 3, cirka 49 (21–96; 95 % KI) och 289 (204–395; 95 % CI) år sedan, respektive. Kredit:Molecular Ecology (2022). DOI:10.1111/mec.16556
Parasiter som orsakar svår diarré kommer sannolikt att bli mer virulenta på grund av den hastighet med vilken de byter ut sitt DNA och utvecklas – enligt ny forskning från University of East Anglia.
Forskare studerade genomerna av Cryptosporidium parvum – en zoonotisk parasit som orsakar svår diarré hos både människor och djur.
De fann att olika härkomster av dessa parasiter i allt större utsträckning byter ut sitt DNA, vilket har hjälpt parasiten att utvecklas snabbare – vilket potentiellt kan resultera i mer virulenta och bättre anpassade stammar.
Den nya studien visar att linjerna Cryptosporidium parvum har utbytt mer DNA under de senaste 200 åren än under hela tiden tidigare.
Och de förklarar detta till globaliseringen och vår allt tätare kontakt med djur, vilket ökar frekvensen av spill-over-händelser.
En av de ledande forskarna, Prof. Cock Van Oosterhout, från UEA:s School of Environmental Sciences, sa:"Cryptosporidium är ett viktigt släkte av zoonotiska parasiter, och det är en av flera mikroorganismer som orsakar diarrésjukdomar hos både människor och vissa boskap. I människor är den ansvarig för cirka 57 000 dödsfall varje år, varav 80 % är bland barn under fem år.
"De allra flesta av dessa finns i låginkomstländer, men utbrott förekommer också i Storbritannien och på andra håll i Europa. Det finns inget effektivt läkemedel eller vaccin tillgängligt, så att förstå överföringen och utvecklingen av denna parasit är avgörande.
"Vi vet att det finns flera härstamningar av parasiten Cryptosporidium parvum, men vi ville förstå mer om hur de utvecklas, och särskilt varför denna art kan bli mer virulent än den var tidigare."
Teamet, ledd av UEA i samarbete med forskare i Italien och Australien, använde helgenomsekvensanalys för att ta reda på mer om dessa genetiska utbyten.
De jämförde 32 hela genomsekvenser från människo- och djurstammar som samlats in i Europa, USA, Egypten och Kina.
Prof. Van Oosterhout sa:"Vi fann att olika härkomster av dessa parasiter i allt större utsträckning byter ut sitt DNA. Under de senaste 200 åren har omkring 22 procent av genomet av dessa parasiter bytts ut.
"Detta är betydligt mer än det DNA som de har utbytt i hela tiden tidigare. Generna som är involverade i virulens verkar vara särskilt påverkade av sådana genetiska utbyten. Genomet hos parasiter som infekterar människor har en del DNA från parasiter som normalt finns i kor och lamm.
"Vi tror att dessa genetiska utbyten hjälper parasiten att utvecklas snabbare, och att detta kan resultera i mer virulenta och bättre anpassade mänskliga parasiter. Detta är verkligen viktigt eftersom, som COVID-19 har visat oss, mänskliga parasiter kan utvecklas snabbt.
"Vi antar att utvecklingshastigheten accelereras ytterligare eftersom parasiten kan utvecklas i flera värdarter. Detta innebär att nya anpassningar som har utvecklats i parasiter som infekterar en värdart nu kan användas av parasiter som infekterar en annan värdart.
"Den ökade anslutningsmöjligheten i vår globaliserade värld och den nära kontakten mellan människor och tama djur ökar frekvensen av spill-over och spillback-händelser.
"Genom att använda helgenomsekvensanalys kan vi nu studera dessa genetiska utbyten och identifiera när och var de äger rum. Detta kommer att hjälpa oss att bättre kontrollera dessa nya infektionssjukdomar av zoonotiska parasiter och patogener," tillade han.
"Nya genetiska utbyten och blandning formar genomet och populationsstrukturen hos en zoonotisk parasit" publiceras i tidskriften Molecular Ecology .